+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human Type 3 IP3 Receptor - Resting State (+IP3/ATP) | ||||||||||||
 マップデータ マップデータ | IP3 Receptor - hIP3R3 Resting State - Tetramer Composite (Chimera 'vop maximum' of four aligned density modified single protomer composite maps) | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | Ion Channel / Calcium Channel / Endoplasmic Reticulum / TRANSPORT PROTEIN | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報sensory perception of bitter taste / DAG and IP3 signaling / inositol 1,3,4,5 tetrakisphosphate binding / sensory perception of umami taste / inositol 1,4,5-trisphosphate-gated calcium channel activity / sensory perception of sweet taste / platelet dense tubular network membrane / Elevation of cytosolic Ca2+ levels / Effects of PIP2 hydrolysis / PLC beta mediated events ...sensory perception of bitter taste / DAG and IP3 signaling / inositol 1,3,4,5 tetrakisphosphate binding / sensory perception of umami taste / inositol 1,4,5-trisphosphate-gated calcium channel activity / sensory perception of sweet taste / platelet dense tubular network membrane / Elevation of cytosolic Ca2+ levels / Effects of PIP2 hydrolysis / PLC beta mediated events / inositol hexakisphosphate binding / inositol 1,4,5 trisphosphate binding / nuclear outer membrane / CLEC7A (Dectin-1) induces NFAT activation / intracellularly gated calcium channel activity / cytoplasmic side of endoplasmic reticulum membrane / brush border / Role of phospholipids in phagocytosis / calcium ion homeostasis / release of sequestered calcium ion into cytosol / Ion homeostasis / FCERI mediated Ca+2 mobilization / phosphatidylinositol binding / FCGR3A-mediated IL10 synthesis / secretory granule membrane / Antigen activates B Cell Receptor (BCR) leading to generation of second messengers / VEGFR2 mediated cell proliferation / sarcoplasmic reticulum / Regulation of insulin secretion / long-term synaptic potentiation / memory / Sensory perception of sweet, bitter, and umami (glutamate) taste / response to calcium ion / Glucagon-like Peptide-1 (GLP1) regulates insulin secretion / sensory perception of taste / apical part of cell / myelin sheath / Ca2+ pathway / positive regulation of cytosolic calcium ion concentration / receptor complex / G protein-coupled receptor signaling pathway / neuronal cell body / calcium ion binding / endoplasmic reticulum membrane / nucleolus / endoplasmic reticulum / nucleoplasm / membrane / plasma membrane / cytoplasm 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.7 Å | ||||||||||||
 データ登録者 データ登録者 | Paknejad N / Sapuru V / Hite RK | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Structural titration reveals Ca-dependent conformational landscape of the IP receptor. 著者: Navid Paknejad / Vinay Sapuru / Richard K Hite /  要旨: Inositol 1,4,5-trisphosphate receptors (IPRs) are endoplasmic reticulum Ca channels whose biphasic dependence on cytosolic Ca gives rise to Ca oscillations that regulate fertilization, cell division ...Inositol 1,4,5-trisphosphate receptors (IPRs) are endoplasmic reticulum Ca channels whose biphasic dependence on cytosolic Ca gives rise to Ca oscillations that regulate fertilization, cell division and cell death. Despite the critical roles of IPR-mediated Ca responses, the structural underpinnings of the biphasic Ca dependence that underlies Ca oscillations are incompletely understood. Here, we collect cryo-EM images of an IPR with Ca concentrations spanning five orders of magnitude. Unbiased image analysis reveals that Ca binding does not explicitly induce conformational changes but rather biases a complex conformational landscape consisting of resting, preactivated, activated, and inhibited states. Using particle counts as a proxy for relative conformational free energy, we demonstrate that Ca binding at a high-affinity site allows IPRs to activate by escaping a low-energy resting state through an ensemble of preactivated states. At high Ca concentrations, IPRs preferentially enter an inhibited state stabilized by a second, low-affinity Ca binding site. Together, these studies provide a mechanistic basis for the biphasic Ca-dependence of IPR channel activity. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_41323.map.gz emd_41323.map.gz | 1.1 GB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-41323-v30.xml emd-41323-v30.xml emd-41323.xml emd-41323.xml | 38 KB 38 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_41323.png emd_41323.png | 116.2 KB | ||
| Filedesc metadata |  emd-41323.cif.gz emd-41323.cif.gz | 8.9 KB | ||
| その他 |  emd_41323_additional_1.map.gz emd_41323_additional_1.map.gz emd_41323_additional_10.map.gz emd_41323_additional_10.map.gz emd_41323_additional_2.map.gz emd_41323_additional_2.map.gz emd_41323_additional_3.map.gz emd_41323_additional_3.map.gz emd_41323_additional_4.map.gz emd_41323_additional_4.map.gz emd_41323_additional_5.map.gz emd_41323_additional_5.map.gz emd_41323_additional_6.map.gz emd_41323_additional_6.map.gz emd_41323_additional_7.map.gz emd_41323_additional_7.map.gz emd_41323_additional_8.map.gz emd_41323_additional_8.map.gz emd_41323_additional_9.map.gz emd_41323_additional_9.map.gz | 255.6 MB 475.7 MB 1.1 GB 255.7 MB 256.6 MB 256 MB 255.9 MB 254.1 MB 255.9 MB 475.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-41323 http://ftp.pdbj.org/pub/emdb/structures/EMD-41323 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41323 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41323 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_41323_validation.pdf.gz emd_41323_validation.pdf.gz | 403.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_41323_full_validation.pdf.gz emd_41323_full_validation.pdf.gz | 403.1 KB | 表示 | |
| XML形式データ |  emd_41323_validation.xml.gz emd_41323_validation.xml.gz | 9.5 KB | 表示 | |
| CIF形式データ |  emd_41323_validation.cif.gz emd_41323_validation.cif.gz | 10.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41323 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41323 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41323 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41323 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8tk8MC  8tkdC  8tkeC  8tkfC  8tkgC  8tkhC  8tkiC  8tl9C  8tlaC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_41323.map.gz / 形式: CCP4 / 大きさ: 1.1 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_41323.map.gz / 形式: CCP4 / 大きさ: 1.1 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | IP3 Receptor - hIP3R3 Resting State - Tetramer Composite (Chimera 'vop maximum' of four aligned density modified single protomer composite maps) | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.629 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
+追加マップ: IP3 Receptor - hIP3R3 Resting State - Consensus Refinement
+追加マップ: IP3 Receptor - hIP3R3 Resting State - Consensus...
+追加マップ: IP3 Receptor - hIP3R3 Resting State - 1...
+追加マップ: IP3 Receptor - hIP3R3 Resting State - Cytosolic...
+追加マップ: IP3 Receptor - hIP3R3 Resting State - JD/TMD...
+追加マップ: IP3 Receptor - hIP3R3 Resting State - BTF1/BTF2/ARM1...
+追加マップ: IP3 Receptor - hIP3R3 Resting State - CLD/ARM3...
+追加マップ: IP3 Receptor - hIP3R3 Resting State - ARM2 Local Refinement (1 Chain)
+追加マップ: IP3 Receptor - hIP3R3 Resting State - Single...
+追加マップ: IP3 Receptor - hIP3R3 Resting State - Consensus...
- 試料の構成要素
試料の構成要素
-全体 : Human Type 3 IP3 Receptor
| 全体 | 名称: Human Type 3 IP3 Receptor |
|---|---|
| 要素 |
|
-超分子 #1: Human Type 3 IP3 Receptor
| 超分子 | 名称: Human Type 3 IP3 Receptor / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 詳細: In the presence of saturating IP3, ATP, and Ca2+ titrated from 1 nM to 10 um |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 1.2 MDa |
-分子 #1: Inositol 1,4,5-trisphosphate receptor type 3
| 分子 | 名称: Inositol 1,4,5-trisphosphate receptor type 3 / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 304.488688 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MSEMSSFLHI GDIVSLYAEG SVNGFISTLG LVDDRCVVEP AAGDLDNPPK KFRDCLFKVC PMNRYSAQKQ YWKAKQTKQD KEKIADVVL LQKLQHAAQM EQKQNDTENK KVHGDVVKYG SVIQLLHMKS NKYLTVNKRL PALLEKNAMR VTLDATGNEG S WLFIQPFW ...文字列: MSEMSSFLHI GDIVSLYAEG SVNGFISTLG LVDDRCVVEP AAGDLDNPPK KFRDCLFKVC PMNRYSAQKQ YWKAKQTKQD KEKIADVVL LQKLQHAAQM EQKQNDTENK KVHGDVVKYG SVIQLLHMKS NKYLTVNKRL PALLEKNAMR VTLDATGNEG S WLFIQPFW KLRSNGDNVV VGDKVILNPV NAGQPLHASN YELSDNAGCK EVNSVNCNTS WKINLFMQFR DHLEEVLKGG DV VRLFHAE QEKFLTCDEY KGKLQVFLRT TLRQSATSAT SSNALWEVEV VHHDPCRGGA GHWNGLYRFK HLATGNYLAA EEN PSYKGD ASDPKAAGMG AQGRTGRRNA GEKIKYCLVA VPHGNDIASL FELDPTTLQK TDSFVPRNSY VRLRHLCTNT WIQS TNVPI DIEEERPIRL MLGTCPTKED KEAFAIVSVP VSEIRDLDFA NDASSMLASA VEKLNEGFIS QNDRRFVIQL LEDLV FFVS DVPNNGQNVL DIMVTKPNRE RQKLMREQNI LKQVFGILKA PFREKGGEGP LVRLEELSDQ KNAPYQHMFR LCYRVL RHS QEDYRKNQEH IAKQFGMMQS QIGYDILAED TITALLHNNR KLLEKHITKT EVETFVSLVR KNREPRFLDY LSDLCVS NH IAIPVTQELI CKCVLDPKNS DILIRTELRP VKEMAQSHEY LSIEYSEEEV WLTWTDKNNE HHEKSVRQLA QEARAGNA H DENVLSYYRY QLKLFARMCL DRQYLAIDEI SQQLGVDLIF LCMADEMLPF DLRASFCHLM LHVHVDRDPQ ELVTPVKFA RLWTEIPTAI TIKDYDSNLN ASRDDKKNKF ANTMEFVEDY LNNVVSEAVP FANEEKNKLT FEVVSLAHNL IYFGFYSFSE LLRLTRTLL GIIDCVQGPP AMLQAYEDPG GKNVRRSIQG VGHMMSTMVL SRKQSVFSAP SLSAGASAAE PLDRSKFEEN E DIVVMETK LKILEILQFI LNVRLDYRIS YLLSVFKKEF VEVFPMQDSG ADGTAPAFDS TTANMNLDRI GEQAEAMFGV GK TSSMLEV DDEGGRMFLR VLIHLTMHDY APLVSGALQL LFKHFSQRQE AMHTFKQVQL LISAQDVENY KVIKSELDRL RTM VEKSEL WVDKKGSGKG EEVEAGAAKD KKERPTDEEG FLHPPGEKSS ENYQIVKGIL ERLNKMCGVG EQMRKKQQRL LKNM DAHKV MLDLLQIPYD KGDAKMMEIL RYTHQFLQKF CAGNPGNQAL LHKHLHLFLT PGLLEAETMQ HIFLNNYQLC SEISE PVLQ HFVHLLATHG RHVQYLDFLH TVIKAEGKYV KKCQDMIMTE LTNAGDDVVV FYNDKASLAH LLDMMKAARD GVEDHS PLM YHISLVDLLA ACAEGKNVYT EIKCTSLLPL EDVVSVVTHE DCITEVKMAY VNFVNHCYVD TEVEMKEIYT SNHIWTL FE NFTLDMARVC SKREKRVADP TLEKYVLSVV LDTINAFFSS PFSENSTSLQ THQTIVVQLL QSTTRLLECP WLQQQHKG S VEACIRTLAM VAKGRAILLP MDLDAHISSM LSSGASCAAA AQRNASSYKA TTRAFPRVTP TANQWDYKNI IEKLQDIIT ALEERLKPLV QAELSVLVDV LHWPELLFLE GSEAYQRCES GGFLSKLIQH TKDLMESEEK LCIKVLRTLQ QMLLKKTKYG DRGNQLRKM LLQNYLQNRK STSRGDLPDP IGTGLDPDWS AIAATQCRLD KEGATKLVCD LITSTKNEKI FQESIGLAIH L LDGGNTEI QKSFHNLMMS DKKSERFFKV LHDRMKRAQQ ETKSTVAVNM NDLGSQPHED REPVDPTTKG RVASFSIPGS SS RYSLGPS LRRGHEVSER VQSSEMGTSV LIMQPILRFL QLLCENHNRD LQNFLRCQNN KTNYNLVCET LQFLDIMCGS TTG GLGLLG LYINEDNVGL VIQTLETLTE YCQGPCHENQ TCIVTHESNG IDIITALILN DISPLCKYRM DLVLQLKDNA SKLL LALME SRHDSENAER ILISLRPQEL VDVIKKAYLQ EEERENSEVS PREVGHNIYI LALQLSRHNK QLQHLLKPVK RIQEE EAEG ISSMLSLNNK QLSQMLKSSA PAQEEEEDPL AYYENHTSQI EIVRQDRSME QIVFPVPGIC QFLTEETKHR LFTTTE QDE QGSKVSDFFD QSSFLHNEME WQRKLRSMPL IYWFSRRMTL WGSISFNLAV FINIIIAFFY PYMEGASTGV LDSPLIS LL FWILICFSIA ALFTKRYSIR PLIVALILRS IYYLGIGPTL NILGALNLTN KIVFVVSFVG NRGTFIRGYK AMVMDMEF L YHVGYILTSV LGLFAHELFY SILLFDLIYR EETLFNVIKS VTRNGRSILL TALLALILVY LFSIVGFLFL KDDFILEVD RLPNNHSTAS PLGMPHGAAA FVDTCSGDKM DCVSGLSVPE VLEEDRELDS TERACDTLLM CIVTVMNHGL RNGGGVGDIL RKPSKDESL FPARVVYDLL FFFIVIIIVL NLIFGVIIDT FADLRSEKQK KEEILKTTCF ICGLERDKFD NKTVSFEEHI K LEHNMWNY LYFIVLVRVK NKTDYTGPES YVAQMIKNKN LDWFPRMRAM SLVSNEGEGE QNEIRILQDK LNSTMKLVSH LT AQLNELK EQMTEQRKRR QRLGFVDVQN CISR UniProtKB: Inositol 1,4,5-trisphosphate receptor type 3 |
-分子 #2: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 2 / コピー数: 4 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-分子 #3: D-MYO-INOSITOL-1,4,5-TRIPHOSPHATE
| 分子 | 名称: D-MYO-INOSITOL-1,4,5-TRIPHOSPHATE / タイプ: ligand / ID: 3 / コピー数: 4 / 式: I3P |
|---|---|
| 分子量 | 理論値: 420.096 Da |
| Chemical component information | 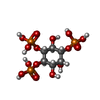 ChemComp-I3P: |
-分子 #4: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 4 / コピー数: 4 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-分子 #5: 1,2-DIOLEOYL-SN-GLYCERO-3-PHOSPHOCHOLINE
| 分子 | 名称: 1,2-DIOLEOYL-SN-GLYCERO-3-PHOSPHOCHOLINE / タイプ: ligand / ID: 5 / コピー数: 24 / 式: PCW |
|---|---|
| 分子量 | 理論値: 787.121 Da |
| Chemical component information |  ChemComp-PCW: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 20 mg/mL |
|---|---|
| 緩衝液 | pH: 8 詳細: 120 mM NaCl, 50 mM Tris-HCl pH 8.0, 0.02% LMNG, 2 mM dithiothreitol, 2 mM EDTA, 2 mM EGTA, 2 mM BAPTA, 2 mM HEDTA, 1 mM ATP, 500 uM fluorinated fos-choline-8, 3 mM free Mg2+, 1-10000 nM free Ca2+ |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 400 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: OTHER |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV 詳細: Custom-made calcium free blotting paper (see publication for details).. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 6 / 実像数: 17733 / 平均露光時間: 3.0 sec. / 平均電子線量: 66.0 e/Å2 詳細: Images were collected at 0.826 A/px magnification on an FEI Krios with Gatan K3 detector at 15 e-/pix/sec with 3 sec exposure (0.05 sec/frame) for a total dose of 66 e-/A2 in automated ...詳細: Images were collected at 0.826 A/px magnification on an FEI Krios with Gatan K3 detector at 15 e-/pix/sec with 3 sec exposure (0.05 sec/frame) for a total dose of 66 e-/A2 in automated fashion using SerialEM. Five datasets were collected during the same session for each Ca2+ concentration on a series of grids that were prepared sequentially resulting in 637 movies at 1 nM, 2150 movies at 10 nM, 6126 movies at 100 nM, 1372 movies at 1 uM, and 3136 movies at 10 uM. A sixth dataset of 4312 movies collected at nominal 100 nM free Ca2+ from a grid prepared later in the sequence was collected as a technical replicate to assess experimental error. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最大 デフォーカス(補正後): 4.3 µm 最小 デフォーカス(補正後): 0.7000000000000001 µm 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 29000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: INSILICO MODEL |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C4 (4回回転対称) / 解像度のタイプ: BY AUTHOR / 解像度: 2.7 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC / 使用した粒子像数: 767556 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
-原子モデル構築 1
| 精密化 | 空間: RECIPROCAL / プロトコル: AB INITIO MODEL / 当てはまり具合の基準: Map to Model FSC |
|---|---|
| 得られたモデル |  PDB-8tk8: |
 ムービー
ムービー コントローラー
コントローラー






















































 Z
Z Y
Y X
X