+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | 2up-1 conformation of HKU1-B S protein after incubation of the receptor | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | HCoV-HKU1 / VIRAL PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報endocytosis involved in viral entry into host cell / host cell endoplasmic reticulum-Golgi intermediate compartment membrane / receptor-mediated virion attachment to host cell / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / host cell plasma membrane / virion membrane / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Human coronavirus HKU1 (isolate N2) (ウイルス) Human coronavirus HKU1 (isolate N2) (ウイルス) | |||||||||
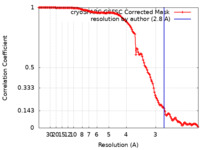
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.8 Å | |||||||||
 データ登録者 データ登録者 | Xia LY / Zhang YY / Zhou Q | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell Res / 年: 2024 ジャーナル: Cell Res / 年: 2024タイトル: Structural basis for the recognition of HCoV-HKU1 by human TMPRSS2. 著者: Lingyun Xia / Yuanyuan Zhang / Qiang Zhou /  | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_38831.map.gz emd_38831.map.gz | 230.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-38831-v30.xml emd-38831-v30.xml emd-38831.xml emd-38831.xml | 14.7 KB 14.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_38831_fsc.xml emd_38831_fsc.xml | 14.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_38831.png emd_38831.png | 94.4 KB | ||
| Filedesc metadata |  emd-38831.cif.gz emd-38831.cif.gz | 6.2 KB | ||
| その他 |  emd_38831_half_map_1.map.gz emd_38831_half_map_1.map.gz emd_38831_half_map_2.map.gz emd_38831_half_map_2.map.gz | 226.6 MB 226.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-38831 http://ftp.pdbj.org/pub/emdb/structures/EMD-38831 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-38831 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-38831 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_38831_validation.pdf.gz emd_38831_validation.pdf.gz | 893 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_38831_full_validation.pdf.gz emd_38831_full_validation.pdf.gz | 892.6 KB | 表示 | |
| XML形式データ |  emd_38831_validation.xml.gz emd_38831_validation.xml.gz | 22.3 KB | 表示 | |
| CIF形式データ |  emd_38831_validation.cif.gz emd_38831_validation.cif.gz | 28.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-38831 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-38831 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-38831 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-38831 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8y1cMC  8y19C  8y1aC  8y1bC  8y1dC  8y1eC  8y1fC  8y1gC  8y1hC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_38831.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_38831.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.087 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_38831_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_38831_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : 2up-1 conformation of HKU1-B S protein after incubation of the re...
| 全体 | 名称: 2up-1 conformation of HKU1-B S protein after incubation of the receptor |
|---|---|
| 要素 |
|
-超分子 #1: 2up-1 conformation of HKU1-B S protein after incubation of the re...
| 超分子 | 名称: 2up-1 conformation of HKU1-B S protein after incubation of the receptor タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Human coronavirus HKU1 (isolate N2) (ウイルス) Human coronavirus HKU1 (isolate N2) (ウイルス) |
-分子 #1: Spike glycoprotein
| 分子 | 名称: Spike glycoprotein / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Human coronavirus HKU1 (isolate N2) (ウイルス) Human coronavirus HKU1 (isolate N2) (ウイルス) |
| 分子量 | 理論値: 143.475344 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MFLIIFILPT TLAVIGDFNC TNSFINDYNK TIPRISEDVV DVSLGLGTYY VLNRVYLNTT LLFTGYFPKS GANFRDLALK GSKYLSTLW YKPPFLSDFN NGIFSKVKNT KLYVNNTLYS EFSTIVIGSV FVNTSYTIVV QPHNGILEIT ACQYTMCEYP H TVCKSKGS ...文字列: MFLIIFILPT TLAVIGDFNC TNSFINDYNK TIPRISEDVV DVSLGLGTYY VLNRVYLNTT LLFTGYFPKS GANFRDLALK GSKYLSTLW YKPPFLSDFN NGIFSKVKNT KLYVNNTLYS EFSTIVIGSV FVNTSYTIVV QPHNGILEIT ACQYTMCEYP H TVCKSKGS IRNESWHIDS SEPLCLFKKN FTYNVSADWL YFHFYQERGV FYAYYADVGM PTTFLFSLYL GTILSHYYVM PL TCKAISS NTDNETLEYW VTPLSRRQYL LNFDEHGVIT NAVDCSSSFL SEIQCKTQSF APNTGVYDLS GFTVKPVATV YRR IPNLPD CDIDNWLNNV SVPSPLNWER RIFSNCNFNL STLLRLVHVD SFSCNNLDKS KIFGSCFNSI TVDKFAIPNR RRDD LQLGS SGFLQSSNYK IDISSSSCQL YYSLPLVNVT INNFNPSSWN RRYGFGSFNV SSYDVVYSDH CFSVNSDFCP CADPS VVNS CVKSKPLSAI CPAGTKYRHC DLDTTLYVNN WCRCSCLPDP ISTYSPNTCP QKKVVVGIGE HCPGLGINEE KCGTQL NHS SCSCSPDAFL GWSFDSCISN NRCNIFSNFI FNGINSGTTC SNDLLYSNTE VSTGVCVNYD LYGITGQGIF KEVSAAY YN NWQNLLYDSN GNIIGFKDFL TNKTYTILPC YSGRVSAAFY QNSSSPALLY RNLKCSYVLN NISFISQPFY FDSYLGCV L NAVNLTSYSV SSCDLRMGSG FCIDYALPSS GSASRGISSP YRFVTFEPFN VSFVNDSVET VGGLFEIQIP TNFTIAGHE EFIQTSSPKV TIDCSAFVCS NYAACHDLLS EYGTFCDNIN SILNEVNDLL DITQLQVANA LMQGVTLSSN LNTNLHSDVD NIDFKSLLG CLGSQCGSSS RSLLEDLLFN KVKLSDVGFV EAYNNCTGGS EIRDLLCVQS FNGIKVLPPI LSETQISGYT T AATVAAMF PPWSAAAGVP FSLNVQYRIN GLGVTMDVLN KNQKLIANAF NKALLSIQNG FTATNSALAK IQSVVNANAQ AL NSLLQQL FNKFGAISSS LQEILSRLDP PEAQVQIDRL INGRLTALNA YVSQQLSDIT LIKAGASRAI EKVNECVKSQ SPR INFCGN GNHILSLVQN APYGLLFIHF SYKPTSFKTV LVSPGLCLSG DRGIAPKQGY FIKQNDSWMF TGSSYYYPEP ISDK NVVFM NSCSVNFTKA PFIYLNNSIP NLSDFEAEFS LWFKNHTSIA PNLTFNSHIN ATFLDLYYEM NVIQESIKSL NSSFI NLKE IGTYEM UniProtKB: Spike glycoprotein |
-分子 #4: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 4 / コピー数: 51 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)