+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of a frog VMAT2 in an apo conformation | |||||||||
 マップデータ マップデータ | XlVMAT2 WT. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Vesicular monoamine transporter / SLC18A2 / MFS / fold / TRANSPORT PROTEIN | |||||||||
| 生物種 | ||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.0 Å | |||||||||
 データ登録者 データ登録者 | Lyu Y / Fu C / Ma H / Sun Z / Su Z / Zhou X | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Engineering of a mammalian VMAT2 for cryo-EM analysis results in non-canonical protein folding. 著者: Ying Lyu / Chunting Fu / Haiyun Ma / Zhaoming Su / Ziyi Sun / Xiaoming Zhou /  要旨: Vesicular monoamine transporter 2 (VMAT2) belongs to the major facilitator superfamily (MFS), and mediates cytoplasmic monoamine packaging into presynaptic vesicles. Here, we present two cryo-EM ...Vesicular monoamine transporter 2 (VMAT2) belongs to the major facilitator superfamily (MFS), and mediates cytoplasmic monoamine packaging into presynaptic vesicles. Here, we present two cryo-EM structures of VMAT2, with a frog VMAT2 adopting a canonical MFS fold and an engineered sheep VMAT2 adopting a non-canonical fold. Both VMAT2 proteins mediate uptake of a selective fluorescent VMAT2 substrate into cells. Molecular docking, substrate binding and transport analysis reveal potential substrate binding mechanism in VMAT2. Meanwhile, caution is advised when interpreting engineered membrane protein structures. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_38390.map.gz emd_38390.map.gz | 59.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-38390-v30.xml emd-38390-v30.xml emd-38390.xml emd-38390.xml | 15.3 KB 15.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
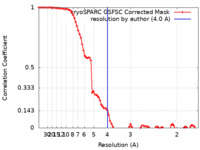
| FSC (解像度算出) |  emd_38390_fsc.xml emd_38390_fsc.xml | 8.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_38390.png emd_38390.png | 85.7 KB | ||
| Filedesc metadata |  emd-38390.cif.gz emd-38390.cif.gz | 5.7 KB | ||
| その他 |  emd_38390_half_map_1.map.gz emd_38390_half_map_1.map.gz emd_38390_half_map_2.map.gz emd_38390_half_map_2.map.gz | 59.5 MB 59.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-38390 http://ftp.pdbj.org/pub/emdb/structures/EMD-38390 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-38390 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-38390 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_38390_validation.pdf.gz emd_38390_validation.pdf.gz | 945.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_38390_full_validation.pdf.gz emd_38390_full_validation.pdf.gz | 944.9 KB | 表示 | |
| XML形式データ |  emd_38390_validation.xml.gz emd_38390_validation.xml.gz | 16.3 KB | 表示 | |
| CIF形式データ |  emd_38390_validation.cif.gz emd_38390_validation.cif.gz | 21 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-38390 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-38390 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-38390 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-38390 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_38390.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_38390.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | XlVMAT2 WT. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.85 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half map B.
| ファイル | emd_38390_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B. | ||||||||||||
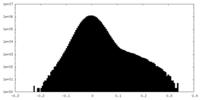
| 投影像・断面図 |
| ||||||||||||
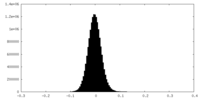
| 密度ヒストグラム |
-ハーフマップ: Half map A.
| ファイル | emd_38390_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A. | ||||||||||||
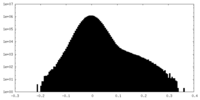
| 投影像・断面図 |
| ||||||||||||
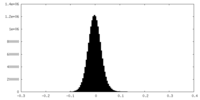
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : XlVMAT2 WT
| 全体 | 名称: XlVMAT2 WT |
|---|---|
| 要素 |
|
-超分子 #1: XlVMAT2 WT
| 超分子 | 名称: XlVMAT2 WT / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種: |
-分子 #1: XlVMAT2
| 分子 | 名称: XlVMAT2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 分子量 | 理論値: 55.624984 KDa |
| 組換発現 | 生物種:  Komagataella pastoris (菌類) Komagataella pastoris (菌類) |
| 配列 | 文字列: MSMTEFTLLR WLREGRQSRK LILLIVFIAL LLDNMLLTVV VPIIPSYLYS MTHESNNTHD GSIPPAAPSG FHSIFSYYDN TTVITTNFT SSDQLQQSAL TPTTSPSLAT PPASADCPKA DSSLLNENVK VGLLFASKAT VQLLTNPFIG PMTNRIGYQI P MFAGFCIM ...文字列: MSMTEFTLLR WLREGRQSRK LILLIVFIAL LLDNMLLTVV VPIIPSYLYS MTHESNNTHD GSIPPAAPSG FHSIFSYYDN TTVITTNFT SSDQLQQSAL TPTTSPSLAT PPASADCPKA DSSLLNENVK VGLLFASKAT VQLLTNPFIG PMTNRIGYQI P MFAGFCIM FVSTIMFAFS GTYTLLFIAR SLQGIGSSCS SVAGMGMLAS VYTDDEERGN AMGVALGGLA MGVLVGPPFG SI LYEFVGK TAPFIVLAVL VLFDGALQLF VLQPSRVQPE SQTGTPLLTL IRDPYILIAA GSICFANMAI AMLEPALPIW MME TMCSRK WQLGVAFLPA SISYLLGTNI FGPLAHKMGR WLCAFIGMIM VGISIICVPF ARNIYGLIAP NFGVGFAIGM VDSS MMPIM GYLVDLRHVS VYGSVYAIAD VAFCMGFAFG PSAGGAIAKS IGFPWLMTII GVVDILFAPL CLFLRSPPAR EEKMA ILMD HKCPVKTKMY TQNSGQPYYT GEEEESESDE |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 8.0 mg/mL |
|---|---|
| 緩衝液 | pH: 6 / 詳細: 150 mm NaCl, 20 mM MES-Na pH 6.0, 0.4 mM DDM |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV / 詳細: blot for 3 s before plunging. |
| 詳細 | This sample was monodisperse. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.6 µm / 最小 デフォーカス(公称値): 0.9 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: AlphaFold / Chain - Initial model type: in silico model |
|---|---|
| 得られたモデル |  PDB-8xiu: |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)