+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of H. thermophilus GroEL-GroES2 asymmetric football complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Chaperone / Chaperonin / ATPase / protein folding | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報chaperonin ATPase / : / isomerase activity / protein folding chaperone / ATP-dependent protein folding chaperone / unfolded protein binding / protein-folding chaperone binding / protein refolding / ATP binding / metal ion binding / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |   Hydrogenobacter thermophilus TK-6 (バクテリア) Hydrogenobacter thermophilus TK-6 (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.6 Å | |||||||||
 データ登録者 データ登録者 | Liao Z / Gopalasingam CC / Kameya M / Gerle C / Shigematsu H / Ishii M / Arakawa T / Fushinobu S | |||||||||
| 資金援助 |  日本, 2件 日本, 2件
| |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2024 ジャーナル: Structure / 年: 2024タイトル: Structural insights into thermophilic chaperonin complexes. 著者: Zengwei Liao / Chai C Gopalasingam / Masafumi Kameya / Christoph Gerle / Hideki Shigematsu / Masaharu Ishii / Takatoshi Arakawa / Shinya Fushinobu /  要旨: Group I chaperonins are dual heptamer protein complexes that play significant roles in protein homeostasis. The structure and function of the Escherichia coli chaperonin are well characterized. ...Group I chaperonins are dual heptamer protein complexes that play significant roles in protein homeostasis. The structure and function of the Escherichia coli chaperonin are well characterized. However, the dynamic properties of chaperonins, such as large ATPase-dependent conformational changes by binding of lid-like co-chaperonin GroES, have made structural analyses challenging, and our understanding of these changes during the turnover of chaperonin complex formation is limited. In this study, we used single-particle cryogenic electron microscopy to investigate the structures of GroES-bound chaperonin complexes from the thermophilic hydrogen-oxidizing bacteria Hydrogenophilus thermoluteolus and Hydrogenobacter thermophilus in the presence of ATP and AMP-PNP. We captured the structure of an intermediate state chaperonin complex, designated as an asymmetric football-shaped complex, and performed analyses to decipher the dynamic structural variations. Our structural analyses of inter- and intra-subunit communications revealed a unique mechanism of complex formation through the binding of a second GroES to a bullet-shaped complex. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_37862.map.gz emd_37862.map.gz | 256.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-37862-v30.xml emd-37862-v30.xml emd-37862.xml emd-37862.xml | 26.3 KB 26.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_37862.png emd_37862.png | 67.8 KB | ||
| マスクデータ |  emd_37862_msk_1.map emd_37862_msk_1.map | 512 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-37862.cif.gz emd-37862.cif.gz | 6.8 KB | ||
| その他 |  emd_37862_additional_1.map.gz emd_37862_additional_1.map.gz emd_37862_additional_2.map.gz emd_37862_additional_2.map.gz emd_37862_half_map_1.map.gz emd_37862_half_map_1.map.gz emd_37862_half_map_2.map.gz emd_37862_half_map_2.map.gz | 484 MB 458.1 MB 474.5 MB 474.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-37862 http://ftp.pdbj.org/pub/emdb/structures/EMD-37862 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37862 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37862 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_37862_validation.pdf.gz emd_37862_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_37862_full_validation.pdf.gz emd_37862_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_37862_validation.xml.gz emd_37862_validation.xml.gz | 19 KB | 表示 | |
| CIF形式データ |  emd_37862_validation.cif.gz emd_37862_validation.cif.gz | 22.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37862 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37862 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37862 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37862 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_37862.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_37862.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.752 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_37862_msk_1.map emd_37862_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
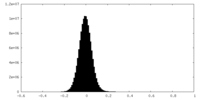
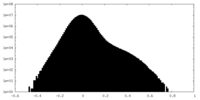
| 密度ヒストグラム |
-追加マップ: #1
| ファイル | emd_37862_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
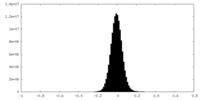
| 密度ヒストグラム |
-追加マップ: #2
| ファイル | emd_37862_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
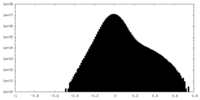
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_37862_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_37862_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : H. thermophilus GroEL-GroES2 Asymmetric football complex
| 全体 | 名称: H. thermophilus GroEL-GroES2 Asymmetric football complex |
|---|---|
| 要素 |
|
-超分子 #1: H. thermophilus GroEL-GroES2 Asymmetric football complex
| 超分子 | 名称: H. thermophilus GroEL-GroES2 Asymmetric football complex タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:   Hydrogenobacter thermophilus TK-6 (バクテリア) Hydrogenobacter thermophilus TK-6 (バクテリア) |
-超分子 #2: Chaperonin GroEL
| 超分子 | 名称: Chaperonin GroEL / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|
-超分子 #3: Co-chaperonin GroES
| 超分子 | 名称: Co-chaperonin GroES / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|
-分子 #1: Chaperonin GroEL
| 分子 | 名称: Chaperonin GroEL / タイプ: protein_or_peptide / ID: 1 / コピー数: 14 / 光学異性体: LEVO / EC番号: chaperonin ATPase |
|---|---|
| 由来(天然) | 生物種:   Hydrogenobacter thermophilus TK-6 (バクテリア) Hydrogenobacter thermophilus TK-6 (バクテリア)株: TK-6 |
| 分子量 | 理論値: 56.600918 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: AAKKVIYGED ARARLKAGVD KLANAVKVTL GPRGREVIIE KKWGTPVVTK DGVTVAKEIE FKDPYENMGA QLVKEVASKT SDVAGDGTT TATVLAQAIF NEGLRAIASG ANPMDIKRGI DKAVETVVNE IKKLSIPVSG RKEIEQVATI SANNDATIGK I IADAMEAV ...文字列: AAKKVIYGED ARARLKAGVD KLANAVKVTL GPRGREVIIE KKWGTPVVTK DGVTVAKEIE FKDPYENMGA QLVKEVASKT SDVAGDGTT TATVLAQAIF NEGLRAIASG ANPMDIKRGI DKAVETVVNE IKKLSIPVSG RKEIEQVATI SANNDATIGK I IADAMEAV GKDGVITVEE SKSAETTLET VQGMQFDRGY LSPYFVTNPD KMEAVLEDPF ILIYEKKISN VKDLLPVLEN VV RAGKPLL IIAEDVEAEA LATLVVNHIK GVIRACAVKA PGFGQRRKDY LQDIAILTGG TAITEELGIK LESVTLDMLG RAD KVIVDK DNTTIVGGKG SKEAIQARIE QIKRQILETT SDYDREKLQE RLAKLSGGVA IIRVGAATEA ELKEKKARVE DAVH ATKAA VEEGIVPGGG VALVRASEAL DNLKVDNADQ QLGIDIIKKA CRTPIRQIAA NSGFEGYVVL EKVLQLGKEK GKNWG FDAG VGDYKDMVEA GIIDPTKVVR VAIQNAASVA GTMLTAEALV AEIPE UniProtKB: Chaperonin GroEL |
-分子 #2: Co-chaperonin GroES
| 分子 | 名称: Co-chaperonin GroES / タイプ: protein_or_peptide / ID: 2 / コピー数: 14 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Hydrogenobacter thermophilus TK-6 (バクテリア) Hydrogenobacter thermophilus TK-6 (バクテリア)株: TK-6 |
| 分子量 | 理論値: 10.642385 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MARLRPLYDK IVVKRMEEQE QKTPSGIIIP DTAKEKPQIG EVIAVGDGKL LSNGQIVSPK VKKGDKVVFN KYAGTEVELD GEKYLIMSE DEVLAVI UniProtKB: Co-chaperonin GroES |
-分子 #3: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 3 / コピー数: 14 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #4: PHOSPHOAMINOPHOSPHONIC ACID-ADENYLATE ESTER
| 分子 | 名称: PHOSPHOAMINOPHOSPHONIC ACID-ADENYLATE ESTER / タイプ: ligand / ID: 4 / コピー数: 14 / 式: ANP |
|---|---|
| 分子量 | 理論値: 506.196 Da |
| Chemical component information |  ChemComp-ANP: |
-分子 #5: POTASSIUM ION
| 分子 | 名称: POTASSIUM ION / タイプ: ligand / ID: 5 / コピー数: 7 / 式: K |
|---|---|
| 分子量 | 理論値: 39.098 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 構成要素:
| ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 10 sec. / 前処理 - 雰囲気: OTHER / 前処理 - 気圧: 0.007 kPa 詳細: glow discharged at 7 Pa, 10 mA, 10 sec by a JEOL JEC-3000FC auto fine coater | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 281 K / 装置: FEI VITROBOT MARK IV / 詳細: blot force 10, blot time 3 s. | ||||||||||||||||||
| 詳細 | 2.5 mg/mL GroEL and 0.46 mg/mL GroES was incubated on a heat block at 45C for 10 min. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL CRYO ARM 300 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 平均露光時間: 2.264 sec. / 平均電子線量: 49.97 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.6 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 60000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)