+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | PSI-LHCI of the red alga Cyanidium caldarium RK-1 (NIES-2137) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Photosystem I / ELECTRON TRANSPORT / PHOTOSYNTHESIS | |||||||||
| 生物種 |  Cyanidium caldarium (真核生物) Cyanidium caldarium (真核生物) | |||||||||
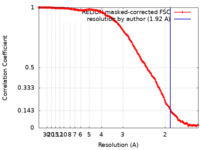
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 1.92 Å | |||||||||
 データ登録者 データ登録者 | Kato K / Hamaguchi T / Nakajima Y / Kawakami K / Yonekura K / Shen JR / Nagao R | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2024 ジャーナル: Proc Natl Acad Sci U S A / 年: 2024タイトル: The structure of PSI-LHCI from provides evolutionary insights into conservation and diversity of red-lineage LHCs. 著者: Koji Kato / Tasuku Hamaguchi / Minoru Kumazawa / Yoshiki Nakajima / Kentaro Ifuku / Shunsuke Hirooka / Yuu Hirose / Shin-Ya Miyagishima / Takehiro Suzuki / Keisuke Kawakami / Naoshi Dohmae / ...著者: Koji Kato / Tasuku Hamaguchi / Minoru Kumazawa / Yoshiki Nakajima / Kentaro Ifuku / Shunsuke Hirooka / Yuu Hirose / Shin-Ya Miyagishima / Takehiro Suzuki / Keisuke Kawakami / Naoshi Dohmae / Koji Yonekura / Jian-Ren Shen / Ryo Nagao /  要旨: Light-harvesting complexes (LHCs) are diversified among photosynthetic organisms, and the structure of the photosystem I-LHC (PSI-LHCI) supercomplex has been shown to be variable depending on the ...Light-harvesting complexes (LHCs) are diversified among photosynthetic organisms, and the structure of the photosystem I-LHC (PSI-LHCI) supercomplex has been shown to be variable depending on the species of organisms. However, the structural and evolutionary correlations of red-lineage LHCs are unknown. Here, we determined a 1.92-Å resolution cryoelectron microscopic structure of a PSI-LHCI supercomplex isolated from the red alga RK-1 (NIES-2137), which is an important taxon in the Cyanidiophyceae. We subsequently investigated the correlations of PSI-LHCIs from different organisms through structural comparisons and phylogenetic analysis. The PSI-LHCI structure obtained shows five LHCI subunits surrounding a PSI-monomer core. The five LHCIs are composed of two Lhcr1s, two Lhcr2s, and one Lhcr3. Phylogenetic analysis of LHCs bound to PSI in the red-lineage algae showed clear orthology of LHCs between and , whereas no orthologous relationships were found between Lhcr1-3 and LHCs in other red-lineage PSI-LHCI structures. These findings provide evolutionary insights into conservation and diversity of red-lineage LHCs associated with PSI. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_37480.map.gz emd_37480.map.gz | 18 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-37480-v30.xml emd-37480-v30.xml emd-37480.xml emd-37480.xml | 35.8 KB 35.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_37480_fsc.xml emd_37480_fsc.xml | 14 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_37480.png emd_37480.png | 110.9 KB | ||
| Filedesc metadata |  emd-37480.cif.gz emd-37480.cif.gz | 9.1 KB | ||
| その他 |  emd_37480_additional_1.map.gz emd_37480_additional_1.map.gz emd_37480_half_map_1.map.gz emd_37480_half_map_1.map.gz emd_37480_half_map_2.map.gz emd_37480_half_map_2.map.gz | 52.2 MB 59.4 MB 59.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-37480 http://ftp.pdbj.org/pub/emdb/structures/EMD-37480 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37480 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37480 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_37480_validation.pdf.gz emd_37480_validation.pdf.gz | 980 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_37480_full_validation.pdf.gz emd_37480_full_validation.pdf.gz | 979.6 KB | 表示 | |
| XML形式データ |  emd_37480_validation.xml.gz emd_37480_validation.xml.gz | 18.1 KB | 表示 | |
| CIF形式データ |  emd_37480_validation.cif.gz emd_37480_validation.cif.gz | 24.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37480 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37480 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37480 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37480 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_37480.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_37480.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.792 Å | ||||||||||||||||||||
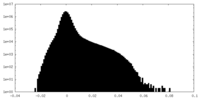
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: #1
| ファイル | emd_37480_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
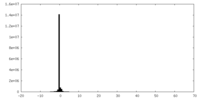
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_37480_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
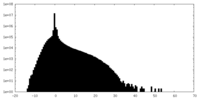
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_37480_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
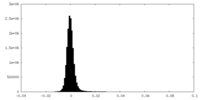
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : PSI-LHCI
+超分子 #1: PSI-LHCI
+分子 #1: Photosystem I P700 chlorophyll a apoprotein A1
+分子 #2: Photosystem I P700 chlorophyll a apoprotein A2
+分子 #3: Photosystem I iron-sulfur center
+分子 #4: Photosystem I reaction center subunit II
+分子 #5: Photosystem I reaction center subunit IV
+分子 #6: Photosystem I reaction center subunit III
+分子 #7: Photosystem I reaction center subunit VIII
+分子 #8: Photosystem I reaction center subunit IX
+分子 #9: Photosystem I reaction center subunit X
+分子 #10: Photosystem I reaction center subunit XI
+分子 #11: Photosystem I reaction center subunit XII
+分子 #12: Photosystem I subunit O
+分子 #13: Lhcr1
+分子 #14: Lhcr2
+分子 #15: Lhcr3
+分子 #16: CHLOROPHYLL A ISOMER
+分子 #17: CHLOROPHYLL A
+分子 #18: PHYLLOQUINONE
+分子 #19: 1,2-DIPALMITOYL-PHOSPHATIDYL-GLYCEROLE
+分子 #20: BETA-CAROTENE
+分子 #21: IRON/SULFUR CLUSTER
+分子 #22: DODECYL-BETA-D-MALTOSIDE
+分子 #23: UNKNOWN LIGAND
+分子 #24: DIGALACTOSYL DIACYL GLYCEROL (DGDG)
+分子 #25: (1~{R})-3,5,5-trimethyl-4-[(1~{E},3~{E},5~{E},7~{E},9~{E},11~{E},...
+分子 #26: water
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.83 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 6.5 / 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil R0.6/1 / メッシュ: 200 | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL CRYO ARM 300 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 検出モード: COUNTING / 平均電子線量: 39.8 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.8 µm |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: Other / Chain - Initial model type: in silico model / 詳細: homology model |
|---|---|
| 精密化 | 空間: RECIPROCAL / プロトコル: FLEXIBLE FIT |
| 得られたモデル |  PDB-8wey: |
 ムービー
ムービー コントローラー
コントローラー




 Z
Z Y
Y X
X