登録情報 データベース : EMDB / ID : EMD-37247タイトル CULLIN3-KLHL22-RBX1 E3 ligase 複合体 : CULLIN3-KLHL22-RBX1 E3 ligaseタンパク質・ペプチド : Kelch-like protein 22タンパク質・ペプチド : Cullin-3タンパク質・ペプチド : E3 ubiquitin-protein ligase RBX1 / 機能・相同性 分子機能 ドメイン・相同性 構成要素
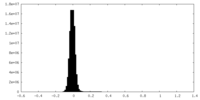
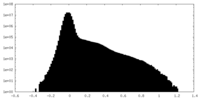
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Homo sapiens (ヒト)手法 / / 解像度 : 3.67 Å Su M-Y 資金援助 Organization Grant number 国 Other government 2022A1515010856
ジャーナル : Structure / 年 : 2023タイトル : Cryo-EM structure of the KLHL22 E3 ligase bound to an oligomeric metabolic enzyme.著者 : Fei Teng / Yang Wang / Ming Liu / Shuyun Tian / Goran Stjepanovic / Ming-Yuan Su / 要旨 : CULLIN-RING ligases constitute the largest group of E3 ubiquitin ligases. While some CULLIN family members recruit adapters before engaging further with different substrate receptors, homo-dimeric ... CULLIN-RING ligases constitute the largest group of E3 ubiquitin ligases. While some CULLIN family members recruit adapters before engaging further with different substrate receptors, homo-dimeric BTB-Kelch family proteins combine adapter and substrate receptor into a single polypeptide for the CULLIN3 family. However, the entire structural assembly and molecular details have not been elucidated to date. Here, we present a cryo-EM structure of the CULLIN3 in complex with Kelch-like protein 22 (KLHL22) and a mitochondrial glutamate dehydrogenase complex I (GDH1) at 3.06 Å resolution. The structure adopts a W-shaped architecture formed by E3 ligase dimers. Three CULLIN3 dimers were found to be dynamically associated with a single GDH1 hexamer. CULLIN3 ligase mediated the polyubiquitination of GDH1 in vitro. Together, these results enabled the establishment of a structural model for understanding the complete assembly of BTB-Kelch proteins with CULLIN3 and how together they recognize oligomeric substrates and target them for ubiquitination. 履歴 登録 2023年8月22日 - ヘッダ(付随情報) 公開 2023年12月13日 - マップ公開 2023年12月13日 - 更新 2023年12月13日 - 現状 2023年12月13日 処理サイト : PDBc / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報
 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 中国, 1件
中国, 1件  引用
引用 ジャーナル: Structure / 年: 2023
ジャーナル: Structure / 年: 2023
 構造の表示
構造の表示 ダウンロードとリンク
ダウンロードとリンク emd_37247.map.gz
emd_37247.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-37247-v30.xml
emd-37247-v30.xml emd-37247.xml
emd-37247.xml EMDBヘッダ
EMDBヘッダ emd_37247.png
emd_37247.png emd-37247.cif.gz
emd-37247.cif.gz emd_37247_additional_1.map.gz
emd_37247_additional_1.map.gz emd_37247_half_map_1.map.gz
emd_37247_half_map_1.map.gz emd_37247_half_map_2.map.gz
emd_37247_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-37247
http://ftp.pdbj.org/pub/emdb/structures/EMD-37247 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37247
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37247 emd_37247_validation.pdf.gz
emd_37247_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_37247_full_validation.pdf.gz
emd_37247_full_validation.pdf.gz emd_37247_validation.xml.gz
emd_37247_validation.xml.gz emd_37247_validation.cif.gz
emd_37247_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37247
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37247 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37247
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37247 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_37247.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_37247.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素 Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト) 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 画像解析
画像解析 ムービー
ムービー コントローラー
コントローラー

















 Z
Z Y
Y X
X