+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | In situ structure of RNA-dependent RNA polymerase in full BAV particles | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | reovirus / RdRp / complex / inner capsid protein / VIRUS | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報viral genome replication / RNA-dependent RNA polymerase activity / nucleotide binding / RNA binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Banna virus (ウイルス) Banna virus (ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.0 Å | |||||||||
 データ登録者 データ登録者 | Li Z / Cao S | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Cryo-EM structures of Banna virus in multiple states reveal stepwise detachment of viral spikes. 著者: Zhiqiang Li / Han Xia / Guibo Rao / Yan Fu / Tingting Chong / Kexing Tian / Zhiming Yuan / Sheng Cao /  要旨: Banna virus (BAV) is the prototype Seadornavirus, a class of reoviruses for which there has been little structural study. Here, we report atomic cryo-EM structures of three states of BAV virions- ...Banna virus (BAV) is the prototype Seadornavirus, a class of reoviruses for which there has been little structural study. Here, we report atomic cryo-EM structures of three states of BAV virions-surrounded by 120 spikes (full virions), 60 spikes (partial virions), or no spikes (cores). BAV cores are double-layered particles similar to the cores of other non-turreted reoviruses, except for an additional protein component in the outer capsid shell, VP10. VP10 was identified to be a cementing protein that plays a pivotal role in the assembly of BAV virions by directly interacting with VP2 (inner capsid), VP8 (outer capsid), and VP4 (spike). Viral spikes (VP4/VP9 heterohexamers) are situated on top of VP10 molecules in full or partial virions. Asymmetrical electrostatic interactions between VP10 monomers and VP4 trimers are disrupted by high pH treatment, which is thus a simple way to produce BAV cores. Low pH treatment of BAV virions removes only the flexible receptor binding protein VP9 and triggers significant conformational changes in the membrane penetration protein VP4. BAV virions adopt distinct spatial organization of their surface proteins compared with other well-studied reoviruses, suggesting that BAV may have a unique mechanism of penetration of cellular endomembranes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_36871.map.gz emd_36871.map.gz | 306.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-36871-v30.xml emd-36871-v30.xml emd-36871.xml emd-36871.xml | 16.1 KB 16.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_36871_fsc.xml emd_36871_fsc.xml | 15.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_36871.png emd_36871.png | 176.7 KB | ||
| Filedesc metadata |  emd-36871.cif.gz emd-36871.cif.gz | 6.5 KB | ||
| その他 |  emd_36871_half_map_1.map.gz emd_36871_half_map_1.map.gz emd_36871_half_map_2.map.gz emd_36871_half_map_2.map.gz | 277.4 MB 277.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-36871 http://ftp.pdbj.org/pub/emdb/structures/EMD-36871 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36871 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36871 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_36871_validation.pdf.gz emd_36871_validation.pdf.gz | 1.3 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_36871_full_validation.pdf.gz emd_36871_full_validation.pdf.gz | 1.3 MB | 表示 | |
| XML形式データ |  emd_36871_validation.xml.gz emd_36871_validation.xml.gz | 23.5 KB | 表示 | |
| CIF形式データ |  emd_36871_validation.cif.gz emd_36871_validation.cif.gz | 31.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36871 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36871 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36871 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36871 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8k43MC  8k42C  8k44C  8k49C  8k4aC  8w9pC  8w9qC  8w9rC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_36871.map.gz / 形式: CCP4 / 大きさ: 347.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_36871.map.gz / 形式: CCP4 / 大きさ: 347.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.95 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_36871_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
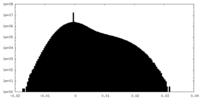
| 投影像・断面図 |
| ||||||||||||
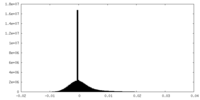
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_36871_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
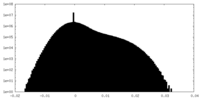
| 投影像・断面図 |
| ||||||||||||
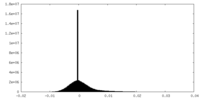
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Banna virus
| 全体 | 名称:  Banna virus (ウイルス) Banna virus (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Banna virus
| 超分子 | 名称: Banna virus / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 77763 / 生物種: Banna virus / Sci species strain: YN15-126-01 / ウイルスタイプ: VIRION / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|
-分子 #1: RNA-directed RNA polymerase (Fragment)
| 分子 | 名称: RNA-directed RNA polymerase (Fragment) / タイプ: protein_or_peptide / ID: 1 / 詳細: OR004518 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Banna virus (ウイルス) Banna virus (ウイルス) |
| 分子量 | 理論値: 138.563344 KDa |
| 配列 | 文字列: MDVQEQFEGY LREEVDLLNK FEDSHFKQLE IFYTKSQTDH VINKDKLQFN ALPFHTTLYK EINGKRVRLG TLLNWTKAER LDTIRHESM IKDERLRRLI DFDYGWIDYA VVLQRYLDEG NTIESANILQ FDGDFVNNIK HPTQKNNFLD IKVIKECETY I LMKDDNGI ...文字列: MDVQEQFEGY LREEVDLLNK FEDSHFKQLE IFYTKSQTDH VINKDKLQFN ALPFHTTLYK EINGKRVRLG TLLNWTKAER LDTIRHESM IKDERLRRLI DFDYGWIDYA VVLQRYLDEG NTIESANILQ FDGDFVNNIK HPTQKNNFLD IKVIKECETY I LMKDDNGI RDPDILRAWE LNSIPEVIEL DVDGETKKFN LRKEMIKRIQ DEAPVYFFCN PYRYAVANLD PNIPEVRLWL EY FIGSEDF FGFNGPNVIV SKSLLAAKRF EVVVNHLRKV AAFELDVESK EVMNEWIKYI MIDPVYRSYK NRGAFGNLNQ HVF ARTKSE GLSWSLIDIG TTNFELKPTK KVAGSYVNKF NLVDDVLVEE SLKDLRNEGL HKMADVTRRM IDADITPENV KKGK LNRLA LSYCGYTGSH SATAMVKQFN GTKDLDPNCD PMFVDIVKDN MRVYMQEGLQ KYPQGSRKSN RLDILFKGGT SSASS TNEH AVVNGRFRYR SELYRERDVN SSTVFKTATP GQYRVVKKIS AKLKSKNANI VTHPMNFINF KVDDLDIVVN AGSRLV RGT RAKRIITPNY GTIYAASLMT VLPAVRLLSS RASNMGALST QGRIGTTYHG ALPHDVMAPQ LAVTSSDDVS KICVAKD FG QFDTSQWGQI SKAHADGVRS MKAHYSMGHD TLVDLDLNDA SFADLLEVTA MSYERPLKYK MNGLVCESAG VKSGELTT Q TRNTTTNISH STVALDDYNN RAYRLNLPKL ELVTDNKVGD DSVEVLRVVD GSPLTPEIAK LYVNCMQDHA DKNHLEISA KRTIVGNNVA EHIKIWVFKG YLALDVFLDS VTSEKNSFSN LNYLEQVNIL YDMAMTLMIR YCSVQACMTQ FCNDMKLLNG IRAGNYTFI PTPKIICAYG TPEICLRAPE IRSFGRYLPI DEDEYSVLND LVASLSTNKP KMDFVAQMFE QNGNQVHGIW L DHFKRKND VNPDGGGIHI SEGLKRLMPE YCERHLNELV YKTLDDKVIR DYTSDIIITN ICKGKLSKAP KLAFFANFYL SL TGFNGVD SPYLTADEGV KNVHRVIGLS YRNTLSTSPT ANVDRILRNN PGSAPAYLTG NDILGVLSDY PYQNWRTVVE LLD ITEPSA TAIIEVATNQ MHAYLADKDL NTANLFDNTS RTYDISDRTY PKFVNITSNL SNSNRRGFQL EAMKHIIYMA RRGI ATLAN THPSKIGNTV YYDY UniProtKB: VP1 |
-分子 #2: VP2
| 分子 | 名称: VP2 / タイプ: protein_or_peptide / ID: 2 / 詳細: OR004519 / コピー数: 11 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Banna virus (ウイルス) Banna virus (ウイルス) |
| 分子量 | 理論値: 108.031008 KDa |
| 配列 | 文字列: MPRKKDQVTK NDDGNQTSDV QTQDFKTAVQ PDTNTAQLIK TYSNPKQRGD KGEIIYDGGL SSKLADVVDK TTEPHNADGA VKDGRIAPV KLDLEKQKLD KLKLFETSPF DPLTIKNNQD VVDKLYATQS SSIQEVVPTK TFATELQFGV TSEDMAKIYG A VAAVSKNV ...文字列: MPRKKDQVTK NDDGNQTSDV QTQDFKTAVQ PDTNTAQLIK TYSNPKQRGD KGEIIYDGGL SSKLADVVDK TTEPHNADGA VKDGRIAPV KLDLEKQKLD KLKLFETSPF DPLTIKNNQD VVDKLYATQS SSIQEVVPTK TFATELQFGV TSEDMAKIYG A VAAVSKNV NSSVTYEVKR GTHELIKVPT IPHNLVLIQS DNGKHALIKE DLGQWPVETG ISLVNQAGVF AVQLANKLGI DK PFVLDAG SNYFTDTSFI DTRKYCTDGL SPREIQKALN RQRAYYDRPE LTISENKTLL SQSIIYPDAD GNDVSIIFSG AMS HAIFTY AQSQWNKNII KLDDYIREIT LTVPKQYRPR RFKEIEHTHG YVYRELNQGS LLPLVDANLK ESSSYYFKKL MSSI SNVPV DARTLQSATA ALAADTGQAV NRAQHVSMLT NRLTTANAPT VRAITVLTCM FKQFRIGMTY ALDPNIMDVA AATCM LLFR PAQSISDEQY RYCLQTMAVF LTNTTYDIVN NDTIDVLKMK LRNQGWPFVE RYNAVEIDMS VEPLRSPGQV GRYYNP FNI DPLTKKHVED RLEEFINQVQ VGRFRNASGN AVGTTLAAFL RACRDKTSAN WRGYSVLVSR YRSLIPNELF ESLRNIS GE YNINPQDEHS FFFALAQINA DDEFIGAIDK ESAEYLDEYA TLARDISNSL TLVKAAFGPL ERTSGSIINH ANNLNKVI N HVFADKPLIS ETMLKILTID GTTGKDGYRN WLDKLVGHNY PVYVEPVVNI MNFISARFVA DSSYFGYTNE IMIMPNHIN VPVDDRFGFR DSPFCTSLPR TIMGNDVRRI SYNVFSMMED IDDVISEGFI LYDAYFNFSY DIMTTDGVTR LKEDILIVTD TGNDIKPIH FYIYFENRND KKLRYESKMN VSYRLYIKTP ACLLPLSDYM RAQHDYVSPS SSRVYIKDPA VVYTRS UniProtKB: Vp2 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL CRYO ARM 300 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.5 µm |
 ムービー
ムービー コントローラー
コントローラー











 Z
Z Y
Y X
X