+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CNE55.664 trimer in complex with broadly neutralizing HIV antibody PGT145 | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | HIV / Envelope trimer / broadly neutralizing antibody / PGT145 / Cryo-EM / VIRAL PROTEIN | ||||||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) / Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
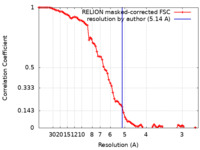
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 5.14 Å | ||||||||||||
 データ登録者 データ登録者 | Chatterjee A / Chen C / Lee K / Mangala Prasad V | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Npj Viruses / 年: 2023 ジャーナル: Npj Viruses / 年: 2023タイトル: An HIV-1 broadly neutralizing antibody overcomes structural and dynamic variation through highly focused epitope targeting. 著者: Edgar A Hodge / Ananya Chatterjee / Chengbo Chen / Gajendra S Naika / Mint Laohajaratsang / Vidya Mangala Prasad / Kelly K Lee /   要旨: The existence of broadly cross-reactive antibodies that can neutralize diverse HIV-1 isolates (bnAbs) has been appreciated for more than a decade. Many high-resolution structures of bnAbs, typically ...The existence of broadly cross-reactive antibodies that can neutralize diverse HIV-1 isolates (bnAbs) has been appreciated for more than a decade. Many high-resolution structures of bnAbs, typically with one or two well-characterized HIV-1 Env glycoprotein trimers, have been reported. However, an understanding of how such antibodies grapple with variability in their antigenic targets across diverse viral isolates has remained elusive. To achieve such an understanding requires first characterizing the extent of structural and antigenic variation embodied in Env, and then identifying how a bnAb overcomes that variation at a structural level. Here, using hydrogen/deuterium-exchange mass spectrometry (HDX-MS) and quantitative measurements of antibody binding kinetics, we show that variation in structural ordering in the V1/V2 apex of Env across a globally representative panel of HIV-1 isolates has a marked effect on antibody association rates and affinities. We also report cryo-EM reconstructions of the apex-targeting PGT145 bnAb bound to two divergent Env that exhibit different degrees of structural dynamics throughout the trimer structures. Parallel HDX-MS experiments demonstrate that PGT145 bnAb has an exquisitely focused footprint at the trimer apex where binding did not yield allosteric changes throughout the rest of the structure. These results demonstrate that structural dynamics are a cryptic determinant of antigenicity, and mature antibodies that have achieved breadth and potency in some cases are able to achieve their broad cross-reactivity by "threading the needle" and binding in a highly focused fashion, thus evading and overcoming the variable properties found in Env from divergent isolates. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_36649.map.gz emd_36649.map.gz | 45.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-36649-v30.xml emd-36649-v30.xml emd-36649.xml emd-36649.xml | 19.1 KB 19.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_36649_fsc.xml emd_36649_fsc.xml | 8.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_36649.png emd_36649.png | 25.9 KB | ||
| Filedesc metadata |  emd-36649.cif.gz emd-36649.cif.gz | 6.8 KB | ||
| その他 |  emd_36649_half_map_1.map.gz emd_36649_half_map_1.map.gz emd_36649_half_map_2.map.gz emd_36649_half_map_2.map.gz | 40.5 MB 40.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-36649 http://ftp.pdbj.org/pub/emdb/structures/EMD-36649 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36649 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36649 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_36649_validation.pdf.gz emd_36649_validation.pdf.gz | 636.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_36649_full_validation.pdf.gz emd_36649_full_validation.pdf.gz | 636.4 KB | 表示 | |
| XML形式データ |  emd_36649_validation.xml.gz emd_36649_validation.xml.gz | 14.6 KB | 表示 | |
| CIF形式データ |  emd_36649_validation.cif.gz emd_36649_validation.cif.gz | 20.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36649 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36649 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36649 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36649 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_36649.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_36649.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.35 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_36649_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_36649_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : CNE55.664 trimer in complex with broadly neutralizing HIV antibod...
| 全体 | 名称: CNE55.664 trimer in complex with broadly neutralizing HIV antibody PGT145 |
|---|---|
| 要素 |
|
-超分子 #1: CNE55.664 trimer in complex with broadly neutralizing HIV antibod...
| 超分子 | 名称: CNE55.664 trimer in complex with broadly neutralizing HIV antibody PGT145 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
-分子 #1: gp120 protein of HIV envelope trimer
| 分子 | 名称: gp120 protein of HIV envelope trimer / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 54.064277 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: AENLWVTVYY GVPVWKDAET TLFCASDAKA YETEKHNVWA THACVPTDPN PQEIHLENVT EEFNMWKNNM VEQMHTDIIS LWDQSLKPC VKLTPLCVTL QCTNVTNNIT DDMRGELKNC SFNMTTELRD KKQKVYSLFY RLDVVQINEN QGNRSNNSNK E YRLINCNT ...文字列: AENLWVTVYY GVPVWKDAET TLFCASDAKA YETEKHNVWA THACVPTDPN PQEIHLENVT EEFNMWKNNM VEQMHTDIIS LWDQSLKPC VKLTPLCVTL QCTNVTNNIT DDMRGELKNC SFNMTTELRD KKQKVYSLFY RLDVVQINEN QGNRSNNSNK E YRLINCNT SAITQACPKV SFEPIPIHYC APAGFAILKC KDKKFNGTGP CPSVSTVQCT HGIKPVVSTQ LLLNGSLAEE EV MIRSENI TNNAKNILVQ FNTPVQINCT RPNNNTRKSI RIGPGQAFYA TGDIIGDIRQ AHCNVSKATW NETLGKVVKQ LRK HFGNNT IIRFANSSGG DLEVTTHSFN CGGEFFYCNT SGLFNSTWIS NTSVQGSNST GSNDSITLPC RIKQIINMWQ RIGQ AMYAP PIQGVIRCVS NITGLILTRD GGSTNSTTET FRPGGGDMRD NWRSELYKYK VVKIEPLGVA PTRCKRRVVG RRRRR R |
-分子 #2: gp41 protein of HIV envelope trimer
| 分子 | 名称: gp41 protein of HIV envelope trimer / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 17.146482 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: AVGIGAVFLG FLGAAGSTMG AASMTLTVQA RNLLSGIVQQ QSNLLRAPEA QQHLLKLTVW GIKQLQARVL AVERYLRDQQ LLGIWGCSG KLICCTNVPW NSSWSNRNLS EIWDNMTWLQ WDKEISNYTQ IIYGLLEESQ NQQEKNEQDL LALD |
-分子 #3: PGT145 antibody fragment, heavy chain
| 分子 | 名称: PGT145 antibody fragment, heavy chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 28.793164 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QASTMDWIWR ILFLVAAATS AHSQVQLVQS GAEVKKPGSS VKVSCKASGN SFSNHDVHWV RQATGQGLEW MGWMSHEGDK TGLAQKFQG RVTITRDSGA STVYMELRGL TADDTAIYYC LTGSKHRLRD YFLYNEYGPN YEEWGDYLAT LDVWGHGTAV T VSSASTKG ...文字列: QASTMDWIWR ILFLVAAATS AHSQVQLVQS GAEVKKPGSS VKVSCKASGN SFSNHDVHWV RQATGQGLEW MGWMSHEGDK TGLAQKFQG RVTITRDSGA STVYMELRGL TADDTAIYYC LTGSKHRLRD YFLYNEYGPN YEEWGDYLAT LDVWGHGTAV T VSSASTKG PSVFPLAPSS KSTSGGTAAL GCLVKDYFPE PVTVSWNSGA LTSGVHTFPA VLQSSGLYSL SSVVTVPSSS LG TQTYICN VNHKPSNTKV DKKVEPKSCD |
-分子 #4: PGT145 antibody fragment, light chain
| 分子 | 名称: PGT145 antibody fragment, light chain / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.95375 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: EVVITQSPLF LPVTPGEAAS LSCKCSHSLQ HSTGANYLAW YLQRPGQTPR LLIHLATHRA SGVPDRFSGS GSGTDFTLKI SRVESDDVG TYYCMQGLHS PWTFGQGTKV EIKRTVAAPS VFIFPPSDEQ LKSGTASVVC LLNNFYPREA KVQWKVDNAL Q SGNSQESV ...文字列: EVVITQSPLF LPVTPGEAAS LSCKCSHSLQ HSTGANYLAW YLQRPGQTPR LLIHLATHRA SGVPDRFSGS GSGTDFTLKI SRVESDDVG TYYCMQGLHS PWTFGQGTKV EIKRTVAAPS VFIFPPSDEQ LKSGTASVVC LLNNFYPREA KVQWKVDNAL Q SGNSQESV TEQDSKDSTY SLSSTLTLSK ADYEKHKVYA CEVTHQGLSS PVTKSFNRGE C |
-分子 #11: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 11 / コピー数: 20 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 43.89 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.75 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー







 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)