+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cyro-EM structure of the Na+/H+ antipoter SOS1 from Arabidopsis thaliana,class2 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Sodium / Proton / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報sodium:proton antiporter activity / chloroplast envelope / regulation of reactive oxygen species metabolic process / sodium ion transport / response to salt stress / potassium ion transmembrane transport / response to reactive oxygen species / response to hydrogen peroxide / response to oxidative stress / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.67 Å | |||||||||
 データ登録者 データ登録者 | Yang GH / Zhang YM / Zhou JQ / Jia YT / Xu X / Fu P / Wu HY | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Plants / 年: 2023 ジャーナル: Nat Plants / 年: 2023タイトル: Structural basis for the activity regulation of Salt Overly Sensitive 1 in Arabidopsis salt tolerance. 著者: Yanming Zhang / Jiaqi Zhou / Xuping Ni / Qinrui Wang / Yutian Jia / Xia Xu / Haoyang Wu / Peng Fu / Han Wen / Yan Guo / Guanghui Yang /  要旨: The plasma membrane Na/H exchanger Salt Overly Sensitive 1 (SOS1) is crucial for plant salt tolerance. Unlike typical sodium/proton exchangers, SOS1 contains a large cytoplasmic domain (CPD) that ...The plasma membrane Na/H exchanger Salt Overly Sensitive 1 (SOS1) is crucial for plant salt tolerance. Unlike typical sodium/proton exchangers, SOS1 contains a large cytoplasmic domain (CPD) that regulates Na/H exchange activity. However, the underlying modulation mechanism remains unclear. Here we report the structures of SOS1 from Arabidopsis thaliana in two conformations, primarily differing in CPD flexibility. The CPD comprises an interfacial domain, a cyclic nucleotide-binding domain-like domain (CNBD-like domain) and an autoinhibition domain. Through yeast cell-based Na tolerance test, we reveal the regulatory role of the interfacial domain and the activation role of the CNBD-like domain. The CPD forms a negatively charged cavity that is connected to the ion binding site. The transport of Na may be coupled with the conformational change of CPD. These findings provide structural and functional insight into SOS1 activity regulation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_36076.map.gz emd_36076.map.gz | 59.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-36076-v30.xml emd-36076-v30.xml emd-36076.xml emd-36076.xml | 13.7 KB 13.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_36076.png emd_36076.png | 54 KB | ||
| Filedesc metadata |  emd-36076.cif.gz emd-36076.cif.gz | 5.6 KB | ||
| その他 |  emd_36076_half_map_1.map.gz emd_36076_half_map_1.map.gz emd_36076_half_map_2.map.gz emd_36076_half_map_2.map.gz | 58.9 MB 58.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-36076 http://ftp.pdbj.org/pub/emdb/structures/EMD-36076 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36076 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36076 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_36076_validation.pdf.gz emd_36076_validation.pdf.gz | 731 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_36076_full_validation.pdf.gz emd_36076_full_validation.pdf.gz | 730.6 KB | 表示 | |
| XML形式データ |  emd_36076_validation.xml.gz emd_36076_validation.xml.gz | 12.2 KB | 表示 | |
| CIF形式データ |  emd_36076_validation.cif.gz emd_36076_validation.cif.gz | 14.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36076 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36076 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36076 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36076 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8jdaMC  8jd9C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_36076.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_36076.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_36076_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
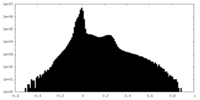
| 投影像・断面図 |
| ||||||||||||
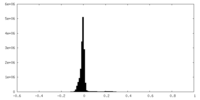
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_36076_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
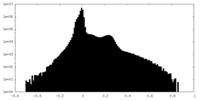
| 投影像・断面図 |
| ||||||||||||
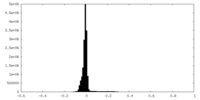
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Homodimer of AtSOS1
| 全体 | 名称: Homodimer of AtSOS1 |
|---|---|
| 要素 |
|
-超分子 #1: Homodimer of AtSOS1
| 超分子 | 名称: Homodimer of AtSOS1 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Na+/H+ antipoter SOS1 from Arabidopsis thaliana
| 分子 | 名称: Na+/H+ antipoter SOS1 from Arabidopsis thaliana / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 配列 | 文字列: MKLESSPVDA VLFVGMSLVL GIASRHLLRG TRVPYTVALL VIGIALGSLE YGAKHNLGKI GHGIRIWNEI DPELLLAVFL PALLFESSFS MEVHQIKRCL GQMVLLAVPG VLISTACLGS LVKVTFPYEW DWKTSLLLGG LLSATDPVAV VALLKELGAS KKLSTIIEGE ...文字列: MKLESSPVDA VLFVGMSLVL GIASRHLLRG TRVPYTVALL VIGIALGSLE YGAKHNLGKI GHGIRIWNEI DPELLLAVFL PALLFESSFS MEVHQIKRCL GQMVLLAVPG VLISTACLGS LVKVTFPYEW DWKTSLLLGG LLSATDPVAV VALLKELGAS KKLSTIIEGE SLMNDGTAIV VFQLFLKMAM GQNSDWSSII KFLLKVALGA VGIGLAFGIA SVIWLKFIFN DTVIEITLTI AVSYFAYYTA QEWAGASGVL TVMTLGMFYA AFARTAFKGD SQKSLHHFWE MVAYIANTLI FILSGVVIAE GILDSDKIAY QGNSWRFLFL LYVYIQLSRV VVVGVLYPLL CRFGYGLDWK ESIILVWSGL RGAVALALSL SVKQSSGNSH ISKETGTLFL FFTGGIVFLT LIVNGSTTQF VLRLLRMDIL PAPKKRILEY TKYEMLNKAL RAFQDLGDDE ELGPADWPTV ESYISSLKGS EGELVHHPHN GSKIGSLDPK SLKDIRMRFL NGVQATYWEM LDEGRISEVT ANILMQSVDE ALDQVSTTLC DWRGLKPHVN FPNYYNFLHS KVVPRKLVTY FAVERLESAC YISAAFLRAH TIARQQLYDF LGESNIGSIV INESEKEGEE AKKFLEKVRS SFPQVLRVVK TKQVTYSVLN HLLGYIENLE KVGLLEEKEI AHLHDAVQTG LKKLLRNPPI VKLPKLSDMI TSHPLSVALP PAFCEPLKHS KKEPMKLRGV TLYKEGSKPT GVWLIFDGIV KWKSKILSNN HSLHPTFSHG STLGLYEVLT GKPYLCDLIT DSMVLCFFID SEKILSLQSD STIDDFLWQE SALVLLKLLR PQIFESVAMQ ELRALVSTES SKLTTYVTGE SIEIDCNSIG LLLEGFVKPV GIKEELISSP AALSPSNGNQ SFHNSSEASG IMRVSFSQQA TQYIVETRAR AIIFNIGAFG ADRTLHRRPS SLTPPRSSSS DQLQRSFRKE HRGLMSWPEN IYAKQQQEIN KTTLSLSERA MQLSIFGSMV NVYRRSVSFG GIYNNKLQDN LLYKKLPLNP AQGLVSAKSE SSIVTKKQLE TRKHACQLPL KGESSTRQNT MVESSDEEDE DEGIVVRIDS PSKIVFRNDL LESAWSHPQF EKGGGSGGGS GGSAWSHPQF EKGSDYKDDD DK |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: OTHER / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.67 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 427505 |
| 初期 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)