+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the Clr6S (Clr6-HDAC) complex from S. pombe | |||||||||
 マップデータ マップデータ | Cryo-EM map of Clr6-HDAC complex | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Rpd3S / Histone deacetylase / HDAC / Clr6 / Rpd3 / Clr6S / Clr6 HDAC / TRANSCRIPTION | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報histone H3K9 deacetylase activity / histone H3K14 deacetylase activity / histone H4K16 deacetylase activity / : / HDACs deacetylate histones / H3-H4 histone complex chaperone activity / SUMOylation of chromatin organization proteins / Rpd3S complex / Rpd3L complex / Rpd3L-Expanded complex ...histone H3K9 deacetylase activity / histone H3K14 deacetylase activity / histone H4K16 deacetylase activity / : / HDACs deacetylate histones / H3-H4 histone complex chaperone activity / SUMOylation of chromatin organization proteins / Rpd3S complex / Rpd3L complex / Rpd3L-Expanded complex / NuRD complex / histone deacetylase / protein lysine deacetylase activity / DNA repair-dependent chromatin remodeling / histone deacetylase activity / Sin3-type complex / NuA4 histone acetyltransferase complex / epigenetic regulation of gene expression / nucleosome binding / transcription initiation-coupled chromatin remodeling / mitotic spindle / transcription corepressor activity / histone binding / chromatin remodeling / DNA repair / DNA damage response / regulation of DNA-templated transcription / chromatin / regulation of transcription by RNA polymerase II / negative regulation of transcription by RNA polymerase II / nucleus / metal ion binding / cytoplasm / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
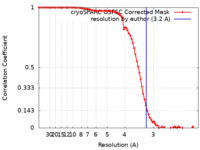
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.2 Å | |||||||||
 データ登録者 データ登録者 | Zhang HQ / Wang X / Wang YN / Liu SM / Zhang Y / Xu K / Ji LT / Kornberg RD | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2023 ジャーナル: Proc Natl Acad Sci U S A / 年: 2023タイトル: Class I histone deacetylase complex: Structure and functional correlates. 著者: Xiao Wang / Yannan Wang / Simiao Liu / Yi Zhang / Ke Xu / Liting Ji / Roger D Kornberg / Heqiao Zhang /   要旨: The Clr6S complex, a class I histone deacetylase complex, functions as a zinc-dependent enzyme to remove acetyl groups from lysine residues in histone tails. We report here the cryo-EM structure of ...The Clr6S complex, a class I histone deacetylase complex, functions as a zinc-dependent enzyme to remove acetyl groups from lysine residues in histone tails. We report here the cryo-EM structure of Clr6S alone and a cryo-EM map of Clr6S in complex with a nucleosome. The active center, revealed at near-atomic resolution, includes features important for catalysis-A water molecule coordinated by zinc, the likely nucleophile for attack on the acetyl-lysine bond, and a loop that may position the substrate for catalysis. The cryo-EM map in the presence of a nucleosome reveals multiple Clr6S-nucleosome contacts and a high degree of relative motion of Clr6S and the nucleosome. Such flexibility may be attributed to interaction at a site in the flexible histone tail and is likely important for the function of the deacetylase, which acts at multiple sites in other histone tails. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_35416.map.gz emd_35416.map.gz | 56.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-35416-v30.xml emd-35416-v30.xml emd-35416.xml emd-35416.xml | 23.2 KB 23.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_35416_fsc.xml emd_35416_fsc.xml | 9.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_35416.png emd_35416.png | 29.3 KB | ||
| Filedesc metadata |  emd-35416.cif.gz emd-35416.cif.gz | 7.9 KB | ||
| その他 |  emd_35416_half_map_1.map.gz emd_35416_half_map_1.map.gz emd_35416_half_map_2.map.gz emd_35416_half_map_2.map.gz | 55.4 MB 55.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-35416 http://ftp.pdbj.org/pub/emdb/structures/EMD-35416 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35416 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35416 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_35416_validation.pdf.gz emd_35416_validation.pdf.gz | 1018.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_35416_full_validation.pdf.gz emd_35416_full_validation.pdf.gz | 1018.2 KB | 表示 | |
| XML形式データ |  emd_35416_validation.xml.gz emd_35416_validation.xml.gz | 16 KB | 表示 | |
| CIF形式データ |  emd_35416_validation.cif.gz emd_35416_validation.cif.gz | 20.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35416 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35416 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35416 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35416 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8ifgMC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_35416.map.gz / 形式: CCP4 / 大きさ: 59.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_35416.map.gz / 形式: CCP4 / 大きさ: 59.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM map of Clr6-HDAC complex | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: half map
| ファイル | emd_35416_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map
| ファイル | emd_35416_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cryo-EM structure of the Clr6-HDAC (Clr6S) complex from S. pombe
| 全体 | 名称: Cryo-EM structure of the Clr6-HDAC (Clr6S) complex from S. pombe |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-EM structure of the Clr6-HDAC (Clr6S) complex from S. pombe
| 超分子 | 名称: Cryo-EM structure of the Clr6-HDAC (Clr6S) complex from S. pombe タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#6 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 600 KDa |
-分子 #1: RbAp48-related WD40 repeat-containing protein prw1
| 分子 | 名称: RbAp48-related WD40 repeat-containing protein prw1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 48.528926 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MAVSAVPHPS KQAQASEEGI NQEKCINEEY KIWKKNSPFL YDLIITRALE WPCMSLQWYP EQQIFAEHGY TEQKMFLGVR ADVGKYLLA VASIQLPYLN QTVPPTTMEG ASAGDESSLR VNISNLYSHP ESVCSAKLMP QDDSCVATVG NYHNDVLVFD K ESFESYSS ...文字列: MAVSAVPHPS KQAQASEEGI NQEKCINEEY KIWKKNSPFL YDLIITRALE WPCMSLQWYP EQQIFAEHGY TEQKMFLGVR ADVGKYLLA VASIQLPYLN QTVPPTTMEG ASAGDESSLR VNISNLYSHP ESVCSAKLMP QDDSCVATVG NYHNDVLVFD K ESFESYSS ASESPLKPKY RLTKHTQPCT SVCWNFLSKG TLVSGSQDAT LSCWDLNAYN ESDSASVLKV HISSHEKQVS DV RFHYKHQ DLLASVSYDQ YLHVHDIRRP DASTKPARSV HAHSGPIHSV AFNPHNDFIL ATCSTDKTIA LWDLRNLNQR LHT LEGHED IVTKISFSPH EEPILASTSA DRRTLVWDLS RIGEDQPAEE AQDGPPELLF MHGGHTSCTI DMDWCPNYNW TMAT AAEDN ILQIWTPSRS IWGNEQLEED ATAYLS UniProtKB: RbAp48-related WD40 repeat-containing protein prw1 |
-分子 #2: Paired amphipathic helix protein pst2
| 分子 | 名称: Paired amphipathic helix protein pst2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 125.011609 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MEQTLAILKN DNSTLVAEMQ NQLVHDFSPN GTALPELDIK AFVQKLGQRL CHRPYVYSAF MDVVKALHNE IVDFPGFIER ISVILRDYP DLLEYLNIFL PSSYKYLLSN SGANFTLQFT TPSGPVSTPS TYVATYNDLP CTYHRAIGFV SRVRRALLSN P EQFFKLQD ...文字列: MEQTLAILKN DNSTLVAEMQ NQLVHDFSPN GTALPELDIK AFVQKLGQRL CHRPYVYSAF MDVVKALHNE IVDFPGFIER ISVILRDYP DLLEYLNIFL PSSYKYLLSN SGANFTLQFT TPSGPVSTPS TYVATYNDLP CTYHRAIGFV SRVRRALLSN P EQFFKLQD SLRKFKNSEC SLSELQTIVT SLLAEHPSLA HEFHNFLPSS IFFGSKPPLG SFPLRGIQSS QFTLSNISDL LS QSRPDNL SPFSHLSNES SDFFKNVKNV LTDVETYHEF LKLLNLYVQG IIDRNILVSR GFGFLKSNSG LWRSFLSLTS LSP EEFLSV YNSACSDFPE CGPSYRLLPV EERNISCSGR DDFAWGILND DWVSHPTWAS EESGFIVQRK TPYEEAMTKL EEER YEFDR HIEATSWTIK SLKKIQNRIN ELPEEERETY TLEEGLGLPS KSIYKKTIKL VYTSEHAEEM FKALERMPCL TLPLV ISRL EEKNEEWKSV KRSLQPGWRS IEFKNYDKSL DSQCVYFKAR DKKNVSSKFL LAEADILRSQ AKLHFPLRSR SAFEFS FVY DNEIVLFDTC YMVCTYIVCN SPSGLKKVEH FFKNILPLHF GLEKDKFSIF LDQVFRGPDY DVNAPNIVGN KPVRRKR SN SITQLTEFVK QPKINGQRES RSAAAARKKE ESGNKSQSNS QNSLSDESGN VTPVSKKQLS QPAAAIKASL KYPSHPDS L LEHQDHAGDT ENEMHDDVDK EQFGYSSMYV FFRLFNLLYE RLYELQRLED QVSIIQQRII PNPVSQKQKI WRDRWNDLS DVPDEKTHYE NTYVMILRLI YGIVDQSAFE DYLRFYYGNK AYKIYTIDKL VWSAAKQVHH IVSDGKYKFV TSLVEQNSSA SPKKNYDDF LYRLEIEKLL NPDEILFRFC WINKFKSFGI KIMKRANLIV DQSLDTQRRV WKKYVQNYRI QKLTEEISYK N YRCPFLCR NIEKERTVEQ LVSRLQTKLL RSAELVSGLQ AKLCLDSFKL LYLPRTEDSY IDASYLRLRD TDFLDCQNKR KQ RWRNRWE SLLKSVRGTS DNTAEVNFDA DINALFIP UniProtKB: Paired amphipathic helix protein pst2 |
-分子 #3: Histone deacetylase clr6
| 分子 | 名称: Histone deacetylase clr6 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO / EC番号: histone deacetylase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 45.773371 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: KKKVSYFYDE DVGNYHYGPQ HPMKPHRVRM VHNLVVNYNL YEKLNVITPV RATRNDMTRC HTDEYIEFLW RVTPDTMEKF QPHQLKFNV GDDCPVFDGL YEFCSISAGG SIGAAQELNS GNAEIAINWA GGLHHAKKRE ASGFCYVNDI ALAALELLKY H QRVLYIDI ...文字列: KKKVSYFYDE DVGNYHYGPQ HPMKPHRVRM VHNLVVNYNL YEKLNVITPV RATRNDMTRC HTDEYIEFLW RVTPDTMEKF QPHQLKFNV GDDCPVFDGL YEFCSISAGG SIGAAQELNS GNAEIAINWA GGLHHAKKRE ASGFCYVNDI ALAALELLKY H QRVLYIDI DVHHGDGVEE FFYTTDRVMT CSFHKFGEYF PGTGHIKDTG IGTGKNYAVN VPLRDGIDDE SYESVFKPVI SH IMQWFRP EAVILQCGTD SLAGDRLGCF NLSMKGHSMC VDFVKSFNLP MICVGGGGYT VRNVARVWTY ETGLLAGEEL DEN LPYNDY LQYYGPDYKL NVLSNNMENH NTRQYLDSIT SEIIENLRNL SFAPSVQMHK TPGDFTFENA EKQNIAKEEI MDER V UniProtKB: Histone deacetylase clr6 |
-分子 #4: Chromatin modification-related protein eaf3
| 分子 | 名称: Chromatin modification-related protein eaf3 / タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 39.191199 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MAVSYKVNER VLCFHGPLLY EAKIVDTEMK GDVTTYLIHY KGWKNSWDEW VEQDRILQWT EENLKTQKEL KNAAISTRQK PTSKKSASS TSKHDSTGVK TSGKRSRESS TVTVDGDSHE LPSRIKTQKS ESPIPQQVKR DGTTDAKNEE TTKPENNEKD D FEEEPPLP ...文字列: MAVSYKVNER VLCFHGPLLY EAKIVDTEMK GDVTTYLIHY KGWKNSWDEW VEQDRILQWT EENLKTQKEL KNAAISTRQK PTSKKSASS TSKHDSTGVK TSGKRSRESS TVTVDGDSHE LPSRIKTQKS ESPIPQQVKR DGTTDAKNEE TTKPENNEKD D FEEEPPLP KHKISVPDVL KLWLVDDWEN ITKNQQLIAI PRNPTVRAAI AAFRESKISH LNNEIDVDVF EQAMAGLVIY FN KCLGNML LYRFERQQYL EIRQQYPDTE MCDLYGVEHL IRLFVSLPEL IDRTNMDSQS IECLLNYIEE FLKYLVLHKD EYF IKEYQN APPNYRSLVG V UniProtKB: Chromatin modification-related protein eaf3 |
-分子 #5: Cph1
| 分子 | 名称: Cph1 / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 45.046082 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MASSINNSSQ PTVPSISNNS HGDSFVNEGP PSNFKNNSLT SSTHSSTDHV NVLPISQDKE MDISSPVKKQ KASYSNKSPN KAPIQKSRG SSLKSHLETE SQQTPVKRRR RKATIRNVDY CSACGGRGLF ICCEGCPCSF HLSCLEPPLT PENIPEGSWF C VTCSIKSH ...文字列: MASSINNSSQ PTVPSISNNS HGDSFVNEGP PSNFKNNSLT SSTHSSTDHV NVLPISQDKE MDISSPVKKQ KASYSNKSPN KAPIQKSRG SSLKSHLETE SQQTPVKRRR RKATIRNVDY CSACGGRGLF ICCEGCPCSF HLSCLEPPLT PENIPEGSWF C VTCSIKSH HPPKHPLSIW SQLYDWIDSQ NPSQYRLPDD LVHYFHGISR GDTGAYKETE GEMDTDEFSA LPTGSSITNL AY CGYCSKP SMGACWVYGC QLCDTFYHKN CKEHAKKCSH DSIGKKGMRV PKNAVVIRTP LVLDTTSNTL NPKVMISGWQ FLM GEFPSD ELLYFPRLPV SCLYKVSEDG LIKDFLYAIG IEAKKFNNER KKRELEVIPP DVKSALLPAR THPNLPIALR TLFN KART UniProtKB: Uncharacterized protein C16C9.05 |
-分子 #6: Cph2
| 分子 | 名称: Cph2 / タイプ: protein_or_peptide / ID: 6 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 68.871703 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MDAKPWNHTS EAFQASILED LKIIQKAGAE RNAKSSHGSI NSRSASPNKA TSRRNRAQNG NSNGRASVDN SDDGSKDDLD YSPSVKRKH VNGEGAEKGD HDTSNNGPSI TKLRRKVRRT YDTKDGFVAW NTLDDDFRPI VPDQERSRKI NPQKGNNNNL L KENKSLKT ...文字列: MDAKPWNHTS EAFQASILED LKIIQKAGAE RNAKSSHGSI NSRSASPNKA TSRRNRAQNG NSNGRASVDN SDDGSKDDLD YSPSVKRKH VNGEGAEKGD HDTSNNGPSI TKLRRKVRRT YDTKDGFVAW NTLDDDFRPI VPDQERSRKI NPQKGNNNNL L KENKSLKT TAKDLSDISS SSMKKANNSS KPLFSGKLTF KANIPVPTSE VVTENNVTRN VTVYSNQKHL GNESENFNDM EG RAEDISS NELLPTPEEY PYRYNNDYCS ACHGPGNFLC CETCPNSFHF TCIDPPIEEK NLPDDAWYCN ECKHHSLYNE LDE QEELES NVKEEGTMVD VWMQLCTYID SHNPIQFHLP HSISSFFRGV GSGVMGEYIE TDVLKHLKSS RRSNGEERDP LLLK SKSGT PILCFRCHKS ALVSQSILAC DYCNSYWHPD CLNPPLATLP SNLRKWKCPN HSDHVTPRYR LPEKAKVIRV GLPRG FKNK GNIVIDENED EPSVQTIQLQ GKIRVVPSKP FKLNFLEQIR DNVINLRKMV EQDEQLCIET FSKFDFYATR DCELPL RIL CDVANDNLEN DDYVLALRDL LRISKWDPNQ PVPAPFDLAN LLSY UniProtKB: Uncharacterized protein C2F7.07c |
-分子 #7: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 7 / コピー数: 3 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-分子 #8: water
| 分子 | 名称: water / タイプ: ligand / ID: 8 / コピー数: 1 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






 Z
Z Y
Y X
X