+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Clostridium thermocellum RNA polymerase transcription open complex with SigI1 and its promoter | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | TRANSCRIPTION OPEN COMPLEX / SIGI / TRANSCRIPTION / TRANSCRIPTION-DNA complex | |||||||||
| 生物種 |  Acetivibrio thermocellus DSM 1313 (バクテリア) / Acetivibrio thermocellus DSM 1313 (バクテリア) /  Acetivibrio thermocellus DSM1313 (バクテリア) / Acetivibrio thermocellus DSM1313 (バクテリア) /  Acetivibrio thermocellus (バクテリア) Acetivibrio thermocellus (バクテリア) | |||||||||
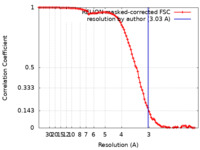
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / ネガティブ染色法 / 解像度: 3.03 Å | |||||||||
 データ登録者 データ登録者 | Li J / Zhang H / Li D / Feng Y / Zhu P | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Structure of the transcription open complex of distinct σ factors. 著者: Jie Li / Haonan Zhang / Dongyu Li / Ya-Jun Liu / Edward A Bayer / Qiu Cui / Yingang Feng / Ping Zhu /   要旨: Bacterial σ factors of the σ-family are widespread in Bacilli and Clostridia and are involved in the heat shock response, iron metabolism, virulence, and carbohydrate sensing. A multiplicity of σ ...Bacterial σ factors of the σ-family are widespread in Bacilli and Clostridia and are involved in the heat shock response, iron metabolism, virulence, and carbohydrate sensing. A multiplicity of σ paralogues in some cellulolytic bacteria have been shown to be responsible for the regulation of the cellulosome, a multienzyme complex that mediates efficient cellulose degradation. Here, we report two structures at 3.0 Å and 3.3 Å of two transcription open complexes formed by two σ factors, SigI1 and SigI6, respectively, from the thermophilic, cellulolytic bacterium, Clostridium thermocellum. These structures reveal a unique, hitherto-unknown recognition mode of bacterial transcriptional promoters, both with respect to domain organization and binding to promoter DNA. The key characteristics that determine the specificities of the σ paralogues were further revealed by comparison of the two structures. Consequently, the σ factors represent a distinct set of the σ-family σ factors, thus highlighting the diversity of bacterial transcription. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_35130.map.gz emd_35130.map.gz | 71.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-35130-v30.xml emd-35130-v30.xml emd-35130.xml emd-35130.xml | 24.5 KB 24.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_35130_fsc.xml emd_35130_fsc.xml | 9.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_35130.png emd_35130.png | 109.4 KB | ||
| マスクデータ |  emd_35130_msk_1.map emd_35130_msk_1.map | 83.7 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-35130.cif.gz emd-35130.cif.gz | 8.4 KB | ||
| その他 |  emd_35130_half_map_1.map.gz emd_35130_half_map_1.map.gz emd_35130_half_map_2.map.gz emd_35130_half_map_2.map.gz | 65.3 MB 65.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-35130 http://ftp.pdbj.org/pub/emdb/structures/EMD-35130 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35130 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35130 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_35130_validation.pdf.gz emd_35130_validation.pdf.gz | 833.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_35130_full_validation.pdf.gz emd_35130_full_validation.pdf.gz | 833.2 KB | 表示 | |
| XML形式データ |  emd_35130_validation.xml.gz emd_35130_validation.xml.gz | 17 KB | 表示 | |
| CIF形式データ |  emd_35130_validation.cif.gz emd_35130_validation.cif.gz | 22.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35130 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35130 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35130 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35130 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_35130.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_35130.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.04 Å | ||||||||||||||||||||||||||||||||||||
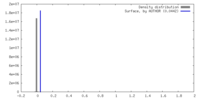
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_35130_msk_1.map emd_35130_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
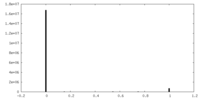
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_35130_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
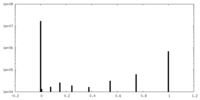
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_35130_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
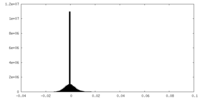
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Clostridium thermocellum RNA polymerase transcription open comple...
+超分子 #1: Clostridium thermocellum RNA polymerase transcription open comple...
+分子 #1: DNA-directed RNA polymerase subunit alpha
+分子 #2: DNA-directed RNA polymerase subunit beta
+分子 #3: DNA-directed RNA polymerase subunit beta'
+分子 #4: DNA-directed RNA polymerase subunit omega
+分子 #5: RNA polymerase sigma factor SigI1
+分子 #6: DNA (80-mer)
+分子 #7: DNA (80-mer)
+分子 #8: ZINC ION
+分子 #9: MAGNESIUM ION
-実験情報
-構造解析
| 手法 | ネガティブ染色法, クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 23.54 mg/mL |
|---|---|
| 緩衝液 | pH: 8 詳細: 20 mM Tris-HCl pH 8.0, 150 mM NaCl, 2 mM DTT, 0.2 mM EDTA, 5% (v/v) glycerol, 10 mM MgCl2 |
| 染色 | タイプ: NEGATIVE / 材質: Uranyl Acetate |
| 凍結 | 凍結剤: ETHANE |
| 詳細 | This sample was monodisperse. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 77.0 K / 最高: 77.0 K |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - 画像ごとのフレーム数: 1-32 / 平均露光時間: 0.25 sec. / 平均電子線量: 1.875 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 最大 デフォーカス(補正後): 2.5 µm / 最小 デフォーカス(補正後): 1.5 µm / 倍率(補正後): 22500 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 22500 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)