+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the Spring Viraemia of Carp Virus ribonucleoprotein Complex | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | Ribonucleoprotein / Complex / VIRUS / VIRUS LIKE PARTICLE-RNA complex | ||||||||||||
| 生物種 |  Sprivirus cyprinus (ウイルス) / Sprivirus cyprinus (ウイルス) /  Trichoplusia ni (イラクサキンウワバ) / Trichoplusia ni (イラクサキンウワバ) /  Sprivivirus cyprinus (ウイルス) Sprivivirus cyprinus (ウイルス) | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.7 Å | ||||||||||||
 データ登録者 データ登録者 | Liu B / Wang ZX / Yang T / Yu DQ / Ouyang Q | ||||||||||||
| 資金援助 |  中国, 3件 中国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: J Virol / 年: 2023 ジャーナル: J Virol / 年: 2023タイトル: Structure of the Spring Viraemia of Carp Virus Ribonucleoprotein Complex Reveals Its Assembly Mechanism and Application in Antiviral Drug Screening. 著者: Zhao-Xi Wang / Bing Liu / Tian Yang / Daqi Yu / Chu Zhang / Liming Zheng / Jin Xie / Bin Liu / Mengxi Liu / Hailin Peng / Luhua Lai / Qi Ouyang / Songying Ouyang / Yong-An Zhang /  要旨: Spring viremia of carp virus (SVCV) is a highly pathogenic infecting the common carp, yet neither a vaccine nor effective therapies are available to treat spring viremia of carp (SVC). Like all ...Spring viremia of carp virus (SVCV) is a highly pathogenic infecting the common carp, yet neither a vaccine nor effective therapies are available to treat spring viremia of carp (SVC). Like all negative-sense viruses, SVCV contains an RNA genome that is encapsidated by the nucleoprotein (N) in the form of a ribonucleoprotein (RNP) complex, which serves as the template for viral replication and transcription. Here, the three-dimensional (3D) structure of SVCV RNP was resolved through cryo-electron microscopy (cryo-EM) at a resolution of 3.7 Å. RNP assembly was stabilized by N and C loops; RNA was wrapped in the groove between the N and C lobes with 9 nt nucleotide per protomer. Combined with mutational analysis, our results elucidated the mechanism of RNP formation. The RNA binding groove of SVCV N was used as a target for drug virtual screening, and it was found suramin had a good antiviral effect. This study provided insights into RNP assembly, and anti-SVCV drug screening was performed on the basis of this structure, providing a theoretical basis and efficient drug screening method for the prevention and treatment of SVC. Aquaculture accounts for about 70% of global aquatic products, and viral diseases severely harm the development of aquaculture industry. Spring viremia of carp virus (SVCV) is the pathogen causing highly contagious spring viremia of carp (SVC) disease in cyprinids, especially common carp (), yet neither a vaccine nor effective therapies are available to treat this disease. In this study, we have elucidated the mechanism of SVCV ribonucleoprotein complex (RNP) formation by resolving the 3D structure of SVCV RNP and screened antiviral drugs based on the structure. It is found that suramin could competitively bind to the RNA binding groove and has good antiviral effects both and . Our study provides a template for rational drug discovery efforts to treat and prevent SVCV infections. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_33810.map.gz emd_33810.map.gz | 3.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-33810-v30.xml emd-33810-v30.xml emd-33810.xml emd-33810.xml | 21 KB 21 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_33810.png emd_33810.png | 89.7 KB | ||
| マスクデータ |  emd_33810_msk_1.map emd_33810_msk_1.map | 18.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-33810.cif.gz emd-33810.cif.gz | 6.8 KB | ||
| その他 |  emd_33810_half_map_1.map.gz emd_33810_half_map_1.map.gz emd_33810_half_map_2.map.gz emd_33810_half_map_2.map.gz | 13.7 MB 13.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-33810 http://ftp.pdbj.org/pub/emdb/structures/EMD-33810 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33810 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33810 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_33810_validation.pdf.gz emd_33810_validation.pdf.gz | 781 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_33810_full_validation.pdf.gz emd_33810_full_validation.pdf.gz | 780.6 KB | 表示 | |
| XML形式データ |  emd_33810_validation.xml.gz emd_33810_validation.xml.gz | 9.8 KB | 表示 | |
| CIF形式データ |  emd_33810_validation.cif.gz emd_33810_validation.cif.gz | 11.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33810 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33810 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33810 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33810 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_33810.map.gz / 形式: CCP4 / 大きさ: 18.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_33810.map.gz / 形式: CCP4 / 大きさ: 18.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.37 Å | ||||||||||||||||||||||||||||||||||||
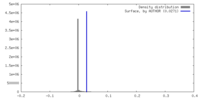
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_33810_msk_1.map emd_33810_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
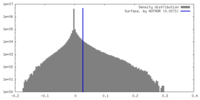
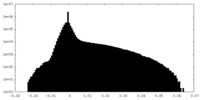
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_33810_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
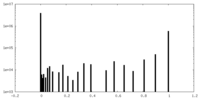
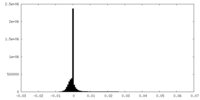
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_33810_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
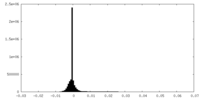
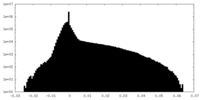
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : ribonucleoprotein complex of the spring viraemia carp virus
| 全体 | 名称: ribonucleoprotein complex of the spring viraemia carp virus |
|---|---|
| 要素 |
|
-超分子 #1: ribonucleoprotein complex of the spring viraemia carp virus
| 超分子 | 名称: ribonucleoprotein complex of the spring viraemia carp virus タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Sprivirus cyprinus (ウイルス) Sprivirus cyprinus (ウイルス) |
-超分子 #2: nucleoprotein
| 超分子 | 名称: nucleoprotein / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
-超分子 #3: RNA
| 超分子 | 名称: RNA / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|
-分子 #1: Nucleoprotein
| 分子 | 名称: Nucleoprotein / タイプ: protein_or_peptide / ID: 1 / コピー数: 11 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Sprivivirus cyprinus (ウイルス) Sprivivirus cyprinus (ウイルス) |
| 分子量 | 理論値: 46.62116 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: RIKTNAAVAA VLPANEDQAD YPSTFFEGGN EIRLYVNRGE KLDVLRQYVY MGLVEKNCRI QHVNAYLYAV LKGERELLEA DWDSFGHKI GIQGDKIGPF NLVRVEDIPD GLPDGKLNAE VSAEDDAWLP LFLLGLYRVG RASETAYRTL LMESLIKQCK A IKSDWVSP ...文字列: RIKTNAAVAA VLPANEDQAD YPSTFFEGGN EIRLYVNRGE KLDVLRQYVY MGLVEKNCRI QHVNAYLYAV LKGERELLEA DWDSFGHKI GIQGDKIGPF NLVRVEDIPD GLPDGKLNAE VSAEDDAWLP LFLLGLYRVG RASETAYRTL LMESLIKQCK A IKSDWVSP VTATHKYFDV WGNDGNYLKI VACVDMFYNH FKKSIKATFR WGTIVSRFKD CAALATLGHV VKITGLTIEE VF TWVLQTE VADELVKMMK PGQEIDKSTS YMPYLIDMGI SAKSPYSTIK NPSFHFWGQL VAALCRSKRA LNARQPDEID SMS ISNASL LMAYALGSSP DIEQQFSTGN TYRKPPKEAS YLVSEEPKNR SVVEWIAWYS DVDNKPTDDM LMMAKRVAGT ISGP RDNSV GKWIKQTYG |
-分子 #2: RNA (99-mer)
| 分子 | 名称: RNA (99-mer) / タイプ: rna / ID: 2 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 分子量 | 理論値: 30.26548 KDa |
| 配列 | 文字列: UUUUUUUUUU UUUUUUUUUU UUUUUUUUUU UUUUUUUUUU UUUUUUUUUU UUUUUUUUUU UUUUUUUUUU UUUUUUUUUU UUUUUUUUU UUUUUUUUUU |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: C-flat-1.2/1.3 / 材質: GOLD / メッシュ: 400 / 支持フィルム - 材質: GRAPHENE / 支持フィルム - トポロジー: CONTINUOUS |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 300 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 45.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm 最小 デフォーカス(公称値): 0.7000000000000001 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-7yg7: |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)