+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the Mycobacterium smegmatis DNA integrity scanning protein (MsDisA). | |||||||||
 マップデータ マップデータ | Combined map and sharpened with Bfactor of -50 in Relion | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | second messengers / stress response / c-di-AMP / cryo-EM / CELL CYCLE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報diadenylate cyclase / diadenylate cyclase activity / DNA repair / DNA binding / ATP binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア) | |||||||||
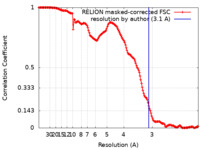
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Gautam S / Vinothkumar KR / Chatterji D | |||||||||
| 資金援助 |  インド, 2件 インド, 2件
| |||||||||
 引用 引用 |  ジャーナル: Protein Sci / 年: 2023 ジャーナル: Protein Sci / 年: 2023タイトル: Regulatory mechanisms of c-di-AMP synthase from Mycobacterium smegmatis revealed by a structure: Function analysis. 著者: Sudhanshu Gautam / Avisek Mahapa / Lahari Yeramala / Apoorv Gandhi / Sushma Krishnan / Vinothkumar Kutti R / Dipankar Chatterji /  要旨: Cyclic-di-nucleotide-based secondary messengers regulate various physiological functions, including stress responses in bacteria. Cyclic diadenosine monophosphate (c-di-AMP) has recently emerged as a ...Cyclic-di-nucleotide-based secondary messengers regulate various physiological functions, including stress responses in bacteria. Cyclic diadenosine monophosphate (c-di-AMP) has recently emerged as a crucial second messenger with implications in processes including osmoregulation, antibiotic resistance, biofilm formation, virulence, DNA repair, ion homeostasis, and sporulation, and has potential therapeutic applications. The contrasting activities of the enzymes diadenylate cyclase (DAC) and phosphodiesterase (PDE) determine the equilibrium levels of c-di-AMP. Although c-di-AMP is suspected of playing an essential role in the pathophysiology of bacterial infections and in regulating host-pathogen interactions, the mechanisms of its regulation remain relatively unexplored in mycobacteria. In this report, we biochemically and structurally characterize the c-di-AMP synthase (MsDisA) from Mycobacterium smegmatis. The enzyme activity is regulated by pH and substrate concentration; conditions of significance in the homoeostasis of c-di-AMP levels. Substrate binding stimulates conformational changes in the protein, and pApA and ppApA are synthetic intermediates detectable when enzyme efficiency is low. Unlike the orthologous Bacillus subtilis enzyme, MsDisA does not bind to, and its activity is not influenced in the presence of DNA. Furthermore, we have determined the cryo-EM structure of MsDisA, revealing asymmetry in its structure in contrast to the symmetric crystal structure of Thermotoga maritima DisA. We also demonstrate that the N-terminal minimal region alone is sufficient and essential for oligomerization and catalytic activity. Our data shed light on the regulation of mycobacterial DisA and possible future directions to pursue. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
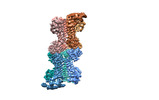
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_33540.map.gz emd_33540.map.gz | 301.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-33540-v30.xml emd-33540-v30.xml emd-33540.xml emd-33540.xml | 18.7 KB 18.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_33540_fsc.xml emd_33540_fsc.xml | 15.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_33540.png emd_33540.png | 39.7 KB | ||
| Filedesc metadata |  emd-33540.cif.gz emd-33540.cif.gz | 6.3 KB | ||
| その他 |  emd_33540_half_map_1.map.gz emd_33540_half_map_1.map.gz emd_33540_half_map_2.map.gz emd_33540_half_map_2.map.gz | 301.8 MB 301.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-33540 http://ftp.pdbj.org/pub/emdb/structures/EMD-33540 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33540 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33540 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_33540_validation.pdf.gz emd_33540_validation.pdf.gz | 736.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_33540_full_validation.pdf.gz emd_33540_full_validation.pdf.gz | 736.2 KB | 表示 | |
| XML形式データ |  emd_33540_validation.xml.gz emd_33540_validation.xml.gz | 23.4 KB | 表示 | |
| CIF形式データ |  emd_33540_validation.cif.gz emd_33540_validation.cif.gz | 31 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33540 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33540 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33540 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33540 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7y0dMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_33540.map.gz / 形式: CCP4 / 大きさ: 325 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_33540.map.gz / 形式: CCP4 / 大きさ: 325 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Combined map and sharpened with Bfactor of -50 in Relion | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||||||||||||||||||
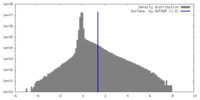
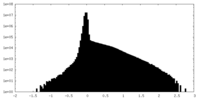
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: One of the half maps from Cryosparc
| ファイル | emd_33540_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | One of the half maps from Cryosparc | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
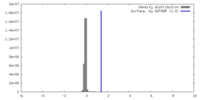
| 密度ヒストグラム |
-ハーフマップ: One of the half maps from Cryosparc
| ファイル | emd_33540_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | One of the half maps from Cryosparc | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
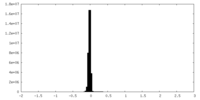
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cryo-EM structure of the Mycobacterium smegmatis DNA integrity sc...
| 全体 | 名称: Cryo-EM structure of the Mycobacterium smegmatis DNA integrity scanning protein (MsDisA). |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-EM structure of the Mycobacterium smegmatis DNA integrity sc...
| 超分子 | 名称: Cryo-EM structure of the Mycobacterium smegmatis DNA integrity scanning protein (MsDisA). タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア) |
| 分子量 | 理論値: 330 KDa |
-分子 #1: DNA integrity scanning protein DisA
| 分子 | 名称: DNA integrity scanning protein DisA / タイプ: protein_or_peptide / ID: 1 / コピー数: 8 / 光学異性体: LEVO / EC番号: diadenylate cyclase |
|---|---|
| 由来(天然) | 生物種:  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア) |
| 分子量 | 理論値: 41.844199 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAVKSGARSG RNVVHLARPT LRETLGRLAP GTPLRDGLER ILRGRTGALI VLGYDDSVEA ICDGGFVLDV RYAPTRLREL SKMDGAVVL SSDGSRILRA NVQLVPDPSI PTDESGTRHR SAERTAIQTG YPVISVSHSM SIVTVYVAGE RHVVPDSATI L SRANQTIA ...文字列: MAVKSGARSG RNVVHLARPT LRETLGRLAP GTPLRDGLER ILRGRTGALI VLGYDDSVEA ICDGGFVLDV RYAPTRLREL SKMDGAVVL SSDGSRILRA NVQLVPDPSI PTDESGTRHR SAERTAIQTG YPVISVSHSM SIVTVYVAGE RHVVPDSATI L SRANQTIA TLERYKGRLD EVSRQLSTAE IEDFVTLRDV MTVVQRLEMV RRISLEIDAD VVELGTDGRQ LKLQLDELVG DN ETARELI VRDYHANPDP PTAAQVAATL EELDSLSDSE LLDFTVLARV FGYPSTAEAQ DSAMSSRGYR AMAAIPRLQF AHV DLLVRS FGSLQNLLAA SADDLQSVDG IGSMWARHIR EGLSLLAEST IADRLAAAAL EHHHHHH UniProtKB: DNA integrity scanning protein DisA |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 構成要素 - 濃度: 75.0 mM / 構成要素 - 式: NaCl / 構成要素 - 名称: Sodium Chloride 詳細: Buffer were made freshly with 75mM NaCl and 50mM Tris (pH 7.5) |
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK IV / 詳細: blotted for 3.5s. |
| 詳細 | The protein sample was made on a holey carbon grid with an additional layer of thin carbon. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 1 / 実像数: 1587 / 平均露光時間: 60.0 sec. / 平均電子線量: 27.75 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 倍率(補正後): 130841 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm 最大 デフォーカス(公称値): 3.3000000000000003 µm 最小 デフォーカス(公称値): 2.1 µm / 倍率(公称値): 75000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 詳細 | Model from AlphaFold was used as the starting point (AF-A0R564-F1-model_v2.pdb). |
|---|---|
| 精密化 | 空間: REAL / プロトコル: OTHER / 温度因子: 138.7 |
| 得られたモデル |  PDB-7y0d: |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)