+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | NuA4 HAT module bound to the nucleosome | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報: / PI5P Regulates TP53 Acetylation / piccolo histone acetyltransferase complex / SUMOylation of transcription cofactors / NuA4 histone acetyltransferase complex / positive regulation of macroautophagy / histone acetyltransferase complex / histone acetyltransferase activity / histone acetyltransferase / methylated histone binding ...: / PI5P Regulates TP53 Acetylation / piccolo histone acetyltransferase complex / SUMOylation of transcription cofactors / NuA4 histone acetyltransferase complex / positive regulation of macroautophagy / histone acetyltransferase complex / histone acetyltransferase activity / histone acetyltransferase / methylated histone binding / meiotic cell cycle / structural constituent of chromatin / nucleosome / chromatin organization / chromatin remodeling / protein heterodimerization activity / DNA repair / DNA-templated transcription / regulation of DNA-templated transcription / regulation of transcription by RNA polymerase II / DNA binding / nucleus / metal ion binding / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
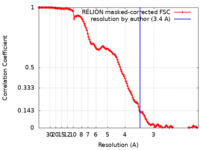
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||
 データ登録者 データ登録者 | Chen Z / Qu K | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2022 ジャーナル: Nature / 年: 2022タイトル: Structure of the NuA4 acetyltransferase complex bound to the nucleosome. 著者: Keke Qu / Kangjing Chen / Hao Wang / Xueming Li / Zhucheng Chen /  要旨: Deoxyribonucleic acid in eukaryotes wraps around the histone octamer to form nucleosomes, the fundamental unit of chromatin. The N termini of histone H4 interact with nearby nucleosomes and play an ...Deoxyribonucleic acid in eukaryotes wraps around the histone octamer to form nucleosomes, the fundamental unit of chromatin. The N termini of histone H4 interact with nearby nucleosomes and play an important role in the formation of high-order chromatin structure and heterochromatin silencing. NuA4 in yeast and its homologue Tip60 complex in mammalian cells are the key enzymes that catalyse H4 acetylation, which in turn regulates chromatin packaging and function in transcription activation and DNA repair. Here we report the cryo-electron microscopy structure of NuA4 from Saccharomyces cerevisiae bound to the nucleosome. NuA4 comprises two major modules: the catalytic histone acetyltransferase (HAT) module and the transcription activator-binding (TRA) module. The nucleosome is mainly bound by the HAT module and is positioned close to a polybasic surface of the TRA module, which is important for the optimal activity of NuA4. The nucleosomal linker DNA carrying the upstream activation sequence is oriented towards the conserved, transcription activator-binding surface of the Tra1 subunit, which suggests a potential mechanism of NuA4 to act as a transcription co-activator. The HAT module recognizes the disk face of the nucleosome through the H2A-H2B acidic patch and nucleosomal DNA, projecting the catalytic pocket of Esa1 to the N-terminal tail of H4 and supporting its function in selective acetylation of H4. Together, our findings illustrate how NuA4 is assembled and provide mechanistic insights into nucleosome recognition and transcription co-activation by a HAT. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_32148.map.gz emd_32148.map.gz | 4.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-32148-v30.xml emd-32148-v30.xml emd-32148.xml emd-32148.xml | 20.1 KB 20.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_32148_fsc.xml emd_32148_fsc.xml | 13.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_32148.png emd_32148.png | 60.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-32148 http://ftp.pdbj.org/pub/emdb/structures/EMD-32148 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32148 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32148 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_32148_validation.pdf.gz emd_32148_validation.pdf.gz | 334.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_32148_full_validation.pdf.gz emd_32148_full_validation.pdf.gz | 334.2 KB | 表示 | |
| XML形式データ |  emd_32148_validation.xml.gz emd_32148_validation.xml.gz | 13.4 KB | 表示 | |
| CIF形式データ |  emd_32148_validation.cif.gz emd_32148_validation.cif.gz | 18.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32148 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32148 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32148 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32148 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7vvuMC  7vvyC  7vvzC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_32148.map.gz / 形式: CCP4 / 大きさ: 202.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_32148.map.gz / 形式: CCP4 / 大きさ: 202.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.0825 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
+全体 : complex of NuA4 HAT module and nucleosome
+超分子 #1: complex of NuA4 HAT module and nucleosome
+分子 #1: Chromatin modification-related protein EAF6
+分子 #2: Chromatin modification-related protein YNG2
+分子 #3: Enhancer of polycomb-like protein 1
+分子 #4: Histone H3
+分子 #5: H4
+分子 #6: Histone H2A
+分子 #7: Histone H2B 1.1
+分子 #8: Histone acetyltransferase ESA1
+分子 #9: DNA (207-mer)
+分子 #10: DNA (207-mer)
+分子 #11: CARBOXYMETHYL COENZYME *A
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.6 |
|---|---|
| 凍結 | 凍結剤: NITROGEN |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: OTHER / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)