登録情報 データベース : EMDB / ID : EMD-32091タイトル Cryo-EM structure of Ams1 bound to the FW domain of Nbr1 複合体 : Ams1, Nbr1 and malE fusion proteinタンパク質・ペプチド : Ams1, Nbr1 and malE fusion proteinリガンド : ZINC IONリガンド : water / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
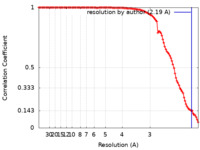
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Chaetomium thermophilum var. thermophilum DSM 1495 (菌類) / Escherichia coli K-12 (大腸菌)手法 / / 解像度 : 2.19 Å Zhang J / Ye K 資金援助 Organization Grant number 国 National Natural Science Foundation of China (NSFC) 32071199, 91940302 Chinese Academy of Sciences XDB37010201 National Basic Research Program of China (973 Program) 2017YFA0504600
ジャーナル : Nat Commun / 年 : 2022タイトル : Structural mechanism of protein recognition by the FW domain of autophagy receptor Nbr1著者 : Zhang J / Wang YY / Pan ZQ / Li Y / Sui J / Du LL / Ye K 履歴 登録 2021年10月20日 - ヘッダ(付随情報) 公開 2022年7月6日 - マップ公開 2022年7月6日 - 更新 2024年6月26日 - 現状 2024年6月26日 処理サイト : PDBj / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報
 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Chaetomium thermophilum var. thermophilum DSM 1495 (菌類) /
Chaetomium thermophilum var. thermophilum DSM 1495 (菌類) / 
 データ登録者
データ登録者 中国, 3件
中国, 3件  引用
引用 ジャーナル: Nat Commun / 年: 2022
ジャーナル: Nat Commun / 年: 2022 構造の表示
構造の表示 ダウンロードとリンク
ダウンロードとリンク emd_32091.map.gz
emd_32091.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-32091-v30.xml
emd-32091-v30.xml emd-32091.xml
emd-32091.xml EMDBヘッダ
EMDBヘッダ emd_32091_fsc.xml
emd_32091_fsc.xml FSCデータファイル
FSCデータファイル emd_32091.png
emd_32091.png emd_32091_msk_1.map
emd_32091_msk_1.map マスクマップ
マスクマップ emd-32091.cif.gz
emd-32091.cif.gz emd_32091_half_map_1.map.gz
emd_32091_half_map_1.map.gz emd_32091_half_map_2.map.gz
emd_32091_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-32091
http://ftp.pdbj.org/pub/emdb/structures/EMD-32091 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32091
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32091 emd_32091_validation.pdf.gz
emd_32091_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_32091_full_validation.pdf.gz
emd_32091_full_validation.pdf.gz emd_32091_validation.xml.gz
emd_32091_validation.xml.gz emd_32091_validation.cif.gz
emd_32091_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32091
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32091 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32091
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32091
 F&H 検索
F&H 検索 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_32091.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_32091.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) emd_32091_msk_1.map
emd_32091_msk_1.map 試料の構成要素
試料の構成要素 Chaetomium thermophilum var. thermophilum DSM 1495 (菌類)
Chaetomium thermophilum var. thermophilum DSM 1495 (菌類)


 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)