| 登録情報 | データベース: EMDB / ID: EMD-31030
|
|---|
| タイトル | Cryo-EM structure of the 2:1 Orc1 BAH domain in complex with nucleosome |
|---|
 マップデータ マップデータ | |
|---|
 試料 試料 | - 複合体: Yeast Orc1 BAH domain in complex with 147-bp nucleosome
- 複合体: Histone
- タンパク質・ペプチド: Histone H3
- タンパク質・ペプチド: Histone H4
- タンパク質・ペプチド: Histone H2A.2
- タンパク質・ペプチド: Histone H2B.2
- 複合体: DNA
- DNA: DNA (147-mer)
- DNA: DNA (147-mer)
- 複合体: Origin recognition complex subunit 1
- タンパク質・ペプチド: Origin recognition complex subunit 1
|
|---|
 キーワード キーワード | nucleosome / transcriptional silencing / chromatin / STRUCTURAL PROTEIN |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
CDC6 association with the ORC:origin complex / sexual sporulation resulting in formation of a cellular spore / cupric reductase (NADH) activity / HATs acetylate histones / global genome nucleotide-excision repair / RNA polymerase I upstream activating factor complex / maintenance of rDNA / Condensation of Prophase Chromosomes / SIRT1 negatively regulates rRNA expression / Activated PKN1 stimulates transcription of AR (androgen receptor) regulated genes KLK2 and KLK3 ...CDC6 association with the ORC:origin complex / sexual sporulation resulting in formation of a cellular spore / cupric reductase (NADH) activity / HATs acetylate histones / global genome nucleotide-excision repair / RNA polymerase I upstream activating factor complex / maintenance of rDNA / Condensation of Prophase Chromosomes / SIRT1 negatively regulates rRNA expression / Activated PKN1 stimulates transcription of AR (androgen receptor) regulated genes KLK2 and KLK3 / Assembly of the ORC complex at the origin of replication / HDACs deacetylate histones / nuclear origin of replication recognition complex / pre-replicative complex assembly involved in nuclear cell cycle DNA replication / Activation of the pre-replicative complex / nuclear pre-replicative complex / nucleosome organization / Activation of ATR in response to replication stress / DNA replication preinitiation complex / Recruitment and ATM-mediated phosphorylation of repair and signaling proteins at DNA double strand breaks / mitotic DNA replication checkpoint signaling / Oxidative Stress Induced Senescence / RMTs methylate histone arginines / SUMOylation of chromatin organization proteins / silent mating-type cassette heterochromatin formation / Orc1 removal from chromatin / RNA Polymerase I Promoter Escape / positive regulation of transcription by RNA polymerase I / nucleolar large rRNA transcription by RNA polymerase I / Estrogen-dependent gene expression / rRNA transcription / DNA replication origin binding / intracellular copper ion homeostasis / DNA replication initiation / Ub-specific processing proteases / nucleosome binding / CENP-A containing nucleosome / aerobic respiration / structural constituent of chromatin / nucleosome / heterochromatin formation / nucleosome assembly / chromatin organization / protein heterodimerization activity / DNA repair / chromatin binding / regulation of DNA-templated transcription / ATP hydrolysis activity / DNA binding / nucleoplasm / ATP binding / metal ion binding / identical protein binding / nucleus類似検索 - 分子機能 : / Origin recognition complex subunit 1 C-terminal winged HTH domain / AAA lid domain / AAA lid domain / : / Bromo adjacent homology domain / BAH domain / Bromo adjacent homology (BAH) domain / Bromo adjacent homology (BAH) domain superfamily / BAH domain profile. ...: / Origin recognition complex subunit 1 C-terminal winged HTH domain / AAA lid domain / AAA lid domain / : / Bromo adjacent homology domain / BAH domain / Bromo adjacent homology (BAH) domain / Bromo adjacent homology (BAH) domain superfamily / BAH domain profile. / : / Histone H2B signature. / Histone H2A conserved site / Histone H2A signature. / Histone H2B / Histone H2B / Histone H2A, C-terminal domain / C-terminus of histone H2A / Histone 2A / Histone H2A / Histone H4, conserved site / Histone H4 signature. / Histone H4 / Histone H4 / CENP-T/Histone H4, histone fold / Centromere kinetochore component CENP-T histone fold / ATPase family associated with various cellular activities (AAA) / ATPase, AAA-type, core / Histone H3 signature 1. / Histone H3 signature 2. / Histone H3 / Histone H3/CENP-A / Histone H2A/H2B/H3 / Core histone H2A/H2B/H3/H4 / Histone-fold / ATPases associated with a variety of cellular activities / AAA+ ATPase domain / P-loop containing nucleoside triphosphate hydrolase類似検索 - ドメイン・相同性 Histone H2B.2 / Histone H4 / Histone H2A.2 / Origin recognition complex subunit 1 / Histone H3類似検索 - 構成要素 |
|---|
| 生物種 |   Saccharomyces cerevisiae S288C (パン酵母) / Saccharomyces cerevisiae S288C (パン酵母) /   Escherichia coli (大腸菌) / Escherichia coli (大腸菌) /   Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母) Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母) |
|---|
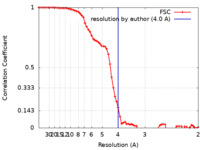
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.0 Å |
|---|
 データ登録者 データ登録者 | Jiang H / Yu C |
|---|
| 資金援助 |  中国, 2件 中国, 2件 | Organization | Grant number | 国 |
|---|
| Ministry of Science and Technology (MoST, China) | 2019YFA0508900, 2018YFE0203300 |  中国 中国 | | National Natural Science Foundation of China (NSFC) | 31991162, 91853204, 31521002 |  中国 中国 |
|
|---|
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Published
タイトル: Nucleosome binding relinquishes the association of the BAH domain of Orc1 with Sir1
著者: Jiang H / Yu C / Liu CP / Han X / Yu Z / Xu RM |
|---|
| 履歴 | | 登録 | 2021年3月4日 | - |
|---|
| ヘッダ(付随情報) 公開 | 2022年9月7日 | - |
|---|
| マップ公開 | 2022年9月7日 | - |
|---|
| 更新 | 2024年10月23日 | - |
|---|
| 現状 | 2024年10月23日 | 処理サイト: PDBj / 状態: 公開 |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報
 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報


 データ登録者
データ登録者 中国, 2件
中国, 2件  引用
引用 ジャーナル: To Be Published
ジャーナル: To Be Published 構造の表示
構造の表示 ダウンロードとリンク
ダウンロードとリンク emd_31030.map.gz
emd_31030.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-31030-v30.xml
emd-31030-v30.xml emd-31030.xml
emd-31030.xml EMDBヘッダ
EMDBヘッダ emd_31030_fsc.xml
emd_31030_fsc.xml FSCデータファイル
FSCデータファイル emd_31030.png
emd_31030.png emd-31030.cif.gz
emd-31030.cif.gz emd_31030_half_map_1.map.gz
emd_31030_half_map_1.map.gz emd_31030_half_map_2.map.gz
emd_31030_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-31030
http://ftp.pdbj.org/pub/emdb/structures/EMD-31030 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31030
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31030 emd_31030_validation.pdf.gz
emd_31030_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_31030_full_validation.pdf.gz
emd_31030_full_validation.pdf.gz emd_31030_validation.xml.gz
emd_31030_validation.xml.gz emd_31030_validation.cif.gz
emd_31030_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31030
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31030 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31030
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31030

 F&H 検索
F&H 検索 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_31030.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_31030.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 画像解析
画像解析
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)