+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of nucleosome-bound Sirtuin 6 deacetylase | |||||||||||||||
 マップデータ マップデータ | Main map from cisTEM, aligned to the common point of reference with the PDB and cryoSPARC map | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | Nucleosome / Sirt6 / aging / DNA damage / repair / deacetylation / diacylation / apo / chromatin / heterochromatin / GENE REGULATION / TRANSFERASE-DNA complex | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報NAD-dependent histone H3K56 deacetylase activity / NAD-dependent histone H3K18 deacetylase activity / NAD-dependent histone H3K9 deacetylase activity / protein delipidation / ketone biosynthetic process / regulation of lipid catabolic process / NAD+-protein-lysine ADP-ribosyltransferase activity / chromosome, subtelomeric region / NAD-dependent protein demyristoylase activity / NAD-dependent protein depalmitoylase activity ...NAD-dependent histone H3K56 deacetylase activity / NAD-dependent histone H3K18 deacetylase activity / NAD-dependent histone H3K9 deacetylase activity / protein delipidation / ketone biosynthetic process / regulation of lipid catabolic process / NAD+-protein-lysine ADP-ribosyltransferase activity / chromosome, subtelomeric region / NAD-dependent protein demyristoylase activity / NAD-dependent protein depalmitoylase activity / NAD+-protein-arginine ADP-ribosyltransferase activity / positive regulation of protein localization to chromatin / DNA damage sensor activity / positive regulation of stem cell differentiation / positive regulation of blood vessel branching / NAD-dependent protein lysine deacetylase activity / retrotransposon silencing / protein acetyllysine N-acetyltransferase / cardiac muscle cell differentiation / positive regulation of chondrocyte proliferation / pericentric heterochromatin formation / positive regulation of telomere maintenance / protein deacetylation / negative regulation of glucose import / protein localization to site of double-strand break / NAD-dependent histone deacetylase activity / TORC2 complex binding / lncRNA binding / negative regulation of glycolytic process / negative regulation of protein localization to chromatin / positive regulation of double-strand break repair / DNA repair-dependent chromatin remodeling / positive regulation of vascular endothelial cell proliferation / negative regulation of protein import into nucleus / regulation of double-strand break repair via homologous recombination / negative regulation of gene expression, epigenetic / positive regulation of stem cell population maintenance / positive regulation of stem cell proliferation / regulation of protein secretion / negative regulation of transcription elongation by RNA polymerase II / NAD+-protein ADP-ribosyltransferase activity / negative regulation of cellular senescence / site of DNA damage / regulation of lipid metabolic process / 転移酵素; グリコシル基を移すもの; 五炭糖残基を移すもの / NAD+-protein poly-ADP-ribosyltransferase activity / negative regulation of tumor necrosis factor-mediated signaling pathway / NAD+ binding / subtelomeric heterochromatin formation / positive regulation of fat cell differentiation / regulation of protein localization to plasma membrane / heterochromatin organization / protein localization to CENP-A containing chromatin / pericentric heterochromatin / negative regulation of gluconeogenesis / Replacement of protamines by nucleosomes in the male pronucleus / CENP-A containing nucleosome / Packaging Of Telomere Ends / response to UV / nucleosome binding / Recognition and association of DNA glycosylase with site containing an affected purine / Cleavage of the damaged purine / Deposition of new CENPA-containing nucleosomes at the centromere / nucleosomal DNA binding / 転移酵素; アシル基を移すもの; アミノアシル基以外のアシル基を移すもの / Recognition and association of DNA glycosylase with site containing an affected pyrimidine / Cleavage of the damaged pyrimidine / Inhibition of DNA recombination at telomere / Meiotic synapsis / RNA Polymerase I Promoter Opening / nucleotidyltransferase activity / Assembly of the ORC complex at the origin of replication / DNA methylation / Condensation of Prophase Chromosomes / positive regulation of protein export from nucleus / ERCC6 (CSB) and EHMT2 (G9a) positively regulate rRNA expression / SIRT1 negatively regulates rRNA expression / Chromatin modifications during the maternal to zygotic transition (MZT) / HCMV Late Events / innate immune response in mucosa / PRC2 methylates histones and DNA / Defective pyroptosis / HDACs deacetylate histones / determination of adult lifespan / RNA Polymerase I Promoter Escape / lipopolysaccharide binding / Nonhomologous End-Joining (NHEJ) / Transcriptional regulation by small RNAs / Formation of the beta-catenin:TCF transactivating complex / circadian regulation of gene expression / protein destabilization / RUNX1 regulates genes involved in megakaryocyte differentiation and platelet function / NoRC negatively regulates rRNA expression / Activated PKN1 stimulates transcription of AR (androgen receptor) regulated genes KLK2 and KLK3 / base-excision repair / B-WICH complex positively regulates rRNA expression / G2/M DNA damage checkpoint / regulation of circadian rhythm / positive regulation of insulin secretion / DNA Damage/Telomere Stress Induced Senescence 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) / | |||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.07 Å | |||||||||||||||
 データ登録者 データ登録者 | Chio US / Rechiche O / Bryll AR / Zhu J / Feldman JL / Peterson CL / Tan S / Armache J-P | |||||||||||||||
| 資金援助 |  米国, 4件 米国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2023 ジャーナル: Sci Adv / 年: 2023タイトル: Cryo-EM structure of the human Sirtuin 6-nucleosome complex. 著者: Un Seng Chio / Othman Rechiche / Alysia R Bryll / Jiang Zhu / Erik M Leith / Jessica L Feldman / Craig L Peterson / Song Tan / Jean-Paul Armache /  要旨: Sirtuin 6 (SIRT6) is a multifaceted protein deacetylase/deacylase and a major target for small-molecule modulators of longevity and cancer. In the context of chromatin, SIRT6 removes acetyl groups ...Sirtuin 6 (SIRT6) is a multifaceted protein deacetylase/deacylase and a major target for small-molecule modulators of longevity and cancer. In the context of chromatin, SIRT6 removes acetyl groups from histone H3 in nucleosomes, but the molecular basis for its nucleosomal substrate preference is unknown. Our cryo-electron microscopy structure of human SIRT6 in complex with the nucleosome shows that the catalytic domain of SIRT6 pries DNA from the nucleosomal entry-exit site and exposes the histone H3 N-terminal helix, while the SIRT6 zinc-binding domain binds to the histone acidic patch using an arginine anchor. In addition, SIRT6 forms an inhibitory interaction with the C-terminal tail of histone H2A. The structure provides insights into how SIRT6 can deacetylate both H3 K9 and H3 K56. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_29735.map.gz emd_29735.map.gz | 91.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-29735-v30.xml emd-29735-v30.xml emd-29735.xml emd-29735.xml | 36.6 KB 36.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_29735.png emd_29735.png | 89.6 KB | ||
| Filedesc metadata |  emd-29735.cif.gz emd-29735.cif.gz | 8.7 KB | ||
| その他 |  emd_29735_additional_1.map.gz emd_29735_additional_1.map.gz emd_29735_additional_2.map.gz emd_29735_additional_2.map.gz emd_29735_additional_3.map.gz emd_29735_additional_3.map.gz emd_29735_half_map_1.map.gz emd_29735_half_map_1.map.gz emd_29735_half_map_2.map.gz emd_29735_half_map_2.map.gz | 95.4 MB 91.5 MB 94.5 MB 10.4 MB 10.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-29735 http://ftp.pdbj.org/pub/emdb/structures/EMD-29735 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29735 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29735 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_29735_validation.pdf.gz emd_29735_validation.pdf.gz | 720.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_29735_full_validation.pdf.gz emd_29735_full_validation.pdf.gz | 720.5 KB | 表示 | |
| XML形式データ |  emd_29735_validation.xml.gz emd_29735_validation.xml.gz | 13.7 KB | 表示 | |
| CIF形式データ |  emd_29735_validation.cif.gz emd_29735_validation.cif.gz | 16.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29735 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29735 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29735 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29735 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8g57MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_29735.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_29735.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Main map from cisTEM, aligned to the common point of reference with the PDB and cryoSPARC map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Main map from cisTEM in original position
| ファイル | emd_29735_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Main map from cisTEM in original position | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
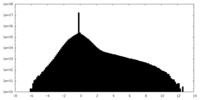
| 密度ヒストグラム |
-追加マップ: Sharpened main map from cisTEM, aligned to the...
| ファイル | emd_29735_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened main map from cisTEM, aligned to the common point of reference with the PDB and cryoSPARC map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Sharpened and aligned main map from cryoSPARC
| ファイル | emd_29735_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened and aligned main map from cryoSPARC | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 1 from cisTEM
| ファイル | emd_29735_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 from cisTEM | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 2 from cisTEM
| ファイル | emd_29735_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 from cisTEM | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Sirt6 deacetylase bound to a nucleosome assembled with 172-bp 601...
| 全体 | 名称: Sirt6 deacetylase bound to a nucleosome assembled with 172-bp 601 Widom DNA |
|---|---|
| 要素 |
|
-超分子 #1: Sirt6 deacetylase bound to a nucleosome assembled with 172-bp 601...
| 超分子 | 名称: Sirt6 deacetylase bound to a nucleosome assembled with 172-bp 601 Widom DNA タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 300 kDa/nm |
-分子 #1: NAD-dependent protein deacylase sirtuin-6
| 分子 | 名称: NAD-dependent protein deacylase sirtuin-6 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO EC番号: 転移酵素; アシル基を移すもの; アミノアシル基以外のアシル基を移すもの |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 39.708508 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GSMSVNYAAG LSPYADKGKC GLPEIFDPPE ELERKVWELA RLVWQSSNVV FHTGAGISTA SGIPDFRGPH GVWTMEERGL APKFDTTFE SARPTQTHMA LVQLERVGLL RFLVSQNVDG LHVRSGFPRD KLAELHGNMF VEECAKCKTQ YVRDTVVGTM G LKATGRLC ...文字列: GSMSVNYAAG LSPYADKGKC GLPEIFDPPE ELERKVWELA RLVWQSSNVV FHTGAGISTA SGIPDFRGPH GVWTMEERGL APKFDTTFE SARPTQTHMA LVQLERVGLL RFLVSQNVDG LHVRSGFPRD KLAELHGNMF VEECAKCKTQ YVRDTVVGTM G LKATGRLC TVAKARGLRA CRGELRDTIL DWEDSLPDRD LALADEASRN ADLSITLGTS LQIRPSGNLP LATKRRGGRL VI VNLQPTK HDRHADLRIH GYVDEVMTRL MKHLGLEIPA WDGPRVLERA LPPLPRPPTP KLEPKEESPT RINGSIPAGP KQE PCAQHN GSEPASPKRE RPTSPAPHRP PKRVKAKAVP SKLN UniProtKB: NAD-dependent protein deacylase sirtuin-6 |
-分子 #2: Histone H3
| 分子 | 名称: Histone H3 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 分子量 | 理論値: 15.004579 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: TKQTARKSTG GKAPRKQLAT KAARKSAPAT GGVKKPHRYR PGTVALREIR RYQKSTELLI RKLPFQRLVR EIAQDFKTDL RFQSSAVMA LQEASEAYLV ALFEDTNLCA IHAKRVTIMP KDIQLARRIR GER UniProtKB: Histone H3 |
-分子 #3: Histone H4
| 分子 | 名称: Histone H4 / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 分子量 | 理論値: 9.704396 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: HRKVLRDNIQ GITKPAIRRL ARRGGVKRIS GLIYEETRGV LKVFLENVIR DAVTYTEHAK RKTVTAMDVV YALKRQGRTL YGFGG UniProtKB: Histone H4 |
-分子 #4: Histone H2A type 1-B/E
| 分子 | 名称: Histone H2A type 1-B/E / タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 14.034355 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SGRGKQGGKA RAKAKTRSSR AGLQFPVGRV HRLLRKGNYS ERVGAGAPVY LAAVLEYLTA EILELAGNAA RDNKKTRIIP RHLQLAIRN DEELNKLLGR VTIAQGGVLP NIQAVLLPKK TESHHKAKGK UniProtKB: Histone H2A type 1-B/E |
-分子 #5: Histone H2B type 1-J
| 分子 | 名称: Histone H2B type 1-J / タイプ: protein_or_peptide / ID: 5 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 13.804045 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: PEPAKSAPAP KKGSKKAVTK AQKKDGKKRK RSRKESYSIY VYKVLKQVHP DTGISSKAMG IMNSFVNDIF ERIAGEASRL AHYNKRSTI TSREIQTAVR LLLPGELAKH AVSEGTKAVT KYTSAK UniProtKB: Histone H2B type 1-J |
-分子 #6: DNA strand 1
| 分子 | 名称: DNA strand 1 / タイプ: dna / ID: 6 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 46.541637 KDa |
| 配列 | 文字列: (DT)(DG)(DC)(DA)(DC)(DA)(DG)(DG)(DA)(DT) (DG)(DT)(DA)(DT)(DA)(DT)(DA)(DT)(DC)(DT) (DG)(DA)(DC)(DA)(DC)(DG)(DT)(DG)(DC) (DC)(DT)(DG)(DG)(DA)(DG)(DA)(DC)(DT)(DA) (DG) (DG)(DG)(DA)(DG)(DT) ...文字列: (DT)(DG)(DC)(DA)(DC)(DA)(DG)(DG)(DA)(DT) (DG)(DT)(DA)(DT)(DA)(DT)(DA)(DT)(DC)(DT) (DG)(DA)(DC)(DA)(DC)(DG)(DT)(DG)(DC) (DC)(DT)(DG)(DG)(DA)(DG)(DA)(DC)(DT)(DA) (DG) (DG)(DG)(DA)(DG)(DT)(DA)(DA)(DT) (DC)(DC)(DC)(DC)(DT)(DT)(DG)(DG)(DC)(DG) (DG)(DT) (DT)(DA)(DA)(DA)(DA)(DC)(DG) (DC)(DG)(DG)(DG)(DG)(DG)(DA)(DC)(DA)(DG) (DC)(DG)(DC) (DG)(DT)(DA)(DC)(DG)(DT) (DG)(DC)(DG)(DT)(DT)(DT)(DA)(DA)(DG)(DC) (DG)(DG)(DT)(DG) (DC)(DT)(DA)(DG)(DA) (DG)(DC)(DT)(DG)(DT)(DC)(DT)(DA)(DC)(DG) (DA)(DC)(DC)(DA)(DA) (DT)(DT)(DG)(DA) (DG)(DC)(DG)(DG)(DC)(DC)(DT)(DC)(DG)(DG) (DC)(DA)(DC)(DC)(DG)(DG) (DG)(DA)(DT) (DT)(DC)(DT)(DC)(DG)(DA)(DT) |
-分子 #7: DNA strand 2
| 分子 | 名称: DNA strand 2 / タイプ: dna / ID: 7 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 46.061348 KDa |
| 配列 | 文字列: (DA)(DT)(DC)(DG)(DA)(DG)(DA)(DA)(DT)(DC) (DC)(DC)(DG)(DG)(DT)(DG)(DC)(DC)(DG)(DA) (DG)(DG)(DC)(DC)(DG)(DC)(DT)(DC)(DA) (DA)(DT)(DT)(DG)(DG)(DT)(DC)(DG)(DT)(DA) (DG) (DA)(DC)(DA)(DG)(DC) ...文字列: (DA)(DT)(DC)(DG)(DA)(DG)(DA)(DA)(DT)(DC) (DC)(DC)(DG)(DG)(DT)(DG)(DC)(DC)(DG)(DA) (DG)(DG)(DC)(DC)(DG)(DC)(DT)(DC)(DA) (DA)(DT)(DT)(DG)(DG)(DT)(DC)(DG)(DT)(DA) (DG) (DA)(DC)(DA)(DG)(DC)(DT)(DC)(DT) (DA)(DG)(DC)(DA)(DC)(DC)(DG)(DC)(DT)(DT) (DA)(DA) (DA)(DC)(DG)(DC)(DA)(DC)(DG) (DT)(DA)(DC)(DG)(DC)(DG)(DC)(DT)(DG)(DT) (DC)(DC)(DC) (DC)(DC)(DG)(DC)(DG)(DT) (DT)(DT)(DT)(DA)(DA)(DC)(DC)(DG)(DC)(DC) (DA)(DA)(DG)(DG) (DG)(DG)(DA)(DT)(DT) (DA)(DC)(DT)(DC)(DC)(DC)(DT)(DA)(DG)(DT) (DC)(DT)(DC)(DC)(DA) (DG)(DG)(DC)(DA) (DC)(DG)(DT)(DG)(DT)(DC)(DA)(DG)(DA)(DT) (DA)(DT)(DA)(DT)(DA)(DC) (DA)(DT)(DC) (DC)(DT)(DG)(DT)(DG)(DC)(DA) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.2 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
詳細: 12.5 mM HEPES pH 7.5, 60 mM KCl, 1.5% glycerol, 1 mM DTT | |||||||||||||||
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 1 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 10 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.2 kPa 詳細: Glass slides were wrapped with fresh parafilm. Tweezers were washed with ethanol, dried, and then used to pick grids from a grid box. Grids were carefully examined and placed on the parafilm- ...詳細: Glass slides were wrapped with fresh parafilm. Tweezers were washed with ethanol, dried, and then used to pick grids from a grid box. Grids were carefully examined and placed on the parafilm-covered slides. These slides were then placed into the PelCO easyGLOW glow discharger, and treated there. | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV | |||||||||||||||
| 詳細 | SIRT6 deacetylase bound to asymmetrical nucleosome with 172 DNA base-pairs |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 64.0 K / 最高: 75.0 K |
| 特殊光学系 | 位相板: OTHER / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 2 / 実像数: 11872 / 平均露光時間: 3.3 sec. / 平均電子線量: 50.0 e/Å2 詳細: Data was collected in Super Resolution mode, thus the image size is 11520 (width) x 8184 (height) |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 最大 デフォーカス(補正後): 2.2 µm / 最小 デフォーカス(補正後): 1.0 µm / 倍率(補正後): 81000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 詳細 | UCSF Chimera was used for manual fitting of the models; It was then used for optimizing the fit by using option Fit in Map. Then Coot was used to analyze the fits, build and adjust the models into the existing densities, and refine parts of the model. Once the model has been built, validated and adjusted, we used phenix.real_space_refine to fix and improve the fit into the densities | ||||||||
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 温度因子: 79 | ||||||||
| 得られたモデル |  PDB-8g57: |
 ムービー
ムービー コントローラー
コントローラー














 Z
Z Y
Y X
X