+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of RNase-untreated RESC-A in trypanosomal RNA editing | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | HEAT repeat / trypanosoma / RNA editing substrate binding complex / gRNA / RNA BINDING PROTEIN-RNA complex | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of mitochondrial mRNA stability / mitochondrial mRNA processing / RNA modification / mitochondrial mRNA editing complex / RNA stabilization / RNA metabolic process / mitochondrial RNA modification / ribonucleoprotein granule / mitochondrial RNA processing / kinetoplast ...regulation of mitochondrial mRNA stability / mitochondrial mRNA processing / RNA modification / mitochondrial mRNA editing complex / RNA stabilization / RNA metabolic process / mitochondrial RNA modification / ribonucleoprotein granule / mitochondrial RNA processing / kinetoplast / RNA processing / mitochondrial matrix / mRNA binding / mitochondrion / RNA binding / cytoplasm 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  | ||||||||||||
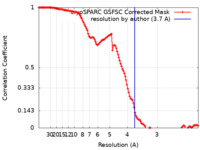
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.7 Å | ||||||||||||
 データ登録者 データ登録者 | Liu S / Wang H / Li X / Zhang F / Lee JKJ / Li Z / Yu C / Zhao X / Hu JJ / Suematsu T ...Liu S / Wang H / Li X / Zhang F / Lee JKJ / Li Z / Yu C / Zhao X / Hu JJ / Suematsu T / Alvarez-Cabrera AL / Liu Q / Zhang L / Huang L / Aphasizheva I / Aphasizhev R / Zhou ZH | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Science / 年: 2023 ジャーナル: Science / 年: 2023タイトル: Structural basis of gRNA stabilization and mRNA recognition in trypanosomal RNA editing. 著者: Shiheng Liu / Hong Wang / Xiaorun Li / Fan Zhang / Jane K J Lee / Zihang Li / Clinton Yu / Jason J Hu / Xiaojing Zhao / Takuma Suematsu / Ana L Alvarez-Cabrera / Qiushi Liu / Liye Zhang / Lan ...著者: Shiheng Liu / Hong Wang / Xiaorun Li / Fan Zhang / Jane K J Lee / Zihang Li / Clinton Yu / Jason J Hu / Xiaojing Zhao / Takuma Suematsu / Ana L Alvarez-Cabrera / Qiushi Liu / Liye Zhang / Lan Huang / Inna Aphasizheva / Ruslan Aphasizhev / Z Hong Zhou /   要旨: In , the editosome, composed of RNA-editing substrate-binding complex (RESC) and RNA-editing catalytic complex (RECC), orchestrates guide RNA (gRNA)-programmed editing to recode cryptic mitochondrial ...In , the editosome, composed of RNA-editing substrate-binding complex (RESC) and RNA-editing catalytic complex (RECC), orchestrates guide RNA (gRNA)-programmed editing to recode cryptic mitochondrial transcripts into messenger RNAs (mRNAs). The mechanism of information transfer from gRNA to mRNA is unclear owing to a lack of high-resolution structures for these complexes. With cryo-electron microscopy and functional studies, we have captured gRNA-stabilizing RESC-A and gRNA-mRNA-binding RESC-B and RESC-C particles. RESC-A sequesters gRNA termini, thus promoting hairpin formation and blocking mRNA access. The conversion of RESC-A into RESC-B or -C unfolds gRNA and allows mRNA selection. The ensuing gRNA-mRNA duplex protrudes from RESC-B, likely exposing editing sites to RECC-catalyzed cleavage, uridine insertion or deletion, and ligation. Our work reveals a remodeling event facilitating gRNA-mRNA hybridization and assembly of a macromolecular substrate for the editosome's catalytic modality. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_29306.map.gz emd_29306.map.gz | 168 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-29306-v30.xml emd-29306-v30.xml emd-29306.xml emd-29306.xml | 24.3 KB 24.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_29306_fsc.xml emd_29306_fsc.xml | 11.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_29306.png emd_29306.png | 101.1 KB | ||
| Filedesc metadata |  emd-29306.cif.gz emd-29306.cif.gz | 8.2 KB | ||
| その他 |  emd_29306_half_map_1.map.gz emd_29306_half_map_1.map.gz emd_29306_half_map_2.map.gz emd_29306_half_map_2.map.gz | 165.3 MB 165.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-29306 http://ftp.pdbj.org/pub/emdb/structures/EMD-29306 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29306 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29306 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_29306_validation.pdf.gz emd_29306_validation.pdf.gz | 870.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_29306_full_validation.pdf.gz emd_29306_full_validation.pdf.gz | 869.8 KB | 表示 | |
| XML形式データ |  emd_29306_validation.xml.gz emd_29306_validation.xml.gz | 20.5 KB | 表示 | |
| CIF形式データ |  emd_29306_validation.cif.gz emd_29306_validation.cif.gz | 26.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29306 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29306 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29306 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29306 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_29306.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_29306.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_29306_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_29306_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : RESC5-tagged isolate without RNase treatment
| 全体 | 名称: RESC5-tagged isolate without RNase treatment |
|---|---|
| 要素 |
|
-超分子 #1: RESC5-tagged isolate without RNase treatment
| 超分子 | 名称: RESC5-tagged isolate without RNase treatment / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #2-#7 詳細: CTS-tagged RESC5 purified from RNase-untreated mitochondrial extract by tandem affinity procedure |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: gRNA
| 分子 | 名称: gRNA / タイプ: rna / ID: 1 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 14.499465 KDa |
| 配列 | 文字列: UAUAUUUUUU UUUUUUUUUU UUUAAAAAAA AAAAAAAAAA AUUUUU |
-分子 #2: RNA-editing substrate-binding complex protein 1 (RESC1)
| 分子 | 名称: RNA-editing substrate-binding complex protein 1 (RESC1) タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 52.60868 KDa |
| 配列 | 文字列: MLRLLRRSIV GSTFNIMVRR QNQGSVSQGA LNMRDQQAAA AENVTPERVW ALWNEGNLFS LSLAQLQGFL SRCGVRTDPA AKKAAVVRQ VEEYLHSKDT TVKGGGQGAA SPQQHQQHGQ QGGYGRWNQA SVMQPETLLD LSQAGFYEGA ANMVPKAFQL L VSDTAPDV ...文字列: MLRLLRRSIV GSTFNIMVRR QNQGSVSQGA LNMRDQQAAA AENVTPERVW ALWNEGNLFS LSLAQLQGFL SRCGVRTDPA AKKAAVVRQ VEEYLHSKDT TVKGGGQGAA SPQQHQQHGQ QGGYGRWNQA SVMQPETLLD LSQAGFYEGA ANMVPKAFQL L VSDTAPDV VVSRVNTTAF PGFPSNTECY TLGASEKDVA IRSRYSKVLQ WCCLNMSNLQ MDGELYVDFG KLLLKPSVMR KN RRIVSSY TLQQRLQVNH PYTWVPTLPE SCLSKIQEQF LQPEGFAPIG KGVQLTYSGT IKRSKDQLHV DLDNKGKVLA VNS AWVNLQ TAWCTHAKGP DVRLLLRSRP PIRRQDVELF ASTPIIKLAD DDVADVLPPE HGQLVYLSED ETRLFERVSD RGVT ITVRE VKRQPLIILR DEEEDPRVEY SLSAHIPANA AKATDVRAVG LTAFELAGRL AGLVAEDFVR EYGCEAKL UniProtKB: Guide RNA associated protein, GAP2 |
-分子 #3: RNA-editing substrate-binding complex protein 2 (RESC2)
| 分子 | 名称: RNA-editing substrate-binding complex protein 2 (RESC2) タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 55.526117 KDa |
| 配列 | 文字列: MLRARLKIFS ALNGATSAFS RAVAPLQIAT RQQSFSAAAP AASGDFSHIT RNTVWGLWNE GNLFSLSVPE LAFFLQEHCR VANVDPRAK KSALVRQVEE ILSAEQQASA TVPQEDNPHA IVVTDYDRAE DALEEADEYG DWGAEPGFED RRELDFMELS P GRMGERYD ...文字列: MLRARLKIFS ALNGATSAFS RAVAPLQIAT RQQSFSAAAP AASGDFSHIT RNTVWGLWNE GNLFSLSVPE LAFFLQEHCR VANVDPRAK KSALVRQVEE ILSAEQQASA TVPQEDNPHA IVVTDYDRAE DALEEADEYG DWGAEPGFED RRELDFMELS P GRMGERYD PLSPRAFQLL HSETATDVGI ASIDPSKLPG QSKVKNALAA IHVAPNDANK MRFRMAFEWC LMNIWNMNMP GE LNIGAGK ALYYRSVAKQ NRNVMPLWTV QKHLYAQHPY AWFAIASESN VAAMESLAAA LNMSIQQERT TSYKVTIRRM AEF FDCELN GQLKCTMMNK PWDRFFVSHY IRSKMPDLRY VVRARHPIKK RIADAYLEAD ILRSTRDSVQ SVLSPELGDV VYCC ERVVR KWAKKTATGV TLQLVETKRT PLIITKAGDE GERLEYEWIV PLPQQAERID IAALTDELWE YGNKLAAALE EGMEE LMVH TMTAVSAY UniProtKB: Mitochondrial guide RNA binding complex subunit 2 |
-分子 #4: RNA-editing substrate-binding complex protein 3 (RESC3)
| 分子 | 名称: RNA-editing substrate-binding complex protein 3 (RESC3) タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 53.146617 KDa |
| 配列 | 文字列: MSNPFEKVAR GIAFKMRSKV HKQGYSNTVM AQQARRLSPT GLLAMERLTE LTALQQRHQC TFDPALRSKA TQILRTLPLL SIDEDPYFT HTQRALRLAA YFGAVDLPVT YALINQHTKN AFMLDAFSMA SFFYTLAKLK HPQTKEIVGI LLPRLREVAP E LIAREAVH ...文字列: MSNPFEKVAR GIAFKMRSKV HKQGYSNTVM AQQARRLSPT GLLAMERLTE LTALQQRHQC TFDPALRSKA TQILRTLPLL SIDEDPYFT HTQRALRLAA YFGAVDLPVT YALINQHTKN AFMLDAFSMA SFFYTLAKLK HPQTKEIVGI LLPRLREVAP E LIAREAVH ILRLLCSIQM ADAQLVKVVT ETVVATAADV PLRDARQCAF ILSETFPEEA QRILGAVEHR LCDDIDMNAD AN EVKTTIL DVCRVVSATC KGPRRLLNSV ARRSMELLPQ LTPLDVAFVL KAFHLSSYRH LRLLRVLSSS LAASFPTSNV TKE HGLAAS IVVQSLAHFY LSGCEEVVVT LVNASVNVLE GLNLALTLLA CVRLRCVSPG VDPAVDALCS GAPMRRYVHN AHSM QVTSR ILYGLAHAGR CRSDEEVAIV LPLLKSVVRT PGALRDDCRG FLLDAVTALG ADGECSNDAL QEQVRKVYER LSQDG GK UniProtKB: Mitochondrial RNA binding protein |
-分子 #5: RNA-editing substrate-binding complex protein 4 (RESC4)
| 分子 | 名称: RNA-editing substrate-binding complex protein 4 (RESC4) タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 120.847914 KDa |
| 配列 | 文字列: MNGRLYCLIR RITSPPVATR LIKEELCLSM AAIARLPLRR DQLAHVTNTE AITTRAQRIS HLCTPTELGM IAEGAEALSC NRFDLADAL IDGAYESVRR AASSTRLSHV SAIARYSASI KTYGNETITT LLKAGASLLQ KNDSVPVLKS FLGVAQSHLT D GEMRVLID ...文字列: MNGRLYCLIR RITSPPVATR LIKEELCLSM AAIARLPLRR DQLAHVTNTE AITTRAQRIS HLCTPTELGM IAEGAEALSC NRFDLADAL IDGAYESVRR AASSTRLSHV SAIARYSASI KTYGNETITT LLKAGASLLQ KNDSVPVLKS FLGVAQSHLT D GEMRVLID EMCAKATEEQ RLCINSIGTQ SLAKDAAKCG EETLTKGNED GDETAVDDEE TQAWDMLRAR QWMLQLVRCG KP PTAAEAV QAMELYAHFA VRDFVLHEKI EDLVLLVLPT GNKFHLNEMH KIVLRSPNLF PRVRNTLGQD HSGVSDVHRA DRG VEWSDD PASSLTTTYT TSRAYSMLLL GQRLSEDIMF DVVQEQSETI PVDVAAQAAC LFAEKGDIPE GVILRLSAEL EHIS PQGVT AFVRAARRDS SGALLPHYAA VLNRFTERDL CDTPLETLLQ MCEVFALPAP RGTSEGDNDS INESQSKFQK ALIVR LFSV IQGSRDVPFL CKVAKAVRAF DANDELIQFV CSSICAQGAL SECEALIAFD MIRCCDFVYE PLLDAMEPVF RRLVES VSA MLEGKSTIND VEVRRCACFA TLQSEFDCPD FETLASLLVH TVEKNVTGCP VELIPSVGLL CVRTRRTSAL YIVGNKL EG NMQQLSDDAI GELARLLVGT ENLATKELAV EFQSVVVSRL LRQQSLPPDV VALSAVVWLR QGDKVGTIDE RSVDYIIK W MYAIGSSVYT DLCLAVHLSA SVESLSNALI DDLPRRLELL TTNEMANAIF GLGEVSDMGA RLSHQLVAER CSDYVVDHS QEFWSGKVIA RLLYGFSRMH CTKRSLYNVF ATRLAHRPVF SLLDQEAISF AIAAFGRVKY LDKKLFDRFT RWILDHSKDL NAAELLLTI RGVSRVMLLN DQLYDDLGSK AAEKVKEFPI ESQCVLLSSF GSLGVEHERL ASRMVSSIAE NREELTDATK A VDVITSLW SMNYDVEDDK HVAQLADWVV QRAEELTDES IGKLCLVLSD TNWRHVPLVR AIAEQSVRLQ GQQSISPKCC RE VLDVLGT FMIHHQGARE NLSALGRSIS KERIQLSEEE EQHLQLLLRR UniProtKB: Mitochondrial RNA binding complex 1 subunit |
-分子 #6: RNA-editing substrate-binding complex protein 5 (RESC5)
| 分子 | 名称: RNA-editing substrate-binding complex protein 5 (RESC5) タイプ: protein_or_peptide / ID: 6 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 43.469484 KDa |
| 配列 | 文字列: MLRHTSRNNA LHAFVRSPHY RTIPSAGPNG IVVNRDMLVH QFRDFYKTLQ HCSLVDKVHL MSERPSVEAL RVADQMVSIG ATFLEMPLT GMEHRATEFM ESMRYVRGAG GPSTLASYLQ DTENCRCNSG DVVCLPNGIA VGHGPRTNAV AHTTLKQLFE V KDDQFSFD ...文字列: MLRHTSRNNA LHAFVRSPHY RTIPSAGPNG IVVNRDMLVH QFRDFYKTLQ HCSLVDKVHL MSERPSVEAL RVADQMVSIG ATFLEMPLT GMEHRATEFM ESMRYVRGAG GPSTLASYLQ DTENCRCNSG DVVCLPNGIA VGHGPRTNAV AHTTLKQLFE V KDDQFSFD VFTLEQEGDA PPLGDYFGFA GSNVLLTWKD EHGLLAVDQY QQKQPHTEMN VVYLEPGCHF LSFYGVDHTI DV LVQKGYE RSMDSIAAAG LNPIPVQWSE MDKLGISMRA AVLPLKFFKA NVGGMLSRNK SRGARWQTHQ LQKGSGSGSA SSG ASAAGS SGASASSGAS AAGSSGASAG HHHHHHHHHH SGSEDQVDPR LIDGKASAWS HPQFEKGGGS GGGSGGSAWS HPQF EK UniProtKB: Mitochondrial RNA binding protein |
-分子 #7: RNA-editing substrate-binding complex protein 6 (RESC6)
| 分子 | 名称: RNA-editing substrate-binding complex protein 6 (RESC6) タイプ: protein_or_peptide / ID: 7 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 57.823484 KDa |
| 配列 | 文字列: MRSALRRCIL RHQGCLRMKQ SLSAFPTVVT GMTRHQGNSL IGTTHGAELS LAGDPQSVSH LSARNIATEA LQMKKLHQER GGNPMLAQQ ARRVLFATSI AGQNLDARSV ALLLNTAVYF GMESDAKLVR ECIDYCLKND KLITVDVLPI VVTACATLKS R DAREVIEM ...文字列: MRSALRRCIL RHQGCLRMKQ SLSAFPTVVT GMTRHQGNSL IGTTHGAELS LAGDPQSVSH LSARNIATEA LQMKKLHQER GGNPMLAQQ ARRVLFATSI AGQNLDARSV ALLLNTAVYF GMESDAKLVR ECIDYCLKND KLITVDVLPI VVTACATLKS R DAREVIEM QAQKAARNAK FLDAKDVTNI ISAFSKTGIN HEKLFAFLSR RVQTLARVGE FEAAHLVILA NAFSRLRYRD KF LFGAIAR RAMSLRERVT VNELVPLIVA FSKIGLKDPK LSKRFATKAM EYVDQMNAEQ VASMFMAFAY FGIRYDQLFG VLT NRAVEL IDEFNAQYIS TTLNAFQRIG INNPELFDNL AERALAVVQD HDARDISKTV TALAHFGLKD EELFKRLASH AASI ADQFD AMGLVNTAHA FARTNFLQQD MAVALSERSV YVCRLLDAGE TRRLLWALAK FQVRDPKILT PVFNRCLALH YDFFA DPTG SEEIEEIFDF YGPNFCPPLY QLYISRGSTP QA UniProtKB: Guide RNA binding protein |
-分子 #8: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 8 / コピー数: 1 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 281.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 検出モード: SUPER-RESOLUTION / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)