+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 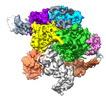 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
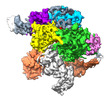
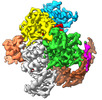
| タイトル | ISDra2 TnpB in complex with reRNA and cognate DNA, conformation 1 (RuvC domain resolved) | |||||||||||||||
 マップデータ マップデータ | Sharpened map. Output of phenix.auto_refine | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | Transposon / TnpB / RuvC domain / Cas12 / IS200/IS605 / reRNA / RNA BINDING PROTEIN | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報transposition / 加水分解酵素; エステル加水分解酵素; 5'-リン酸モノエステル産生エンドデオキシリボヌクレアーゼ / DNA recombination / endonuclease activity / DNA binding / RNA binding / metal ion binding 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  Deinococcus radiodurans R1 (放射線耐性) / synthetic construct (人工物) Deinococcus radiodurans R1 (放射線耐性) / synthetic construct (人工物) | |||||||||||||||
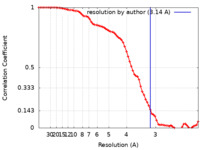
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.14 Å | |||||||||||||||
 データ登録者 データ登録者 | Sasnauskas G / Tamulaitiene G / Carabias A / Karvelis T / Druteika G / Silanskas A / Montoya G / Venclovas C / Kazlauskas D / Siksnys V | |||||||||||||||
| 資金援助 |  デンマーク, 4件 デンマーク, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2023 ジャーナル: Nature / 年: 2023タイトル: TnpB structure reveals minimal functional core of Cas12 nuclease family. 著者: Giedrius Sasnauskas / Giedre Tamulaitiene / Gytis Druteika / Arturo Carabias / Arunas Silanskas / Darius Kazlauskas / Česlovas Venclovas / Guillermo Montoya / Tautvydas Karvelis / Virginijus Siksnys /  要旨: The widespread TnpB proteins of IS200/IS605 transposon family have recently emerged as the smallest RNA-guided nucleases capable of targeted genome editing in eukaryotic cells. Bioinformatic analysis ...The widespread TnpB proteins of IS200/IS605 transposon family have recently emerged as the smallest RNA-guided nucleases capable of targeted genome editing in eukaryotic cells. Bioinformatic analysis identified TnpB proteins as the likely predecessors of Cas12 nucleases, which along with Cas9 are widely used for targeted genome manipulation. Whereas Cas12 family nucleases are well characterized both biochemically and structurally, the molecular mechanism of TnpB remains unknown. Here we present the cryogenic-electron microscopy structures of the Deinococcus radiodurans TnpB-reRNA (right-end transposon element-derived RNA) complex in DNA-bound and -free forms. The structures reveal the basic architecture of TnpB nuclease and the molecular mechanism for DNA target recognition and cleavage that is supported by biochemical experiments. Collectively, these results demonstrate that TnpB represents the minimal structural and functional core of the Cas12 protein family and provide a framework for developing TnpB-based genome editing tools. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28656.map.gz emd_28656.map.gz | 28.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28656-v30.xml emd-28656-v30.xml emd-28656.xml emd-28656.xml | 23.6 KB 23.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_28656_fsc.xml emd_28656_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_28656.png emd_28656.png | 149.3 KB | ||
| マスクデータ |  emd_28656_msk_1.map emd_28656_msk_1.map | 30.5 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-28656.cif.gz emd-28656.cif.gz | 6.7 KB | ||
| その他 |  emd_28656_additional_1.map.gz emd_28656_additional_1.map.gz emd_28656_half_map_1.map.gz emd_28656_half_map_1.map.gz emd_28656_half_map_2.map.gz emd_28656_half_map_2.map.gz | 15.4 MB 28.4 MB 28.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28656 http://ftp.pdbj.org/pub/emdb/structures/EMD-28656 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28656 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28656 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_28656_validation.pdf.gz emd_28656_validation.pdf.gz | 778.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_28656_full_validation.pdf.gz emd_28656_full_validation.pdf.gz | 778 KB | 表示 | |
| XML形式データ |  emd_28656_validation.xml.gz emd_28656_validation.xml.gz | 12.7 KB | 表示 | |
| CIF形式データ |  emd_28656_validation.cif.gz emd_28656_validation.cif.gz | 17.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28656 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28656 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28656 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28656 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28656.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28656.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map. Output of phenix.auto_refine | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_28656_msk_1.map emd_28656_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Unsharpened map. Output of CryoSPARC local refinement job.
| ファイル | emd_28656_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened map. Output of CryoSPARC local refinement job. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Output of CryoSPARC local refinement job.
| ファイル | emd_28656_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Output of CryoSPARC local refinement job. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Output of CryoSPARC local refinement job.
| ファイル | emd_28656_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Output of CryoSPARC local refinement job. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : ISDra2 TnpB in complex with reRNA and cognate DNA
| 全体 | 名称: ISDra2 TnpB in complex with reRNA and cognate DNA |
|---|---|
| 要素 |
|
-超分子 #1: ISDra2 TnpB in complex with reRNA and cognate DNA
| 超分子 | 名称: ISDra2 TnpB in complex with reRNA and cognate DNA / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Deinococcus radiodurans R1 (放射線耐性) Deinococcus radiodurans R1 (放射線耐性) |
-分子 #1: RNA-guided DNA endonuclease TnpB
| 分子 | 名称: RNA-guided DNA endonuclease TnpB / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO EC番号: 加水分解酵素; エステル加水分解酵素; 5'-リン酸モノエステル産生エンドデオキシリボヌクレアーゼ |
|---|---|
| 由来(天然) | 生物種:  Deinococcus radiodurans R1 (放射線耐性) Deinococcus radiodurans R1 (放射線耐性)株: ATCC 13939 / DSM 20539 / JCM 16871 / LMG 4051 / NBRC 15346 / NCIMB 9279 / R1 / VKM B-1422 |
| 分子量 | 理論値: 46.484289 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MIRNKAFVVR LYPNAAQTEL INRTLGSARF VYNHFLARRI AAYKESGKGL TYGQTSSELT LLKQAEETSW LSEVDKFALQ NSLKNLETA YKNFFRTVKQ SGKKVGFPRF RKKRTGESYR TQFTNNNIQI GEGRLKLPKL GWVKTKGQQD IQGKILNVTV R RIHEGHYE ...文字列: MIRNKAFVVR LYPNAAQTEL INRTLGSARF VYNHFLARRI AAYKESGKGL TYGQTSSELT LLKQAEETSW LSEVDKFALQ NSLKNLETA YKNFFRTVKQ SGKKVGFPRF RKKRTGESYR TQFTNNNIQI GEGRLKLPKL GWVKTKGQQD IQGKILNVTV R RIHEGHYE ASVLCEVEIP YLPAAPKFAA GVDVGIKDFA IVTDGVRFKH EQNPKYYRST LKRLRKAQQT LSRRKKGSAR YG KAKTKLA RIHKRIVNKR QDFLHKLTTS LVREYEIIGT EHLKPDNMRK NRRLALSISD AGWGEFIRQL EYKAAWYGRL VSK VSPYFP SSQLCHDCGF KNPEVKNLAV RTWTCPNCGE THDRDENAAL NIRREALVAA GISDTLNAHG GYVRPASAGN GLRS ENHAT LVV UniProtKB: RNA-guided DNA endonuclease TnpB |
-分子 #2: RNA (150-MER)
| 分子 | 名称: RNA (150-MER) / タイプ: rna / ID: 2 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  Deinococcus radiodurans R1 (放射線耐性) Deinococcus radiodurans R1 (放射線耐性) |
| 分子量 | 理論値: 48.408633 KDa |
| 配列 | 文字列: CAUUCGGCGU GAAGCGUUGG UGGCUGCGGG AAUCUCAGAC ACCUUAAACG CUCAUGGAGG CUAUGUCAGA CCUGCUUCGG CGGGCAAUG GUCUGCGAAG UGAGAAUCAC GCGACUUUAG UCGUGUGAGG UUCAAGAGUC CCUUGGCGCC C GENBANK: GENBANK: AE000513.1 |
-分子 #3: DNA (43-MER)
| 分子 | 名称: DNA (43-MER) / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 13.310532 KDa |
| 配列 | 文字列: (DG)(DT)(DC)(DA)(DT)(DG)(DG)(DG)(DC)(DG) (DC)(DC)(DA)(DA)(DG)(DG)(DG)(DA)(DC)(DT) (DC)(DA)(DT)(DC)(DA)(DA)(DG)(DC)(DG) (DA)(DC)(DG)(DG)(DT)(DT)(DG)(DA)(DT)(DC) (DT) (DC)(DA)(DG) |
-分子 #4: DNA (43-MER)
| 分子 | 名称: DNA (43-MER) / タイプ: dna / ID: 4 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 13.221472 KDa |
| 配列 | 文字列: (DG)(DT)(DC)(DA)(DT)(DG)(DG)(DG)(DC)(DG) (DC)(DC)(DA)(DA)(DG)(DG)(DG)(DA)(DC)(DT) (DC)(DA)(DT)(DC)(DA)(DA)(DC)(DC)(DG) (DT)(DC)(DG)(DC)(DT)(DT)(DG)(DA)(DT)(DC) (DT) (DC)(DA)(DG) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.7 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 支持フィルム - Film thickness: 12 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 45 sec. / 前処理 - 雰囲気: AIR | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / 実像数: 1981 / 平均電子線量: 30.6 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 92000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
 ムービー
ムービー コントローラー
コントローラー







 Z
Z Y
Y X
X