+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of human ADAM10-Tspan15 complex bound to 11G2 vFab | |||||||||
 マップデータ マップデータ | Map post-processed with DeepEMhancer using the high resolution model | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Protease / Metalloprotease / Tetraspanin / Sheddase / Adhesion / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of membrane protein ectodomain proteolysis / constitutive protein ectodomain proteolysis / ADAM10 endopeptidase / regulation of vasculature development / epidermal growth factor receptor ligand maturation / monocyte activation / metalloendopeptidase activity involved in amyloid precursor protein catabolic process / postsynapse organization / protein catabolic process at postsynapse / Constitutive Signaling by NOTCH1 t(7;9)(NOTCH1:M1580_K2555) Translocation Mutant ...regulation of membrane protein ectodomain proteolysis / constitutive protein ectodomain proteolysis / ADAM10 endopeptidase / regulation of vasculature development / epidermal growth factor receptor ligand maturation / monocyte activation / metalloendopeptidase activity involved in amyloid precursor protein catabolic process / postsynapse organization / protein catabolic process at postsynapse / Constitutive Signaling by NOTCH1 t(7;9)(NOTCH1:M1580_K2555) Translocation Mutant / pore complex assembly / regulation of Notch signaling pathway / perinuclear endoplasmic reticulum / positive regulation of T cell chemotaxis / tetraspanin-enriched microdomain / metallodipeptidase activity / NOTCH4 Activation and Transmission of Signal to the Nucleus / negative regulation of cell adhesion / adherens junction organization / regulation of neurotransmitter receptor localization to postsynaptic specialization membrane / clathrin-coated vesicle / regulation of postsynapse organization / Golgi-associated vesicle / cochlea development / Signaling by EGFR / negative regulation of Notch signaling pathway / pore complex / amyloid precursor protein catabolic process / tertiary granule membrane / Collagen degradation / protein maturation / membrane protein ectodomain proteolysis / EPH-ephrin mediated repulsion of cells / extracellular matrix disassembly / response to tumor necrosis factor / specific granule membrane / Notch signaling pathway / Constitutive Signaling by NOTCH1 HD Domain Mutants / NOTCH2 Activation and Transmission of Signal to the Nucleus / Degradation of the extracellular matrix / Activated NOTCH1 Transmits Signal to the Nucleus / synaptic membrane / protein localization to plasma membrane / integrin-mediated signaling pathway / NOTCH3 Activation and Transmission of Signal to the Nucleus / adherens junction / Post-translational protein phosphorylation / protein processing / metalloendopeptidase activity / Constitutive Signaling by NOTCH1 PEST Domain Mutants / Constitutive Signaling by NOTCH1 HD+PEST Domain Mutants / SH3 domain binding / Regulation of Insulin-like Growth Factor (IGF) transport and uptake by Insulin-like Growth Factor Binding Proteins (IGFBPs) / metallopeptidase activity / integrin binding / cell junction / cell-cell signaling / late endosome membrane / positive regulation of cell growth / endopeptidase activity / in utero embryonic development / molecular adaptor activity / postsynaptic density / nuclear body / positive regulation of cell migration / Amyloid fiber formation / endoplasmic reticulum lumen / Golgi membrane / axon / protein phosphorylation / negative regulation of gene expression / intracellular membrane-bounded organelle / focal adhesion / signaling receptor binding / glutamatergic synapse / dendrite / positive regulation of cell population proliferation / Neutrophil degranulation / protein kinase binding / Golgi apparatus / enzyme binding / cell surface / protein homodimerization activity / extracellular exosome / membrane / nucleus / metal ion binding / plasma membrane / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Lipper CH / Blacklow SC | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2023 ジャーナル: Cell / 年: 2023タイトル: Structural basis for membrane-proximal proteolysis of substrates by ADAM10. 著者: Colin H Lipper / Emily D Egan / Khal-Hentz Gabriel / Stephen C Blacklow /  要旨: The endopeptidase ADAM10 is a critical catalyst for the regulated proteolysis of key drivers of mammalian development, physiology, and non-amyloidogenic cleavage of APP as the primary α-secretase. ...The endopeptidase ADAM10 is a critical catalyst for the regulated proteolysis of key drivers of mammalian development, physiology, and non-amyloidogenic cleavage of APP as the primary α-secretase. ADAM10 function requires the formation of a complex with a C8-tetraspanin protein, but how tetraspanin binding enables positioning of the enzyme active site for membrane-proximal cleavage remains unknown. We present here a cryo-EM structure of a vFab-ADAM10-Tspan15 complex, which shows that Tspan15 binding relieves ADAM10 autoinhibition and acts as a molecular measuring stick to position the enzyme active site about 20 Å from the plasma membrane for membrane-proximal substrate cleavage. Cell-based assays of N-cadherin shedding establish that the positioning of the active site by the interface between the ADAM10 catalytic domain and the bound tetraspanin influences selection of the preferred cleavage site. Together, these studies reveal the molecular mechanism underlying ADAM10 proteolysis at membrane-proximal sites and offer a roadmap for its modulation in disease. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28580.map.gz emd_28580.map.gz | 88.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28580-v30.xml emd-28580-v30.xml emd-28580.xml emd-28580.xml | 23.7 KB 23.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_28580.png emd_28580.png | 14.4 KB | ||
| その他 |  emd_28580_half_map_1.map.gz emd_28580_half_map_1.map.gz emd_28580_half_map_2.map.gz emd_28580_half_map_2.map.gz | 95.6 MB 95.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28580 http://ftp.pdbj.org/pub/emdb/structures/EMD-28580 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28580 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28580 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8esvMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28580.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28580.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map post-processed with DeepEMhancer using the high resolution model | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.32 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_28580_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
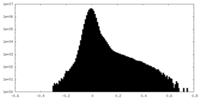
| 投影像・断面図 |
| ||||||||||||
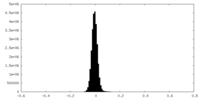
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_28580_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
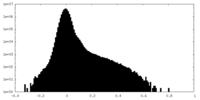
| 投影像・断面図 |
| ||||||||||||
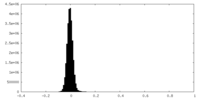
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : ADAM10-Tspan15-11G2 Fab complex
+超分子 #1: ADAM10-Tspan15-11G2 Fab complex
+超分子 #2: Disintegrin and metalloproteinase domain-containing protein 10 (E...
+超分子 #3: 11G2 Fab Heavy Chain, 11G2 Fab Light Chain
+分子 #1: Disintegrin and metalloproteinase domain-containing protein 10
+分子 #2: Tetraspanin-15
+分子 #3: 11G2 Fab Heavy Chain
+分子 #4: 11G2 Fab Light Chain
+分子 #7: 2-acetamido-2-deoxy-beta-D-glucopyranose
+分子 #8: CALCIUM ION
+分子 #9: ZINC ION
+分子 #10: 4-(N-HYDROXYAMINO)-2R-ISOBUTYL-2S-(2-THIENYLTHIOMETHYL)SUCCINYL-L...
+分子 #11: CHOLESTEROL HEMISUCCINATE
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.1 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
| |||||||||||||||
| グリッド | モデル: Quantifoil / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV / 詳細: Blot for 7 seconds with a blot force of 15. | |||||||||||||||
| 詳細 | This sample was monodisperse |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 10037 / 平均露光時間: 1.3 sec. / 平均電子線量: 51.99 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: PDB ENTRY PDBモデル - PDB ID: 詳細: PDB 6BE6 was used for the initial model of ADAM10, 6BDZ was used for 11G2 vFab |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 3.3 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC (ver. 3.3.1) / 使用した粒子像数: 178031 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: cryoSPARC (ver. 3.3.1) |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: cryoSPARC (ver. 3.3.1) |
-原子モデル構築 1
| 精密化 | 空間: REAL |
|---|---|
| 得られたモデル |  PDB-8esv: |
 ムービー
ムービー コントローラー
コントローラー
















 Z
Z Y
Y X
X