+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of 337 Fab in complex with recombinant shortened Plasmodium falciparum circumsporozoite protein (rsCSP) | |||||||||
 マップデータ マップデータ | Cryo-EM structure of 337 Fab in complex with recombinant shortened Plasmodium falciparum circumsporozoite protein (rsCSP) | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | malaria antibody / PfCSP / IMMUNE SYSTEM | |||||||||
| 生物種 |   Homo sapiens (ヒト) / Homo sapiens (ヒト) /  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.7 Å | |||||||||
 データ登録者 データ登録者 | Martin GM / Ward AB | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Affinity-matured homotypic interactions induce spectrum of PfCSP structures that influence protection from malaria infection. 著者: Gregory M Martin / Jonathan L Torres / Tossapol Pholcharee / David Oyen / Yevel Flores-Garcia / Grace Gibson / Re'em Moskovitz / Nathan Beutler / Diana D Jung / Jeffrey Copps / Wen-Hsin Lee / ...著者: Gregory M Martin / Jonathan L Torres / Tossapol Pholcharee / David Oyen / Yevel Flores-Garcia / Grace Gibson / Re'em Moskovitz / Nathan Beutler / Diana D Jung / Jeffrey Copps / Wen-Hsin Lee / Gonzalo Gonzalez-Paez / Daniel Emerling / Randall S MacGill / Emily Locke / C Richter King / Fidel Zavala / Ian A Wilson / Andrew B Ward /   要旨: The generation of high-quality antibody responses to Plasmodium falciparum (Pf) circumsporozoite protein (PfCSP), the primary surface antigen of Pf sporozoites, is paramount to the development of an ...The generation of high-quality antibody responses to Plasmodium falciparum (Pf) circumsporozoite protein (PfCSP), the primary surface antigen of Pf sporozoites, is paramount to the development of an effective malaria vaccine. Here we present an in-depth structural and functional analysis of a panel of potent antibodies encoded by the immunoglobulin heavy chain variable (IGHV) gene IGHV3-33, which is among the most prevalent and potent antibody families induced in the anti-PfCSP immune response and targets the Asn-Ala-Asn-Pro (NANP) repeat region. Cryo-electron microscopy (cryo-EM) reveals a remarkable spectrum of helical antibody-PfCSP structures stabilized by homotypic interactions between tightly packed fragments antigen binding (Fabs), many of which correlate with somatic hypermutation. We demonstrate a key role of these mutated homotypic contacts for high avidity binding to PfCSP and in protection from Pf malaria infection. Together, these data emphasize the importance of anti-homotypic affinity maturation in the frequent selection of IGHV3-33 antibodies and highlight key features underlying the potent protection of this antibody family. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27787.map.gz emd_27787.map.gz | 37.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27787-v30.xml emd-27787-v30.xml emd-27787.xml emd-27787.xml | 17.8 KB 17.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_27787.png emd_27787.png | 162.4 KB | ||
| Filedesc metadata |  emd-27787.cif.gz emd-27787.cif.gz | 5.8 KB | ||
| その他 |  emd_27787_half_map_1.map.gz emd_27787_half_map_1.map.gz emd_27787_half_map_2.map.gz emd_27787_half_map_2.map.gz | 37.1 MB 37.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27787 http://ftp.pdbj.org/pub/emdb/structures/EMD-27787 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27787 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27787 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_27787_validation.pdf.gz emd_27787_validation.pdf.gz | 905.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_27787_full_validation.pdf.gz emd_27787_full_validation.pdf.gz | 904.7 KB | 表示 | |
| XML形式データ |  emd_27787_validation.xml.gz emd_27787_validation.xml.gz | 11.4 KB | 表示 | |
| CIF形式データ |  emd_27787_validation.cif.gz emd_27787_validation.cif.gz | 13.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27787 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27787 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27787 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27787 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27787.map.gz / 形式: CCP4 / 大きさ: 40.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27787.map.gz / 形式: CCP4 / 大きさ: 40.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of 337 Fab in complex with recombinant shortened Plasmodium falciparum circumsporozoite protein (rsCSP) | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.026 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Cryo-EM structure of 337 Fab in complex with...
| ファイル | emd_27787_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of 337 Fab in complex with recombinant shortened Plasmodium falciparum circumsporozoite protein (rsCSP) | ||||||||||||
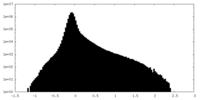
| 投影像・断面図 |
| ||||||||||||
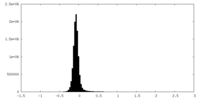
| 密度ヒストグラム |
-ハーフマップ: Cryo-EM structure of 337 Fab in complex with...
| ファイル | emd_27787_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of 337 Fab in complex with recombinant shortened Plasmodium falciparum circumsporozoite protein (rsCSP) | ||||||||||||
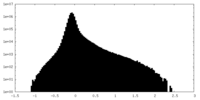
| 投影像・断面図 |
| ||||||||||||
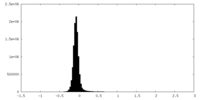
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : 337 Fab - rsCSP complex
| 全体 | 名称: 337 Fab - rsCSP complex |
|---|---|
| 要素 |
|
-超分子 #1: 337 Fab - rsCSP complex
| 超分子 | 名称: 337 Fab - rsCSP complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|
-超分子 #2: Circumsporozoite protein
| 超分子 | 名称: Circumsporozoite protein / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #3: 337 Fab heavy chain, 337 Fab light chain
| 超分子 | 名称: 337 Fab heavy chain, 337 Fab light chain / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2-#3 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Circumsporozoite protein
| 分子 | 名称: Circumsporozoite protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 30.232156 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: YGSSSNTRVL NELNYDNAGT NLYNELEMNY YGKQENWYSL KKNSRSLGEN DDGNNEDNEK LRKPKHKKLK QPADGNPDPN ANPNVDPNA NPNVDPNANP NVDPNANPNA NPNANPNANP NANPNANPNA NPNANPNANP NANPNANPNA NPNANPNANP N ANPNANPN ...文字列: YGSSSNTRVL NELNYDNAGT NLYNELEMNY YGKQENWYSL KKNSRSLGEN DDGNNEDNEK LRKPKHKKLK QPADGNPDPN ANPNVDPNA NPNVDPNANP NVDPNANPNA NPNANPNANP NANPNANPNA NPNANPNANP NANPNANPNA NPNANPNANP N ANPNANPN KNNQGNGQGH NMPNDPNRNV DENANANSAV KNNNNEEPSD KHIKEYLNKI QNSLSTEWSP CSVTCGNGIQ VR IKPGSAN KPKDELDYAN DIEKKICKME KCSSVFNVVN S |
-分子 #2: 337 Fab heavy chain
| 分子 | 名称: 337 Fab heavy chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 24.31923 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: QVQLVESGGG VVQPGRSLRL SCAASGFSFS TYGMHWVRQA PGKGLEWVAL IWHDGSKKFY SESVRGRFTI SRDNSMDTLY LQMSSLRAE DTAVYYCARD GADYFDSTGG AFDIWGQGTM VIVSSASTKG PSVFPLAPSS KSTSGGTAAL GCLVKDYFPE P VTVSWNSG ...文字列: QVQLVESGGG VVQPGRSLRL SCAASGFSFS TYGMHWVRQA PGKGLEWVAL IWHDGSKKFY SESVRGRFTI SRDNSMDTLY LQMSSLRAE DTAVYYCARD GADYFDSTGG AFDIWGQGTM VIVSSASTKG PSVFPLAPSS KSTSGGTAAL GCLVKDYFPE P VTVSWNSG ALTSGVHTFP AVLQSSGLYS LSSVVTVPSS SLGTQTYICN VNHKPSNTKV DKKVEPKSCD |
-分子 #3: 337 Fab light chain
| 分子 | 名称: 337 Fab light chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.321887 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: EIVMTQSPAT LSVSPGERAT LSCRASQSVS SNLAWYQQKP GQAPRLLIYG ASTRATGIPA RFSGSGSGTE FTLTISSLQS EDFATYYCQ QYKTWWTFGQ GTKVEIKRTV AAPSVFIFPP SDEQLKSGTA SVVCLLNNFY PREAKVQWKV DNALQSGNSQ E SVTEQDSK ...文字列: EIVMTQSPAT LSVSPGERAT LSCRASQSVS SNLAWYQQKP GQAPRLLIYG ASTRATGIPA RFSGSGSGTE FTLTISSLQS EDFATYYCQ QYKTWWTFGQ GTKVEIKRTV AAPSVFIFPP SDEQLKSGTA SVVCLLNNFY PREAKVQWKV DNALQSGNSQ E SVTEQDSK DSTYSLSSTL TLSKADYEKH KVYACEVTHQ GLSSPVTKSF NRGEC |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 2.7 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 461179 |
| 初期 角度割当 | タイプ: PROJECTION MATCHING |
| 最終 角度割当 | タイプ: PROJECTION MATCHING |
 ムービー
ムービー コントローラー
コントローラー


















 Z
Z Y
Y X
X