+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Importin-4 bound to RanGTP | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | Importin / Karyopherin / GTPase / nuclear import / PROTEIN TRANSPORT | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of cell cycle phase transition / Postmitotic nuclear pore complex (NPC) reformation / regulation of nucleocytoplasmic transport / exonucleolytic trimming to generate mature 3'-end of 5.8S rRNA from tricistronic rRNA transcript (SSU-rRNA, 5.8S rRNA, LSU-rRNA) / nuclear import signal receptor activity / poly(A)+ mRNA export from nucleus / nuclear localization sequence binding / nucleus organization / protein localization to nucleus / ribosomal subunit export from nucleus ...regulation of cell cycle phase transition / Postmitotic nuclear pore complex (NPC) reformation / regulation of nucleocytoplasmic transport / exonucleolytic trimming to generate mature 3'-end of 5.8S rRNA from tricistronic rRNA transcript (SSU-rRNA, 5.8S rRNA, LSU-rRNA) / nuclear import signal receptor activity / poly(A)+ mRNA export from nucleus / nuclear localization sequence binding / nucleus organization / protein localization to nucleus / ribosomal subunit export from nucleus / small GTPase binding / protein import into nucleus / GTPase activity / chromatin / GTP binding / protein-containing complex / membrane / nucleus / cytoplasm / cytosol 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 7.1 Å | ||||||||||||
 データ登録者 データ登録者 | Bernardes NE / Fung HYJ / Li Y / Chen Z / Chook YM | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2022 ジャーナル: Proc Natl Acad Sci U S A / 年: 2022タイトル: Structure of IMPORTIN-4 bound to the H3-H4-ASF1 histone-histone chaperone complex. 著者: Natália Elisa Bernardes / Ho Yee Joyce Fung / Yang Li / Zhe Chen / Yuh Min Chook /  要旨: IMPORTIN-4, the primary nuclear import receptor of core histones H3 and H4, binds the H3-H4 dimer and histone chaperone ASF1 prior to nuclear import. However, how H3-H3-ASF1 is recognized for ...IMPORTIN-4, the primary nuclear import receptor of core histones H3 and H4, binds the H3-H4 dimer and histone chaperone ASF1 prior to nuclear import. However, how H3-H3-ASF1 is recognized for transport cannot be explained by available crystal structures of IMPORTIN-4-histone tail peptide complexes. Our 3.5-Å IMPORTIN-4-H3-H4-ASF1 cryoelectron microscopy structure reveals the full nuclear import complex and shows a binding mode different from suggested by previous structures. The N-terminal half of IMPORTIN-4 clamps the globular H3-H4 domain and H3 αN helix, while its C-terminal half binds the H3 N-terminal tail weakly; tail contribution to binding energy is negligible. ASF1 binds H3-H4 without contacting IMPORTIN-4. Together, ASF1 and IMPORTIN-4 shield nucleosomal H3-H4 surfaces to chaperone and import it into the nucleus where RanGTP binds IMPORTIN-4, causing large conformational changes to release H3-H4-ASF1. This work explains how full-length H3-H4 binds IMPORTIN-4 in the cytoplasm and how it is released in the nucleus. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27780.map.gz emd_27780.map.gz | 30.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27780-v30.xml emd-27780-v30.xml emd-27780.xml emd-27780.xml | 17.3 KB 17.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_27780.png emd_27780.png | 76.2 KB | ||
| Filedesc metadata |  emd-27780.cif.gz emd-27780.cif.gz | 6.3 KB | ||
| その他 |  emd_27780_half_map_1.map.gz emd_27780_half_map_1.map.gz emd_27780_half_map_2.map.gz emd_27780_half_map_2.map.gz | 55.2 MB 55.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27780 http://ftp.pdbj.org/pub/emdb/structures/EMD-27780 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27780 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27780 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_27780_validation.pdf.gz emd_27780_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_27780_full_validation.pdf.gz emd_27780_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_27780_validation.xml.gz emd_27780_validation.xml.gz | 12.1 KB | 表示 | |
| CIF形式データ |  emd_27780_validation.cif.gz emd_27780_validation.cif.gz | 14.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27780 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27780 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27780 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27780 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8dyoMC  7unkC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27780.map.gz / 形式: CCP4 / 大きさ: 59.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27780.map.gz / 形式: CCP4 / 大きさ: 59.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.094 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
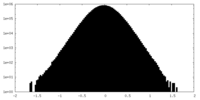
-ハーフマップ: #2
| ファイル | emd_27780_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
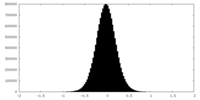
-ハーフマップ: #1
| ファイル | emd_27780_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of Importin-4 bound to RanGTP
| 全体 | 名称: Complex of Importin-4 bound to RanGTP |
|---|---|
| 要素 |
|
-超分子 #1: Complex of Importin-4 bound to RanGTP
| 超分子 | 名称: Complex of Importin-4 bound to RanGTP / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Importin-4
| 分子 | 名称: Importin-4 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 118.83207 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MESAGLEQLL RELLLPDTER IRRATEQLQI VLRAPAALPA LCDLLASAAD PQIRQFAAVL TRRRLNTRWR RLAAEQRESL KSLILTALQ RETEHCVSLS LAQLSATIFR KEGLEAWPQL LQLLQHSTHS PHSPEREMGL LLLSVVVTSR PEAFQPHHRE L LRLLNETL ...文字列: MESAGLEQLL RELLLPDTER IRRATEQLQI VLRAPAALPA LCDLLASAAD PQIRQFAAVL TRRRLNTRWR RLAAEQRESL KSLILTALQ RETEHCVSLS LAQLSATIFR KEGLEAWPQL LQLLQHSTHS PHSPEREMGL LLLSVVVTSR PEAFQPHHRE L LRLLNETL GEVGSPGLLF YSLRTLTTMA PYLSTEDVPL ARMLVPKLIM AMQTLIPIDE AKACEALEAL DELLESEVPV IT PYLSEVL TFCLEVARNV ALGNAIRIRI LCCLTFLVKV KSKALLKNRL LPPLLHTLFP IVAAEPPPGQ LDPEDQDSEE EEL EIELMG ETPKHFAVQV VDMLALHLPP EKLCPQLMPM LEEALRSESP YQRKAGLLVL AVLSDGAGDH IRQRLLPPLL QIVC KGLED PSQVVRNAAL FALGQFSENL QPHISSYSRE VMPLLLAYLK SVPLGHTHHL AKACYALENF VENLGPKVQP YLPEL MECM LQLLRNPSSP RAKELAVSAL GAIATAAQAS LLPYFPAIME HLREFLLTGR EDLQPVQIQS LETLGVLARA VGEPMR PLA EECCQLGLGL CDQVDDPDLR RCTYSLFAAL SGLMGEGLAP HLEQITTLML LSLRSTEGIV PQYDGSSSFL LFDDESD GE EEEELMDEDV EEEDDSEISG YSVENAFFDE KEDTCAAVGE ISVNTSVAFL PYMESVFEEV FKLLECPHLN VRKAAHEA L GQFCCALHKA CQSCPSEPNT AALQAALARV VPSYMQAVNR ERERQVVMAV LEALTGVLRS CGTLTLKPPG RLAELCGVL KAVLQRKTAC QDTDEEEEEE DDDQAEYDAM LLEHAGEAIP ALAAAAGGDS FAPFFAGFLP LLVCKTKQGC TVAEKSFAVG TLAETIQGL GAASAQFVSR LLPVLLSTAQ EADPEVRSNA IFGMGVLAEH GGHPAQEHFP KLLGLLFPLL ARERHDRVRD N ICGALARL LMASPTRKPE PQVLAALLHA LPLKEDLEEW VTIGRLFSFL YQSSPDQVID VAPELLRICS LILADNKIPP DT KAALLLL LTFLAKQHTD SFQAALGSLP VDKAQELQAV LGLS UniProtKB: Importin-4 |
-分子 #2: GTP-binding nuclear protein GSP1/CNR1
| 分子 | 名称: GTP-binding nuclear protein GSP1/CNR1 / タイプ: protein_or_peptide / ID: 2 詳細: Truncated yeast Ran (residues 1-179) with the Q71L mutation コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 24.825357 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSAPAANGEV PTFKLVLVGD GGTGKTTFVK RHLTGEFEKK YIATIGVEVH PLSFYTNFGE IKFDVWDTAG LEKFGGLRDG YYINAQCAI IMFDVTSRIT YKNVPNWHRD LVRVCENIPI VLCGNKVDVK ERKVKAKTIT FHRKKNLQYY DISAKSNYNF E KPFLWLAR ...文字列: MSAPAANGEV PTFKLVLVGD GGTGKTTFVK RHLTGEFEKK YIATIGVEVH PLSFYTNFGE IKFDVWDTAG LEKFGGLRDG YYINAQCAI IMFDVTSRIT YKNVPNWHRD LVRVCENIPI VLCGNKVDVK ERKVKAKTIT FHRKKNLQYY DISAKSNYNF E KPFLWLAR KLAGNPQLEF VASPALAPPE VQVDEQLMQQ YQQEMEQATA LPLPDEDDAD L UniProtKB: GTP-binding nuclear protein GSP1/CNR1 |
-分子 #3: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 3 / コピー数: 1 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #4: GUANOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: GUANOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 4 / コピー数: 1 / 式: GTP |
|---|---|
| 分子量 | 理論値: 523.18 Da |
| Chemical component information |  ChemComp-GTP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.6 µm 最小 デフォーカス(公称値): 1.4000000000000001 µm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 7.1 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 16089 |
| 初期 角度割当 | タイプ: RANDOM ASSIGNMENT / ソフトウェア - 名称: cryoSPARC (ver. 3.3.1) |
| 最終 角度割当 | タイプ: OTHER / ソフトウェア - 名称: cryoSPARC (ver. 3.3.1) |
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-8dyo: |
 ムービー
ムービー コントローラー
コントローラー







 Z
Z Y
Y X
X