+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Avs3 bound to phage PhiV-1 terminase, C2 refinement of Cap4 nuclease domain | ||||||||||||
 マップデータ マップデータ | Avs3 bound to gp19 terminase, C2 refinement of Cap4 nuclease domain | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | phage defense / pattern-recognition receptor / nlr / stand / atpase / ANTIVIRAL PROTEIN | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報viral terminase, large subunit / viral DNA genome packaging / 加水分解酵素; エステル加水分解酵素; 5'-リン酸モノエステル産生エンドデオキシリボヌクレアーゼ / chromosome organization / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / endonuclease activity / ATP hydrolysis activity / ATP binding / metal ion binding 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Salmonella enterica (サルモネラ菌) / Salmonella enterica (サルモネラ菌) /  Escherichia phage PhiV-1 (ヒト免疫不全ウイルス) Escherichia phage PhiV-1 (ヒト免疫不全ウイルス) | ||||||||||||
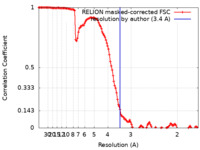
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | ||||||||||||
 データ登録者 データ登録者 | Wilkinson ME / Gao L / Strecker J / Makarova KS / Macrae RK / Koonin EV / Zhang F | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Science / 年: 2022 ジャーナル: Science / 年: 2022タイトル: Prokaryotic innate immunity through pattern recognition of conserved viral proteins. 著者: Linyi Alex Gao / Max E Wilkinson / Jonathan Strecker / Kira S Makarova / Rhiannon K Macrae / Eugene V Koonin / Feng Zhang /  要旨: Many organisms have evolved specialized immune pattern-recognition receptors, including nucleotide-binding oligomerization domain-like receptors (NLRs) of the STAND superfamily that are ubiquitous in ...Many organisms have evolved specialized immune pattern-recognition receptors, including nucleotide-binding oligomerization domain-like receptors (NLRs) of the STAND superfamily that are ubiquitous in plants, animals, and fungi. Although the roles of NLRs in eukaryotic immunity are well established, it is unknown whether prokaryotes use similar defense mechanisms. Here, we show that antiviral STAND (Avs) homologs in bacteria and archaea detect hallmark viral proteins, triggering Avs tetramerization and the activation of diverse N-terminal effector domains, including DNA endonucleases, to abrogate infection. Cryo-electron microscopy reveals that Avs sensor domains recognize conserved folds, active-site residues, and enzyme ligands, allowing a single Avs receptor to detect a wide variety of viruses. These findings extend the paradigm of pattern recognition of pathogen-specific proteins across all three domains of life. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27421.map.gz emd_27421.map.gz | 166 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27421-v30.xml emd-27421-v30.xml emd-27421.xml emd-27421.xml | 22.7 KB 22.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_27421_fsc.xml emd_27421_fsc.xml | 12.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_27421.png emd_27421.png | 92.2 KB | ||
| マスクデータ |  emd_27421_msk_1.map emd_27421_msk_1.map | 178 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-27421.cif.gz emd-27421.cif.gz | 8.1 KB | ||
| その他 |  emd_27421_half_map_1.map.gz emd_27421_half_map_1.map.gz emd_27421_half_map_2.map.gz emd_27421_half_map_2.map.gz | 139.1 MB 139.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27421 http://ftp.pdbj.org/pub/emdb/structures/EMD-27421 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27421 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27421 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_27421_validation.pdf.gz emd_27421_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_27421_full_validation.pdf.gz emd_27421_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_27421_validation.xml.gz emd_27421_validation.xml.gz | 20 KB | 表示 | |
| CIF形式データ |  emd_27421_validation.cif.gz emd_27421_validation.cif.gz | 26.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27421 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27421 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27421 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27421 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8dgcMC  8dgfC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27421.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27421.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Avs3 bound to gp19 terminase, C2 refinement of Cap4 nuclease domain | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8697 Å | ||||||||||||||||||||||||||||||||||||
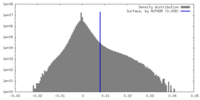
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_27421_msk_1.map emd_27421_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 1
| ファイル | emd_27421_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 2
| ファイル | emd_27421_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Avs3 bound to phage PhiV-1 terminase
| 全体 | 名称: Avs3 bound to phage PhiV-1 terminase |
|---|---|
| 要素 |
|
-超分子 #1: Avs3 bound to phage PhiV-1 terminase
| 超分子 | 名称: Avs3 bound to phage PhiV-1 terminase / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 分子量 | 理論値: 1.21 MDa |
-分子 #1: SeAvs3
| 分子 | 名称: SeAvs3 / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Salmonella enterica (サルモネラ菌) Salmonella enterica (サルモネラ菌) |
| 分子量 | 理論値: 236.796469 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSDSLLVRTS RDGDQFHYLW AARRALRLLE PQSTLVALTI EGASTTEMGS QPVVEDGEEL IDIAEYYGSN ELATATTVRY MQLKHSTMH SDTPFPPSGL QKTIEGFATR YKALIQKIPV ETLRTKLEFW FVTNRPVSSS FSEAINDAAN QHVTRHPHDL A KLEKFTGL ...文字列: MSDSLLVRTS RDGDQFHYLW AARRALRLLE PQSTLVALTI EGASTTEMGS QPVVEDGEEL IDIAEYYGSN ELATATTVRY MQLKHSTMH SDTPFPPSGL QKTIEGFATR YKALIQKIPV ETLRTKLEFW FVTNRPVSSS FSEAINDAAN QHVTRHPHDL A KLEKFTGL QGAELSIFCQ LLHIEGQQDD LWSQRNILLR ESAGYLPDLD TEAPLKLKEL VNRKALTESA ANPSITRMDV LR ALGVDET DLFPAPCRIE RIENSVSRTQ EATLVQRVVE AFGAPVIIHA DAGVGKSIFS THIEEHLPTG SVSILYDCFG LGQ YRNASS YRHHHRTALV QMANEMASRG LCHPLIPNAG TGISQYMRAF LHRLSQSISI LRASEPLAVL CIIIDAADNA QMAA EEIGE TRSFIKDLIR EKLPDGVCLV ALCRPYRREL LDPPPEALTL SLQTFNRDET AAHLHQKFPD ASESDVDEFH RLSSC NPRV QALSLSQNLP LNDTLRLLGP NPKTVEDTIG EVLEKSIARL RDTAGISERA QIDTICSALA ILRPLIPLSV LSAISG VAG SAIKSFALDL GRPLIVSGET IQFFDEPAET WFQRRFRPSA ADLHQFITKL RPLTKDSSYA ASVLPALMLE GNQLSEL IE LAISSQALPE TSAVERRDIE LQRLQFALKA ALRTGRYQDA AKLALKAGGE CAGDNRQRVL LRDNIDLAAK FVGSNGVQ E LVSRNAFPDT GWPGSRNAYY AAILSEYPEL SGEARSRLRL TMEWLTNWSQ LPDDERSRQN VTDQDRAVML IACLNIHGA EAAARELRRW RPRKLSFDAG KIVAMQLLAH ARYDELDQLA IAAGNDISLV MGIVLEARKL HRPVAEQAIR RTWRLLKSQR VSIKDRNHA NNQTIAAITG MVEMALIQSV CTESESIQLL DRYLPKVPPY ALTSEYSKER VAYVRAYALQ ANLMGSQLAL S DLASTEVK KELMAEKRHG ESDDLRQLKQ YSGVLIPWYN LWAKVILGKT RKADLESELS DTQKESTAIK GHSYSEHSLS SN EIANVWF DILIEAGNVS KDDVENIIKW SQHKGNRVFT PTLHRFSSVC AEISGLGELS YHFAELALSL WRDEHSDAQI KAD GYIDLS RSLISLDEPE AKEYFNQAIE VTNKLGDENL SRWEAILDLA EYVAGKTQVP PEISYKLARC AELTREYVDR DKHF AWSDT VEILAELCPS SALAIISRWR DRTFGNHRSI LAWTIEHLVK KNKINALDAL PLITFENDWH KCDLLDSVLS SCTDD KDKI MAFEVVYHYT KFNVQNIQNL KKLDAISTSL GIEHTELKER ISGLQHTETV SKKSSLSSND NEQGHDQEWE SIFKDC DLS SIDGISAAYE KFRNVPEFYS KETFIKKAIS RVKTGKECSF ITAIGAIFHW GLYDFKYILE SIPDEWTSRL SIKTTLA GL IKEYCQRFCM RIRKSRVYEI FPFSLASRLS GISEKEIFGI TLEAIAESPE PANSDRLFSL PGLLVSKLES NEALDVLS Y ALDLFDEVLK DEDGDGPWNE KLSPPTHVED SLAGYIWARL GSPEAEMRWQ AAHAVLALCR MSRTCVIQGI FQHAINATT LPFCDRNLPF YTLHAQLWLM IAAARVALDD GKSLIPNIGY FYHYATTDQP HVLIRHFAAR TLLALHDSDL ISIPAQEENK LRNINQSTT LPVLDKVEDH RGEDSYTFGI DFGPYWLKPL GRCFGVSQKQ LEPEMLRIIR DVLGFKGSRN WDEDERNKRR Y YQDRDNHH SHGSYPRVDD YHFYLSYHAM FMTAGQLLAT KPLVGSDYDD VEDVFQDWLR RHDISRNDHR WLADRRDIPP KE RSSWLNS SSDNRDEWLA SISENVFNET LCPSPGLLTL WGRWSDVCSD RKESIIVHSA LVSPERSLSL LRALQTTKNV YDY KIPDAG DNLEIDHAHY QLKGWIKDIA EYCGIDEFDP WAGNVRFPIP EPASFIIDAM KLTTDKDHRV WYSPSDVEPA MISS IWGHL SGKNDEEKSH GYRLCASIHF IKSALETFNM DLILEVDVDR YSRNSRYERN NENELDNIPS STRLFLFRHD GTIHT LYGN YRNGEKTS |
-分子 #2: Terminase, large subunit
| 分子 | 名称: Terminase, large subunit / タイプ: protein_or_peptide / ID: 2 / コピー数: 4 / 光学異性体: LEVO EC番号: 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 |
|---|---|
| 由来(天然) | 生物種:  Escherichia phage PhiV-1 (ヒト免疫不全ウイルス) Escherichia phage PhiV-1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 66.345469 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSQSQEAKNA LIIAQLKGDF VAFLFVLWKA LNLPKPTKCQ IDMARTLANG DHKKFILQAF RGIGKSFITC AFVVWVLWRD PQLKVLIVS ASKERADANS IFIKNIIDLL PFLSELKPRP GQRDSVISFD VGLAKPDHSP SVKSVGITGQ LTGSRADIII A DDVEVPGN ...文字列: MSQSQEAKNA LIIAQLKGDF VAFLFVLWKA LNLPKPTKCQ IDMARTLANG DHKKFILQAF RGIGKSFITC AFVVWVLWRD PQLKVLIVS ASKERADANS IFIKNIIDLL PFLSELKPRP GQRDSVISFD VGLAKPDHSP SVKSVGITGQ LTGSRADIII A DDVEVPGN SSTSSAREKL WTLVTEFAAL LKPLPTSRVI YLGTPQTEMT LYKELEDNKG YSTVIWPAQY PRNDAEALYY GD RLAPMLK AEYDEGFELL RGQPTDPVRF DMDDLREREL EYGKAGYTLQ FMLNPNLSDA EKYPLRLRDA IVCAVDPERA PLS YQWLPN RQNRNEELPN VGLKGDDIHA FHTCSSRTAE YQSKILVIDP SGRGKDETGY AVLYSLNGYI YLMEVGGFRG GYDD ATLEK LAKKAKQWKV QTVVHESNFG DGMFGKIFSP ILLKHHKCAL EEIRAKGMKE MRICDTIEPL MGAHKLVIRD EVIRE DYQT ARDLDGKHDV RYSAFYQMTR MTRERGAVAH DDRIDAIALG IEYLREGMLV DSRVGEEEMT LEFLEHHMEK QTIGGD QIH SFDVGGVDIY YEDEDGGSSF IEW UniProtKB: Terminase, large subunit |
-分子 #3: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 3 / コピー数: 8 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-分子 #4: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 4 / コピー数: 8 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 30.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)