+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | AT from first module of the pikromycin synthase | |||||||||
 マップデータ マップデータ | Local refinement map for one PikAT1 monomer from the engineered synthase Pik127. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | acyltransferase / methylmalonyl-specific / polyketide synthase / BIOSYNTHETIC PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報10-deoxymethynolide synthase / narbonolide synthase / macrolide biosynthetic process / DIM/DIP cell wall layer assembly / fatty acid synthase activity / acyltransferase activity, transferring groups other than amino-acyl groups / phosphopantetheine binding / 3-oxoacyl-[acyl-carrier-protein] synthase activity / fatty acid biosynthetic process / plasma membrane / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Streptomyces venezuelae (バクテリア) Streptomyces venezuelae (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.86 Å | |||||||||
 データ登録者 データ登録者 | Keatinge-Clay AT / Dickinson MS / Miyazawa T / McCool RS | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2022 ジャーナル: Structure / 年: 2022タイトル: Priming enzymes from the pikromycin synthase reveal how assembly-line ketosynthases catalyze carbon-carbon chemistry. 著者: Miles S Dickinson / Takeshi Miyazawa / Ryan S McCool / Adrian T Keatinge-Clay /  要旨: The first domain of modular polyketide synthases (PKSs) is most commonly a ketosynthase (KS)-like enzyme, KS, that primes polyketide synthesis. Unlike downstream KSs that fuse α-carboxyacyl groups ...The first domain of modular polyketide synthases (PKSs) is most commonly a ketosynthase (KS)-like enzyme, KS, that primes polyketide synthesis. Unlike downstream KSs that fuse α-carboxyacyl groups to growing polyketide chains, it performs an extension-decoupled decarboxylation of these groups to generate primer units. When Pik127, a model triketide synthase constructed from modules of the pikromycin synthase, was studied by cryoelectron microscopy (cryo-EM), the dimeric didomain comprised of KS and the neighboring methylmalonyl-selective acyltransferase (AT) dominated the class averages and yielded structures at 2.5- and 2.8-Å resolution, respectively. Comparisons with ketosynthases complexed with their substrates revealed the conformation of the (2S)-methylmalonyl-S-phosphopantetheinyl portion of KS and KS substrates prior to decarboxylation. Point mutants of Pik127 probed the roles of residues in the KS active site, while an AT-swapped version of Pik127 demonstrated that KS can also decarboxylate malonyl groups. Mechanisms for how KS and KS domains catalyze carbon-carbon chemistry are proposed. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27094.map.gz emd_27094.map.gz | 1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27094-v30.xml emd-27094-v30.xml emd-27094.xml emd-27094.xml | 20.5 KB 20.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_27094_fsc.xml emd_27094_fsc.xml | 10.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_27094.png emd_27094.png | 108.2 KB | ||
| Filedesc metadata |  emd-27094.cif.gz emd-27094.cif.gz | 6.7 KB | ||
| その他 |  emd_27094_half_map_1.map.gz emd_27094_half_map_1.map.gz emd_27094_half_map_2.map.gz emd_27094_half_map_2.map.gz | 95.5 MB 95.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27094 http://ftp.pdbj.org/pub/emdb/structures/EMD-27094 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27094 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27094 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_27094_validation.pdf.gz emd_27094_validation.pdf.gz | 838.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_27094_full_validation.pdf.gz emd_27094_full_validation.pdf.gz | 838 KB | 表示 | |
| XML形式データ |  emd_27094_validation.xml.gz emd_27094_validation.xml.gz | 16.5 KB | 表示 | |
| CIF形式データ |  emd_27094_validation.cif.gz emd_27094_validation.cif.gz | 20.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27094 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27094 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27094 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27094 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8czcMC  7uwrC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27094.map.gz / 形式: CCP4 / 大きさ: 1.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27094.map.gz / 形式: CCP4 / 大きさ: 1.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Local refinement map for one PikAT1 monomer from the engineered synthase Pik127. | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.81 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: First half map
| ファイル | emd_27094_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | First half map | ||||||||||||
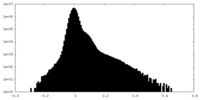
| 投影像・断面図 |
| ||||||||||||
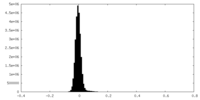
| 密度ヒストグラム |
-ハーフマップ: Second half map
| ファイル | emd_27094_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Second half map | ||||||||||||
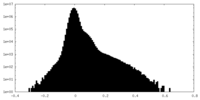
| 投影像・断面図 |
| ||||||||||||
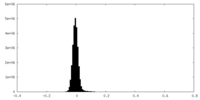
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Pik127
| 全体 | 名称: Pik127 |
|---|---|
| 要素 |
|
-超分子 #1: Pik127
| 超分子 | 名称: Pik127 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Polyketide synthase engineered from the first, second, and seventh modules of the pikromycin synthase as described in Miyazawa et al., 2021, Chem. Commun. 57:8762-8765 |
|---|---|
| 由来(天然) | 生物種:  Streptomyces venezuelae (バクテリア) / 株: 15439 Streptomyces venezuelae (バクテリア) / 株: 15439 |
| 分子量 | 理論値: 410 KDa |
-分子 #1: Narbonolide/10-deoxymethynolide synthase PikA1, modules 1 and 2
| 分子 | 名称: Narbonolide/10-deoxymethynolide synthase PikA1, modules 1 and 2 タイプ: protein_or_peptide / ID: 1 / 詳細: First AT domain of pikromycin synthase / コピー数: 1 / 光学異性体: LEVO / EC番号: 10-deoxymethynolide synthase |
|---|---|
| 由来(天然) | 生物種:  Streptomyces venezuelae (バクテリア) Streptomyces venezuelae (バクテリア) |
| 分子量 | 理論値: 33.011234 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GVGRVAFVFP GQGTQWAGMG AELLDSSAVF AAAMAECEAA LSPYVDWSLE AVVRQAPGAP TLERVDVVQP VTFAVMVSLA RVWQHHGVT PQAVVGHSQG EIAAAYVAGA LSLDDAARVV TLRSKSIAAH LAGKGGMLSL ALSEDAVLER LAGFDGLSVA A VNGPTATV ...文字列: GVGRVAFVFP GQGTQWAGMG AELLDSSAVF AAAMAECEAA LSPYVDWSLE AVVRQAPGAP TLERVDVVQP VTFAVMVSLA RVWQHHGVT PQAVVGHSQG EIAAAYVAGA LSLDDAARVV TLRSKSIAAH LAGKGGMLSL ALSEDAVLER LAGFDGLSVA A VNGPTATV VSGDPVQIEE LARACEADGV RARVIPVDYA SHSRQVEIIE SELAEVLAGL SPQAPRVPFF STLEGAWITE PV LDGGYWY RNLRHRVGFA PAVETLATDE GFTHFVEVSA HPVLTMALPG TVTGLATLRR DNGGQDRLVA SLAEAWAN UniProtKB: Narbonolide/10-deoxymethynolide synthase PikA1, modules 1 and 2 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.5 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
詳細: Buffer was made fresh because TCEP rapidly degrades in phosphate | |||||||||||||||
| グリッド | モデル: C-flat-2/2 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 12 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 30 sec. | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV 詳細: Blotted for 3 seconds with a force of "1" before plunging.. | |||||||||||||||
| 詳細 | This sample was monodisperse. It was incubated with its substrates, methylmalonyl-CoA and NADPH, for 30 min. at 25 C before freezing. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 詳細 | Collected 4284 movies at 0 degrees tilt and 2912 movies at -30 degrees tilt |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 7196 / 平均露光時間: 5.0 sec. / 平均電子線量: 70.0 e/Å2 / 詳細: 20 frames per second |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 倍率(補正後): 61728 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー








 Z
Z Y
Y X
X