+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
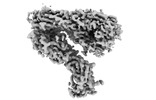
| タイトル | Integrin alphaM/beta2 ectodomain | |||||||||
 マップデータ マップデータ | Composite map of sharpened global and local reconstructions | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Integrin / Complement / Receptor / CELL ADHESION | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報ectodermal cell differentiation / positive regulation of neutrophil degranulation / cellular extravasation / integrin alphaM-beta2 complex / response to Gram-positive bacterium / response to curcumin / positive regulation of microglial cell mediated cytotoxicity / cell-cell adhesion via plasma-membrane adhesion molecules / complement component C3b binding / vertebrate eye-specific patterning ...ectodermal cell differentiation / positive regulation of neutrophil degranulation / cellular extravasation / integrin alphaM-beta2 complex / response to Gram-positive bacterium / response to curcumin / positive regulation of microglial cell mediated cytotoxicity / cell-cell adhesion via plasma-membrane adhesion molecules / complement component C3b binding / vertebrate eye-specific patterning / complement-mediated synapse pruning / Toll Like Receptor 4 (TLR4) Cascade / leukocyte migration involved in inflammatory response / complement receptor mediated signaling pathway / integrin complex / heterotypic cell-cell adhesion / cargo receptor activity / leukocyte cell-cell adhesion / phagocytosis, engulfment / negative regulation of dopamine metabolic process / cell adhesion mediated by integrin / forebrain development / amyloid-beta clearance / tertiary granule membrane / plasma membrane raft / positive regulation of protein targeting to membrane / Integrin cell surface interactions / response to mechanical stimulus / phagocytosis / endothelial cell migration / specific granule membrane / cell adhesion molecule binding / positive regulation of superoxide anion generation / heat shock protein binding / receptor-mediated endocytosis / response to ischemia / cell-matrix adhesion / integrin-mediated signaling pathway / Cell surface interactions at the vascular wall / microglial cell activation / cell-cell adhesion / integrin binding / positive regulation of angiogenesis / positive regulation of nitric oxide biosynthetic process / response to estradiol / amyloid-beta binding / Interleukin-4 and Interleukin-13 signaling / cell adhesion / membrane raft / innate immune response / external side of plasma membrane / Neutrophil degranulation / protein-containing complex binding / cell surface / extracellular space / extracellular exosome / metal ion binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.7 Å | |||||||||
 データ登録者 データ登録者 | Goldsmith JA / McLellan JS | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell Rep / 年: 2022 ジャーナル: Cell Rep / 年: 2022タイトル: Structural basis for non-canonical integrin engagement by Bordetella adenylate cyclase toxin. 著者: Jory A Goldsmith / Andrea M DiVenere / Jennifer A Maynard / Jason S McLellan /  要旨: Integrins are ubiquitous cell-surface heterodimers that are exploited by pathogens and toxins, including leukotoxins that target β integrins on phagocytes. The Bordetella adenylate cyclase toxin ...Integrins are ubiquitous cell-surface heterodimers that are exploited by pathogens and toxins, including leukotoxins that target β integrins on phagocytes. The Bordetella adenylate cyclase toxin (ACT) uses the αβ integrin as a receptor, but the structural basis for integrin binding and neutralization by antibodies is poorly understood. Here, we use cryoelectron microscopy to determine a 2.7 Å resolution structure of an ACT fragment bound to αβ. This structure reveals that ACT interacts with the headpiece and calf-2 of the α subunit in a non-canonical manner specific to bent, inactive αβ. Neutralizing antibody epitopes map to ACT residues involved in α binding, providing the basis for antibody-mediated attachment inhibition. Furthermore, binding to αβ positions the essential ACT acylation sites, which are conserved among toxins exported by type I secretion systems, at the cell membrane. These findings reveal a structural mechanism for integrin-mediated attachment and explain antibody-mediated neutralization of ACT intoxication. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26739.map.gz emd_26739.map.gz | 119 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26739-v30.xml emd-26739-v30.xml emd-26739.xml emd-26739.xml | 12.3 KB 12.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_26739.png emd_26739.png | 53.3 KB | ||
| Filedesc metadata |  emd-26739.cif.gz emd-26739.cif.gz | 6.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26739 http://ftp.pdbj.org/pub/emdb/structures/EMD-26739 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26739 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26739 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_26739_validation.pdf.gz emd_26739_validation.pdf.gz | 363.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_26739_full_validation.pdf.gz emd_26739_full_validation.pdf.gz | 363.3 KB | 表示 | |
| XML形式データ |  emd_26739_validation.xml.gz emd_26739_validation.xml.gz | 6.8 KB | 表示 | |
| CIF形式データ |  emd_26739_validation.cif.gz emd_26739_validation.cif.gz | 7.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26739 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26739 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26739 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26739 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7usmMC  7uslC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26739.map.gz / 形式: CCP4 / 大きさ: 147.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26739.map.gz / 形式: CCP4 / 大きさ: 147.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Composite map of sharpened global and local reconstructions | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.073 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Integrin alphaM/beta2 ectodomain
| 全体 | 名称: Integrin alphaM/beta2 ectodomain |
|---|---|
| 要素 |
|
-超分子 #1: Integrin alphaM/beta2 ectodomain
| 超分子 | 名称: Integrin alphaM/beta2 ectodomain / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Integrin alpha-M
| 分子 | 名称: Integrin alpha-M / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 128.455289 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: FNLDTENAMT FQENARGFGQ SVVQLQGSRV VVGAPQEIVA ANQRGSLYQC DYSTGSCEPI RLQVPVEAVN MSLGLSLAAT TSPPQLLAC GPTVHQTCSE NTYVKGLCFL FGSNLRQQPQ KFPEALRGCP QEDSDIAFLI DGSGSIIPHD FRRMKEFVST V MEQLKKSK ...文字列: FNLDTENAMT FQENARGFGQ SVVQLQGSRV VVGAPQEIVA ANQRGSLYQC DYSTGSCEPI RLQVPVEAVN MSLGLSLAAT TSPPQLLAC GPTVHQTCSE NTYVKGLCFL FGSNLRQQPQ KFPEALRGCP QEDSDIAFLI DGSGSIIPHD FRRMKEFVST V MEQLKKSK TLFSLMQYSE EFRIHFTFKE FQNNPNPRSL VKPITQLLGR THTATGIRKV VRELFNITNG ARKNAFKILV VI TDGEKFG DPLGYEDVIP EADREGVIRY VIGVGDAFRS EKSRQELNTI ASKPPRDHVF QVNNFEALKT IQNQLREKIF AIE GTQTGS SSSFEHEMSQ EGFSAAITSN GPLLSTVGSY DWAGGVFLYT SKEKSTFINM TRVDSDMNDA YLGYAAAIIL RNRV QSLVL GAPRYQHIGL VAMFRQNTGM WESNANVKGT QIGAYFGASL CSVDVDSNGS TDLVLIGAPH YYEQTRGGQV SVCPL PRGR ARWQCDAVLY GEQGQPWGRF GAALTVLGDV NGDKLTDVAI GAPGEEDNRG AVYLFHGTSG SGISPSHSQR IAGSKL SPR LQYFGQSLSG GQDLTMDGLV DLTVGAQGHV LLLRSQPVLR VKAIMEFNPR EVARNVFECN DQVVKGKEAG EVRVCLH VQ KSTRDRLREG QIQSVVTYDL ALDSGRPHSR AVFNETKNST RRQTQVLGLT QTCETLKLQL PNCIEDPVSP IVLRLNFS L VGTPLSAFGN LRPVLAEDAQ RLFTALFPFE KNCGNDNICQ DDLSITFSFM SLDCLVVGGP REFNVTVTVR NDGEDSYRT QVTFFFPLDL SYRKVSTLQN QRSQRSWRLA CESASSTEVS GALKSTSCSI NHPIFPENSE VTFNITFDVD SKASLGNKLL LKANVTSEN NMPRTNKTEF QLELPVKYAV YMVVTSHGVS TKYLNFTASE NTSRVMQHQY QVSNLGQRSL PISLVFLVPV R LNQTVIWD RPQVTFSENL SSTCHTKERL PSHSDFLAEL RKAPVVNCSI AVCQRIQCDI PFFGIQEEFN ATLKGNLSFD WY IKTSHNH LLIVSTAEIL FNDSVFTLLP GQGAFVRSQT ETKVEPFEVP NGCGGLEVLF QGPGENAQCE KELQALEKEN AQL EWELQA LEKELAQWSH PQFEKGGGSG GGGSGGSAWS HPQFEK UniProtKB: Integrin alpha-M |
-分子 #2: Integrin beta
| 分子 | 名称: Integrin beta / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 80.784938 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QECTKFKVSS CRECIESGPG CTWCQKLNFT GPGDPDSIRC DTRPQLLMRG CAADDIMDPT SLAETQEDHN GGQKQLSPQK VTLYLRPGQ AAAFNVTFRR AKGYPIDLYY LMDLSYSMLD DLRNVKKLGG DLLRALNEIT ESGRIGFGSF VDKTVLPFVN T HPDKLRNP ...文字列: QECTKFKVSS CRECIESGPG CTWCQKLNFT GPGDPDSIRC DTRPQLLMRG CAADDIMDPT SLAETQEDHN GGQKQLSPQK VTLYLRPGQ AAAFNVTFRR AKGYPIDLYY LMDLSYSMLD DLRNVKKLGG DLLRALNEIT ESGRIGFGSF VDKTVLPFVN T HPDKLRNP CPNKEKECQP PFAFRHVLKL TNNSNQFQTE VGKQLISGNL DAPEGGLDAM MQVAACPEEI GWRNVTRLLV FA TDDGFHF AGDGKLGAIL TPNDGRCHLE DNLYKRSNEF DYPSVGQLAH KLAENNIQPI FAVTSRMVKT YEKLTEIIPK SAV GELSED SSNVVHLIKN AYNKLSSRVF LDHNALPDTL KVTYDSFCSN GVTHRNQPRG DCDGVQINVP ITFQVKVTAT ECIQ EQSFV IRALGFTDIV TVQVLPQCEC RCRDQSRDRS LCHGKGFLEC GICRCDTGYI GKNCECQTQG RSSQELEGSC RKDNN SIIC SGLGDCVCGQ CLCHTSDVPG KLIYGQYCEC DTINCERYNG QVCGGPGRGL CFCGKCRCHP GFEGSACQCE RTTEGC LNP RRVECSGRGR CRCNVCECHS GYQLPLCQEC PGCPSPCGKY ISCAECLKFE KGPFGKNCSA ACPGLQLSNN PVKGRTC KE RDSEGCWVAY TLEQQDGMDR YLIYVDESRE CVAGPDGCGL EVLFQGPGKN AQCKKKLQAL KKKNAQLKWK LQALKKKL A QGGHHHHHH UniProtKB: Integrin beta |
-分子 #4: CALCIUM ION
| 分子 | 名称: CALCIUM ION / タイプ: ligand / ID: 4 / コピー数: 4 / 式: CA |
|---|---|
| 分子量 | 理論値: 40.078 Da |
-分子 #5: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 5 / コピー数: 9 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 80.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: PDB ENTRY PDBモデル - PDB ID: |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 2.7 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 1114678 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー





















 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)





















