登録情報 データベース : EMDB / ID : EMD-25113タイトル DNA-PK complex of DNA end processing 複合体 : Complex of DNA-PKcs, KU70, Ku80, Artemis and DNAタンパク質・ペプチド : DNA-dependent protein kinase catalytic subunitタンパク質・ペプチド : X-ray repair cross-complementing protein 6タンパク質・ペプチド : X-ray repair cross-complementing protein 5タンパク質・ペプチド : Protein artemisDNA : Hairpin_1リガンド : MAGNESIUM IONリガンド : ADENOSINE-5'-TRIPHOSPHATEリガンド : INOSITOL HEXAKISPHOSPHATEリガンド : ZINC ION機能・相同性 分子機能 ドメイン・相同性 構成要素
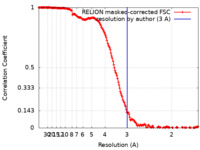
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Human (ヒト) / Homo sapiens (ヒト) / synthetic construct (人工物) 手法 / / 解像度 : 3.0 Å Liu L / Li J / Chen X / Yang W / Gellert M 資金援助 Organization Grant number 国 National Institutes of Health/National Institute of Diabetes and Digestive and Kidney Disease (NIH/NIDDK) M.G., DK036167 National Institutes of Health/National Institute of Diabetes and Digestive and Kidney Disease (NIH/NIDDK) W.Y., DK036147 National Institutes of Health/National Institute of Diabetes and Digestive and Kidney Disease (NIH/NIDDK) DK036144
ジャーナル : Mol Cell / 年 : 2022タイトル : Autophosphorylation transforms DNA-PK from protecting to processing DNA ends.著者 : Lan Liu / Xuemin Chen / Jun Li / Huaibin Wang / Christopher J Buehl / Noah J Goff / Katheryn Meek / Wei Yang / Martin Gellert / 要旨 : The DNA-dependent protein kinase (DNA-PK) initially protects broken DNA ends but then promotes their processing during non-homologous end joining (NHEJ). Before ligation by NHEJ, DNA hairpin ends ... The DNA-dependent protein kinase (DNA-PK) initially protects broken DNA ends but then promotes their processing during non-homologous end joining (NHEJ). Before ligation by NHEJ, DNA hairpin ends generated during V(D)J recombination must be opened by the Artemis nuclease, together with autophosphorylated DNA-PK. Structures of DNA-PK bound to DNA before and after phosphorylation, and in complex with Artemis and a DNA hairpin, reveal an essential functional switch. When bound to open DNA ends in its protection mode, DNA-PK is inhibited for cis-autophosphorylation of the so-called ABCDE cluster but activated for phosphorylation of other targets. In contrast, DNA hairpin ends promote cis-autophosphorylation. Phosphorylation of four Thr residues in ABCDE leads to gross structural rearrangement of DNA-PK, widening the DNA binding groove for Artemis recruitment and hairpin cleavage. Meanwhile, Artemis locks DNA-PK into the kinase-inactive state. Kinase activity and autophosphorylation of DNA-PK are regulated by different DNA ends, feeding forward to coordinate NHEJ events. 履歴 登録 2021年10月6日 - ヘッダ(付随情報) 公開 2022年1月12日 - マップ公開 2022年1月12日 - 更新 2022年1月19日 - 現状 2022年1月19日 処理サイト : RCSB / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 マップデータ
マップデータ 試料
試料 機能・相同性情報
機能・相同性情報 Human (ヒト) /
Human (ヒト) /  Homo sapiens (ヒト) / synthetic construct (人工物)
Homo sapiens (ヒト) / synthetic construct (人工物) データ登録者
データ登録者 米国, 3件
米国, 3件  引用
引用 ジャーナル: Mol Cell / 年: 2022
ジャーナル: Mol Cell / 年: 2022
 構造の表示
構造の表示 ムービービューア
ムービービューア SurfView
SurfView Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク emd_25113.map.gz
emd_25113.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-25113-v30.xml
emd-25113-v30.xml emd-25113.xml
emd-25113.xml EMDBヘッダ
EMDBヘッダ emd_25113_fsc.xml
emd_25113_fsc.xml FSCデータファイル
FSCデータファイル emd_25113.png
emd_25113.png emd_25113_msk_1.map
emd_25113_msk_1.map マスクマップ
マスクマップ emd_25113_half_map_1.map.gz
emd_25113_half_map_1.map.gz emd_25113_half_map_2.map.gz
emd_25113_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-25113
http://ftp.pdbj.org/pub/emdb/structures/EMD-25113 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25113
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25113 emd_25113_validation.pdf.gz
emd_25113_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_25113_full_validation.pdf.gz
emd_25113_full_validation.pdf.gz emd_25113_validation.xml.gz
emd_25113_validation.xml.gz emd_25113_validation.cif.gz
emd_25113_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25113
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25113 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25113
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25113 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_25113.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_25113.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) emd_25113_msk_1.map
emd_25113_msk_1.map 試料の構成要素
試料の構成要素 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 画像解析
画像解析
 ムービー
ムービー コントローラー
コントローラー






























 Z
Z Y
Y X
X