+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of human NKCC1 K289NA492E bound with bumetanide | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | ION TRANSPORT / TRANSPORT PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of cell volume / positive regulation of aspartate secretion / transepithelial ammonium transport / regulation of matrix metallopeptidase secretion / cell body membrane / metal ion transmembrane transporter activity / inorganic anion import across plasma membrane / inorganic cation import across plasma membrane / chloride:monoatomic cation symporter activity / sodium:potassium:chloride symporter activity ...positive regulation of cell volume / positive regulation of aspartate secretion / transepithelial ammonium transport / regulation of matrix metallopeptidase secretion / cell body membrane / metal ion transmembrane transporter activity / inorganic anion import across plasma membrane / inorganic cation import across plasma membrane / chloride:monoatomic cation symporter activity / sodium:potassium:chloride symporter activity / Cation-coupled Chloride cotransporters / potassium ion transmembrane transporter activity / transepithelial chloride transport / intracellular chloride ion homeostasis / negative regulation of vascular wound healing / ammonium transmembrane transport / sodium ion homeostasis / ammonium channel activity / chloride ion homeostasis / cell projection membrane / cellular response to chemokine / T cell chemotaxis / potassium ion homeostasis / intracellular sodium ion homeostasis / sodium ion import across plasma membrane / cell volume homeostasis / cellular response to potassium ion / hyperosmotic response / regulation of spontaneous synaptic transmission / gamma-aminobutyric acid signaling pathway / maintenance of blood-brain barrier / potassium ion import across plasma membrane / intracellular potassium ion homeostasis / lateral plasma membrane / transport across blood-brain barrier / monoatomic ion transport / sodium ion transmembrane transport / cytoplasmic vesicle membrane / basal plasma membrane / chloride transmembrane transport / cell periphery / cell projection / Hsp90 protein binding / extracellular vesicle / protein-folding chaperone binding / cell body / basolateral plasma membrane / neuron projection / apical plasma membrane / neuronal cell body / protein kinase binding / extracellular exosome / membrane / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.6 Å | |||||||||
 データ登録者 データ登録者 | Zhao YX / Cao E | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Structural basis for inhibition of the Cation-chloride cotransporter NKCC1 by the diuretic drug bumetanide 著者: Zhao Y / Roy K / Vidossich P / Cancedda L / De Vivo M / Forbush B / Cao E | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_24812.map.gz emd_24812.map.gz | 59.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-24812-v30.xml emd-24812-v30.xml emd-24812.xml emd-24812.xml | 15.4 KB 15.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_24812.png emd_24812.png | 36.3 KB | ||
| Filedesc metadata |  emd-24812.cif.gz emd-24812.cif.gz | 6.3 KB | ||
| その他 |  emd_24812_additional_1.map.gz emd_24812_additional_1.map.gz | 59.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-24812 http://ftp.pdbj.org/pub/emdb/structures/EMD-24812 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24812 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24812 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_24812_validation.pdf.gz emd_24812_validation.pdf.gz | 494 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_24812_full_validation.pdf.gz emd_24812_full_validation.pdf.gz | 493.6 KB | 表示 | |
| XML形式データ |  emd_24812_validation.xml.gz emd_24812_validation.xml.gz | 6.3 KB | 表示 | |
| CIF形式データ |  emd_24812_validation.cif.gz emd_24812_validation.cif.gz | 7.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24812 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24812 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24812 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24812 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7s1yMC  7s1xC  7s1zC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
| 電子顕微鏡画像生データ |  EMPIAR-11048 (タイトル: Cryo-EM structure of human NKCC1 K289NA492E bound with bumetanide EMPIAR-11048 (タイトル: Cryo-EM structure of human NKCC1 K289NA492E bound with bumetanideData size: 2.1 TB Data #1: Cryo-EM structure of human NKCC1 K289NA492E bound with bumetanide [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_24812.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_24812.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: #1
| ファイル | emd_24812_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : hNKCC1 K289NA492E bound with bumetanide
| 全体 | 名称: hNKCC1 K289NA492E bound with bumetanide |
|---|---|
| 要素 |
|
-超分子 #1: hNKCC1 K289NA492E bound with bumetanide
| 超分子 | 名称: hNKCC1 K289NA492E bound with bumetanide / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Solute carrier family 12 member 2
| 分子 | 名称: Solute carrier family 12 member 2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 131.841594 KDa |
| 組換発現 | 生物種: Mammalian expression vector BsrGI-MCS-pcDNA3.1 (その他) |
| 配列 | 文字列: GAMGSEPRPT APSSGAPGLA GVGETPSAAA LAAARVELPG TAVPSVPEDA APASRDGGGV RDEGPAAAGD GLGRPLGPTP SQSRFQVDL VSENAGRAAA AAAAAAAAAA AAGAGAGAKQ TPADGEASGE SEPAKGSEEA KGRFRVNFVD PAASSSAEDS L SDAAGVGV ...文字列: GAMGSEPRPT APSSGAPGLA GVGETPSAAA LAAARVELPG TAVPSVPEDA APASRDGGGV RDEGPAAAGD GLGRPLGPTP SQSRFQVDL VSENAGRAAA AAAAAAAAAA AAGAGAGAKQ TPADGEASGE SEPAKGSEEA KGRFRVNFVD PAASSSAEDS L SDAAGVGV DGPNVSFQNG GDTVLSEGSS LHSGGGGSGH HQHYYYDTHT NTYYLRTFGH NTMDAVPRID HYRHTAAQLG EK LLRPSLA ELHDELEKEP FEDGFANGEE STPTRDAVVT YTAESKGVVK FGWINGVLVR CMLNIWGVML FIRLSWIVGQ AGI GLSVLV IMMATVVTTI TGLSTSAIAT NGFVRGGGAY YLISRSLGPE FGGAIGLIFA FANAVAVAMY VVGFAETVVE LLKE HSILM IDEINDIRII GAITVVILLG ISVAGMEWEA KAQIVLLVIL LLAIGDFVIG TFIPLESKKP KGFFGYKSEI FNENF GPDF REEETFFSVF EIFFPAATGI LAGANISGDL ADPQSALPKG TLLAILITTL VYVGIAVSVG SCVVRDATGN VNDTIV TEL TNCTSAACKL NFDFSSCESS PCSYGLMNNF QVMSMVSGFT PLISAGIFSA TLSSALASLV SAPKIFQALC KDNIYPA FQ MFAKGYGKNN EPLRGYILTF LIALGFILIA ELNVIAPIIS NFFLASYALI NFSVFHASLA KSPGWRPAFK YYNMWISL L GAILCCIVMF VINWWAALLT YVIVLGLYIY VTYKKPDVNW GSSTQALTYL NALQHSIRLS GVEDHVKNFR PQCLVMTGA PNSRPALLHL VHDFTKNVGL MICGHVHMGP RRQAMKEMSI DQAKYQRWLI KNKMKAFYAP VHADDLREGA QYLMQAAGLG RMKPNTLVL GFKKDWLQAD MRDVDMYINL FHDAFDIQYG VVVIRLKEGL DISHLQGQEE LLSSQEKSPG TKDVVVSVEY S KKSDLDTS KPLSEKPITH KVEEEDGKTA TQPLLKKESK GPIVPLNVAD QKLLEASTQF QKKQGKNTID VWWLFDDGGL TL LIPYLLT TKKKWKDCKI RVFIGGKINR IDHDRRAMAT LLSKFRIDFS DIMVLGDINT KPKKENIIAF EEIIEPYRLH EDD KEQDIA DKMKEDEPWR ITDNELELYK TKTYRQIRLN ELLKEHSSTA NIIVMSLPVA RKGAVSSALY MAWLEALSKD LPPI LLVRG NHQSVLTFYS UniProtKB: Solute carrier family 12 member 2 |
-分子 #2: POTASSIUM ION
| 分子 | 名称: POTASSIUM ION / タイプ: ligand / ID: 2 / コピー数: 2 / 式: K |
|---|---|
| 分子量 | 理論値: 39.098 Da |
-分子 #3: CHLORIDE ION
| 分子 | 名称: CHLORIDE ION / タイプ: ligand / ID: 3 / コピー数: 2 / 式: CL |
|---|---|
| 分子量 | 理論値: 35.453 Da |
-分子 #4: 3-(butylamino)-4-phenoxy-5-sulfamoylbenzoic acid
| 分子 | 名称: 3-(butylamino)-4-phenoxy-5-sulfamoylbenzoic acid / タイプ: ligand / ID: 4 / コピー数: 2 / 式: 82U |
|---|---|
| 分子量 | 理論値: 364.416 Da |
| Chemical component information | 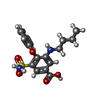 ChemComp-82U: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 1.175 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-7s1y: |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)