[日本語] English
 万見
万見- EMDB-24783: CryoEM structure of Vibrio cholerae transposon Tn6677 AAA+ ATPase TnsC -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of Vibrio cholerae transposon Tn6677 AAA+ ATPase TnsC | |||||||||
 マップデータ マップデータ | post-processed map with deepEMhacer | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | CRISPR / transposon / ATPase / AAA+ / TnsC / DNA BINDING PROTEIN | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | Hoffmann FT / Kim M | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2022 ジャーナル: Nature / 年: 2022タイトル: Selective TnsC recruitment enhances the fidelity of RNA-guided transposition. 著者: Florian T Hoffmann / Minjoo Kim / Leslie Y Beh / Jing Wang / Phuc Leo H Vo / Diego R Gelsinger / Jerrin Thomas George / Christopher Acree / Jason T Mohabir / Israel S Fernández / Samuel H Sternberg /   要旨: Bacterial transposons are pervasive mobile genetic elements that use distinct DNA-binding proteins for horizontal transmission. For example, Escherichia coli Tn7 homes to a specific attachment site ...Bacterial transposons are pervasive mobile genetic elements that use distinct DNA-binding proteins for horizontal transmission. For example, Escherichia coli Tn7 homes to a specific attachment site using TnsD, whereas CRISPR-associated transposons use type I or type V Cas effectors to insert downstream of target sites specified by guide RNAs. Despite this targeting diversity, transposition invariably requires TnsB, a DDE-family transposase that catalyses DNA excision and insertion, and TnsC, a AAA+ ATPase that is thought to communicate between transposase and targeting proteins. How TnsC mediates this communication and thereby regulates transposition fidelity has remained unclear. Here we use chromatin immunoprecipitation with sequencing to monitor in vivo formation of the type I-F RNA-guided transpososome, enabling us to resolve distinct protein recruitment events before integration. DNA targeting by the TniQ-Cascade complex is surprisingly promiscuous-hundreds of genomic off-target sites are sampled, but only a subset of those sites is licensed for TnsC and TnsB recruitment, revealing a crucial proofreading checkpoint. To advance the mechanistic understanding of interactions responsible for transpososome assembly, we determined structures of TnsC using cryogenic electron microscopy and found that ATP binding drives the formation of heptameric rings that thread DNA through the central pore, thereby positioning the substrate for downstream integration. Collectively, our results highlight the molecular specificity imparted by consecutive factor binding to genomic target sites during RNA-guided transposition, and provide a structural roadmap to guide future engineering efforts. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_24783.map.gz emd_24783.map.gz | 1.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-24783-v30.xml emd-24783-v30.xml emd-24783.xml emd-24783.xml | 20.5 KB 20.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
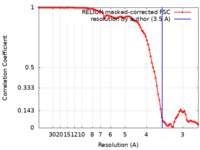
| FSC (解像度算出) |  emd_24783_fsc.xml emd_24783_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_24783.png emd_24783.png | 112.4 KB | ||
| マスクデータ |  emd_24783_msk_1.map emd_24783_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-24783.cif.gz emd-24783.cif.gz | 5.7 KB | ||
| その他 |  emd_24783_additional_1.map.gz emd_24783_additional_1.map.gz emd_24783_additional_2.map.gz emd_24783_additional_2.map.gz emd_24783_half_map_1.map.gz emd_24783_half_map_1.map.gz emd_24783_half_map_2.map.gz emd_24783_half_map_2.map.gz | 49.7 MB 5.2 MB 49.7 MB 49.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-24783 http://ftp.pdbj.org/pub/emdb/structures/EMD-24783 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24783 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24783 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_24783_validation.pdf.gz emd_24783_validation.pdf.gz | 638.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_24783_full_validation.pdf.gz emd_24783_full_validation.pdf.gz | 638.4 KB | 表示 | |
| XML形式データ |  emd_24783_validation.xml.gz emd_24783_validation.xml.gz | 16.1 KB | 表示 | |
| CIF形式データ |  emd_24783_validation.cif.gz emd_24783_validation.cif.gz | 20.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24783 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24783 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24783 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24783 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_24783.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_24783.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | post-processed map with deepEMhacer | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.36333 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_24783_msk_1.map emd_24783_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
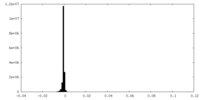
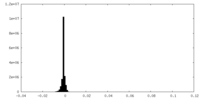
| 密度ヒストグラム |
-追加マップ: unsharpened map
| ファイル | emd_24783_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
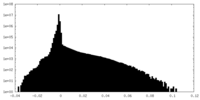
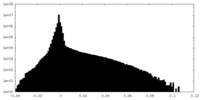
| 密度ヒストグラム |
-追加マップ: Relion3 post-processed map
| ファイル | emd_24783_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Relion3 post-processed map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
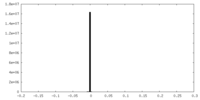
| 密度ヒストグラム |
-ハーフマップ: half-map-1
| ファイル | emd_24783_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half-map-1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
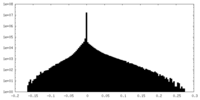
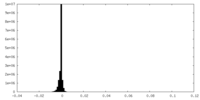
| 密度ヒストグラム |
-ハーフマップ: half-map-2
| ファイル | emd_24783_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half-map-2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
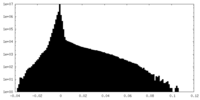
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : VchTnsC
| 全体 | 名称: VchTnsC |
|---|---|
| 要素 |
|
-超分子 #1: VchTnsC
| 超分子 | 名称: VchTnsC / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 238 KDa |
-分子 #1: Tn6677 Vibrio cholerae transposon TnsC (VchTnsC)
| 分子 | 名称: Tn6677 Vibrio cholerae transposon TnsC (VchTnsC) / タイプ: protein_or_peptide / ID: 1 / コピー数: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 37.596043 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSETREARIS RAKRAFVSTP SVRKILSYMD RCRDLSDLES EPTCMMVYGA SGVGKTTVIK KYLNQNRRES EAGGDIIPVL HIELPDNAK PVDAARELLV EMGDPLALYE TDLARLTKRL TELIPAVGVK LIIIDEFQHL VEERSNRVLT QVGNWLKMIL N KTKCPIVI ...文字列: MSETREARIS RAKRAFVSTP SVRKILSYMD RCRDLSDLES EPTCMMVYGA SGVGKTTVIK KYLNQNRRES EAGGDIIPVL HIELPDNAK PVDAARELLV EMGDPLALYE TDLARLTKRL TELIPAVGVK LIIIDEFQHL VEERSNRVLT QVGNWLKMIL N KTKCPIVI FGMPYSKVVL QANSQLHGRF SIQVELRPFS YNGGRGVFKT FLEYLDKALP FEKQAGLANE SLQKKLYAFS QG NMRSLRN LIYQASIEAI DNQHETITEE DFVFASKLTS GDKPNSWKNP FEEGVEVTED MLRPPPKDIG WEDYLRHSTP RVS KPGRNK NFFE |
-分子 #2: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 2 / コピー数: 7 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.2 |
|---|---|
| グリッド | モデル: Homemade / 材質: GOLD / メッシュ: 300 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 70.0 K / 最高: 80.0 K |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 25.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 51.0 µm 最大 デフォーカス(補正後): 1.9000000000000001 µm 最小 デフォーカス(補正後): 0.55 µm / 倍率(補正後): 65000 / 照射モード: OTHER / 撮影モード: BRIGHT FIELD / Cs: 0.001 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 65000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: BACKBONE TRACE |
|---|---|
| 得られたモデル |  PDB-7rzy: |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)