+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-24551 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | MicroED structure of the human adenosine receptor at 2.8A | |||||||||||||||
 マップデータ マップデータ | 2mFo-dFc map of A2A AR by MicroED | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | MEMBRANE PROTEIN | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of acetylcholine secretion, neurotransmission / positive regulation of circadian sleep/wake cycle, sleep / regulation of norepinephrine secretion / negative regulation of alpha-beta T cell activation / Adenosine P1 receptors / G protein-coupled adenosine receptor activity / G protein-coupled adenosine receptor signaling pathway / response to purine-containing compound / sensory perception / positive regulation of urine volume ...positive regulation of acetylcholine secretion, neurotransmission / positive regulation of circadian sleep/wake cycle, sleep / regulation of norepinephrine secretion / negative regulation of alpha-beta T cell activation / Adenosine P1 receptors / G protein-coupled adenosine receptor activity / G protein-coupled adenosine receptor signaling pathway / response to purine-containing compound / sensory perception / positive regulation of urine volume / NGF-independant TRKA activation / Surfactant metabolism / synaptic transmission, dopaminergic / : / inhibitory postsynaptic potential / negative regulation of vascular permeability / synaptic transmission, cholinergic / type 5 metabotropic glutamate receptor binding / positive regulation of glutamate secretion / blood circulation / response to caffeine / intermediate filament / eating behavior / presynaptic active zone / alpha-actinin binding / membrane depolarization / regulation of calcium ion transport / asymmetric synapse / axolemma / : / cellular defense response / prepulse inhibition / phagocytosis / response to amphetamine / presynaptic modulation of chemical synaptic transmission / excitatory postsynaptic potential / positive regulation of synaptic transmission, glutamatergic / neuron projection morphogenesis / regulation of mitochondrial membrane potential / synaptic transmission, glutamatergic / positive regulation of long-term synaptic potentiation / locomotory behavior / central nervous system development / astrocyte activation / positive regulation of synaptic transmission, GABAergic / positive regulation of protein secretion / apoptotic signaling pathway / positive regulation of apoptotic signaling pathway / electron transport chain / adenylate cyclase-modulating G protein-coupled receptor signaling pathway / adenylate cyclase-activating G protein-coupled receptor signaling pathway / negative regulation of inflammatory response / vasodilation / blood coagulation / cell-cell signaling / presynaptic membrane / G alpha (s) signalling events / postsynaptic membrane / negative regulation of neuron apoptotic process / periplasmic space / electron transfer activity / calmodulin binding / response to xenobiotic stimulus / inflammatory response / iron ion binding / negative regulation of cell population proliferation / neuronal cell body / glutamatergic synapse / lipid binding / apoptotic process / dendrite / heme binding / protein-containing complex binding / regulation of DNA-templated transcription / enzyme binding / identical protein binding / membrane / plasma membrane 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||
| 手法 | 電子線結晶学 / クライオ電子顕微鏡法 / 解像度: 2.79 Å | |||||||||||||||
 データ登録者 データ登録者 | Martynowycz MW / Shiriaeva A | |||||||||||||||
| 資金援助 |  米国, 4件 米国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2021 ジャーナル: Proc Natl Acad Sci U S A / 年: 2021タイトル: MicroED structure of the human adenosine receptor determined from a single nanocrystal in LCP. 著者: Michael W Martynowycz / Anna Shiriaeva / Xuanrui Ge / Johan Hattne / Brent L Nannenga / Vadim Cherezov / Tamir Gonen /  要旨: G protein-coupled receptors (GPCRs), or seven-transmembrane receptors, are a superfamily of membrane proteins that are critically important to physiological processes in the human body. Determining ...G protein-coupled receptors (GPCRs), or seven-transmembrane receptors, are a superfamily of membrane proteins that are critically important to physiological processes in the human body. Determining high-resolution structures of GPCRs without bound cognate signaling partners, such as a G protein, requires crystallization in lipidic cubic phase (LCP). GPCR crystals grown in LCP are often too small for traditional X-ray crystallography. These microcrystals are ideal for investigation by microcrystal electron diffraction (MicroED), but the gel-like nature of LCP makes traditional approaches to MicroED sample preparation insurmountable. Here, we show that the structure of a human A adenosine receptor can be determined by MicroED after converting the LCP into the sponge phase followed by focused ion-beam milling. We determined the structure of the A adenosine receptor to 2.8-Å resolution and resolved an antagonist in its orthosteric ligand-binding site, as well as four cholesterol molecules bound around the receptor. This study lays the groundwork for future structural studies of lipid-embedded membrane proteins by MicroED using single microcrystals that would be impossible with other crystallographic methods. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_24551.map.gz emd_24551.map.gz | 4.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-24551-v30.xml emd-24551-v30.xml emd-24551.xml emd-24551.xml | 16.9 KB 16.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_24551.png emd_24551.png | 35.2 KB | ||
| Filedesc metadata |  emd-24551.cif.gz emd-24551.cif.gz | 6.4 KB | ||
| Filedesc structureFactors |  emd_24551_sf.cif.gz emd_24551_sf.cif.gz | 294.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-24551 http://ftp.pdbj.org/pub/emdb/structures/EMD-24551 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24551 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24551 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_24551_validation.pdf.gz emd_24551_validation.pdf.gz | 498.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_24551_full_validation.pdf.gz emd_24551_full_validation.pdf.gz | 498.4 KB | 表示 | |
| XML形式データ |  emd_24551_validation.xml.gz emd_24551_validation.xml.gz | 4.6 KB | 表示 | |
| CIF形式データ |  emd_24551_validation.cif.gz emd_24551_validation.cif.gz | 5.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24551 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24551 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24551 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24551 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7rm5MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_24551.map.gz / 形式: CCP4 / 大きさ: 4.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_24551.map.gz / 形式: CCP4 / 大きさ: 4.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 2mFo-dFc map of A2A AR by MicroED | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X: 0.667 Å / Y: 0.669 Å / Z: 0.698 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 20 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Adenosine receptor A2a/Soluble cytochrome b562 chimera
| 全体 | 名称: Adenosine receptor A2a/Soluble cytochrome b562 chimera |
|---|---|
| 要素 |
|
-超分子 #1: Adenosine receptor A2a/Soluble cytochrome b562 chimera
| 超分子 | 名称: Adenosine receptor A2a/Soluble cytochrome b562 chimera タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 58.72 KDa |
-分子 #1: Adenosine receptor A2a/Soluble cytochrome b562 chimera
| 分子 | 名称: Adenosine receptor A2a/Soluble cytochrome b562 chimera タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 49.974281 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKTIIALSYI FCLVFADYKD DDDGAPPIMG SSVYITVELA IAVLAILGNV LVCWAVWLNS NLQNVTNYFV VSLAAADIAV GVLAIPFAI TISTGFCAAC HGCLFIACFV LVLTQSSIFS LLAIAIDRYI AIRIPLRYNG LVTGTRAKGI IAICWVLSFA I GLTPMLGW ...文字列: MKTIIALSYI FCLVFADYKD DDDGAPPIMG SSVYITVELA IAVLAILGNV LVCWAVWLNS NLQNVTNYFV VSLAAADIAV GVLAIPFAI TISTGFCAAC HGCLFIACFV LVLTQSSIFS LLAIAIDRYI AIRIPLRYNG LVTGTRAKGI IAICWVLSFA I GLTPMLGW NNCGQPKEGK NHSQGCGEGQ VACLFEDVVP MNYMVYFNFF ACVLVPLLLM LGVYLRIFLA ARRQLADLED NW ETLNDNL KVIEKADNAA QVKDALTKMR AAALDAQKAT PPKLEDKSPD SPEMKDFRHG FDILVGQIDD ALKLANEGKV KEA QAAAEQ LKTTRNAYIQ KYLERARSTL QKEVHAAKSL AIIVGLFALC WLPLHIINCF TFFCPDCSHA PLWLMYLAIV LSHT NSVVN PFIYAYRIRE FRQTFRKIIR SHVLRQQEPF KAHHHHHHHH HH UniProtKB: Adenosine receptor A2a, Soluble cytochrome b562, Adenosine receptor A2a |
-分子 #2: 4-{2-[(7-amino-2-furan-2-yl[1,2,4]triazolo[1,5-a][1,3,5]triazin-5...
| 分子 | 名称: 4-{2-[(7-amino-2-furan-2-yl[1,2,4]triazolo[1,5-a][1,3,5]triazin-5-yl)amino]ethyl}phenol タイプ: ligand / ID: 2 / コピー数: 1 / 式: ZMA |
|---|---|
| 分子量 | 理論値: 337.336 Da |
| Chemical component information | 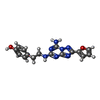 ChemComp-ZMA: |
-分子 #3: CHOLESTEROL
| 分子 | 名称: CHOLESTEROL / タイプ: ligand / ID: 3 / コピー数: 4 / 式: CLR |
|---|---|
| 分子量 | 理論値: 386.654 Da |
| Chemical component information |  ChemComp-CLR: |
-分子 #4: SODIUM ION
| 分子 | 名称: SODIUM ION / タイプ: ligand / ID: 4 / コピー数: 1 |
|---|---|
| 分子量 | 理論値: 22.99 Da |
-分子 #5: water
| 分子 | 名称: water / タイプ: ligand / ID: 5 / コピー数: 6 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 電子線結晶学 |
| 試料の集合状態 | 3D array |
- 試料調製
試料調製
| 濃度 | 10 mg/mL |
|---|---|
| 緩衝液 | pH: 5 詳細: 25-28% (v/v) PEG 400, 0.04-0.06M sodium thiocyanate, 2% (v/v) 2,5-hexanediol, 100mM sodium citrate, pH 5.0, Lipid Cubic Phase (LCP), temperature 293K |
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 298 K / 装置: LEICA EM GP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 77.0 K / 最高: 83.0 K |
| 撮影 | フィルム・検出器のモデル: FEI CETA (4k x 4k) / デジタル化 - サイズ - 横: 2048 pixel / デジタル化 - サイズ - 縦: 2048 pixel / 撮影したグリッド数: 1 / 回折像の数: 134 / 平均露光時間: 3.0 sec. / 平均電子線量: 0.01 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: DIFFRACTION / Cs: 2.7 mm / 最大 デフォーカス(公称値): 0.0 µm / 最小 デフォーカス(公称値): 0.0 µm / カメラ長: 1900 mm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN / 傾斜角度: -40.0, 40.0, 0.6 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー


















