+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23773 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the Neisseria gonorrhoeae ribonucleotide reductase in the inactive state | |||||||||||||||
 マップデータ マップデータ | ||||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | inactive complex / ribonucleotide reductase / OXIDOREDUCTASE | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報ribonucleoside-diphosphate reductase / ribonucleoside-diphosphate reductase activity, thioredoxin disulfide as acceptor / deoxyribonucleotide biosynthetic process / DNA replication / ATP binding 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  Neisseria gonorrhoeae (淋菌) Neisseria gonorrhoeae (淋菌) | |||||||||||||||
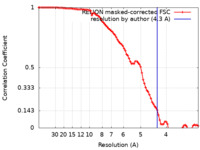
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.3 Å | |||||||||||||||
 データ登録者 データ登録者 | Levitz TS / Drennan CL | |||||||||||||||
| 資金援助 |  米国, 4件 米国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: J Struct Biol / 年: 2022 ジャーナル: J Struct Biol / 年: 2022タイトル: Effects of chameleon dispense-to-plunge speed on particle concentration, complex formation, and final resolution: A case study using the Neisseria gonorrhoeae ribonucleotide reductase inactive complex. 著者: Talya S Levitz / Edward J Brignole / Ivan Fong / Michele C Darrow / Catherine L Drennan /   要旨: Ribonucleotide reductase (RNR) is an essential enzyme that converts ribonucleotides to deoxyribonucleotides and is a promising antibiotic target, but few RNRs have been structurally characterized. We ...Ribonucleotide reductase (RNR) is an essential enzyme that converts ribonucleotides to deoxyribonucleotides and is a promising antibiotic target, but few RNRs have been structurally characterized. We present the use of the chameleon, a commercially-available piezoelectric cryogenic electron microscopy plunger, to address complex denaturation in the Neisseria gonorrhoeae class Ia RNR. Here, we characterize the extent of denaturation of the ring-shaped complex following grid preparation using a traditional plunger and using a chameleon with varying dispense-to-plunge times. We also characterize how dispense-to-plunge time influences the amount of protein sample required for grid preparation and preferred orientation of the sample. We demonstrate that the fastest dispense-to-plunge time of 54 ms is sufficient for generation of a data set that produces a high quality structure, and that a traditional plunging technique or slow chameleon dispense-to-plunge times generate data sets limited in resolution by complex denaturation. The 4.3 Å resolution structure of Neisseria gonorrhoeae class Ia RNR in the inactive α4β4 oligomeric state solved using the chameleon with a fast dispense-to-plunge time yields molecular information regarding similarities and differences to the well studied Escherichia coli class Ia RNR α4β4 ring. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23773.map.gz emd_23773.map.gz | 55.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23773-v30.xml emd-23773-v30.xml emd-23773.xml emd-23773.xml | 16.6 KB 16.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_23773_fsc.xml emd_23773_fsc.xml | 8.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_23773.png emd_23773.png | 89.5 KB | ||
| Filedesc metadata |  emd-23773.cif.gz emd-23773.cif.gz | 6.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23773 http://ftp.pdbj.org/pub/emdb/structures/EMD-23773 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23773 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23773 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_23773_validation.pdf.gz emd_23773_validation.pdf.gz | 665.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_23773_full_validation.pdf.gz emd_23773_full_validation.pdf.gz | 665 KB | 表示 | |
| XML形式データ |  emd_23773_validation.xml.gz emd_23773_validation.xml.gz | 10.2 KB | 表示 | |
| CIF形式データ |  emd_23773_validation.cif.gz emd_23773_validation.cif.gz | 13.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23773 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23773 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23773 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23773 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23773.map.gz / 形式: CCP4 / 大きさ: 59.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23773.map.gz / 形式: CCP4 / 大きさ: 59.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.5998 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Ribonucleotide reductase inactive complex
| 全体 | 名称: Ribonucleotide reductase inactive complex |
|---|---|
| 要素 |
|
-超分子 #1: Ribonucleotide reductase inactive complex
| 超分子 | 名称: Ribonucleotide reductase inactive complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Neisseria gonorrhoeae (淋菌) Neisseria gonorrhoeae (淋菌) |
| 分子量 | 理論値: 528 KDa |
-分子 #1: Ribonucleoside-diphosphate reductase subunit alpha
| 分子 | 名称: Ribonucleoside-diphosphate reductase subunit alpha / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO / EC番号: ribonucleoside-diphosphate reductase |
|---|---|
| 由来(天然) | 生物種:  Neisseria gonorrhoeae (淋菌) / 株: ATCC 700825 / FA 1090 Neisseria gonorrhoeae (淋菌) / 株: ATCC 700825 / FA 1090 |
| 分子量 | 理論値: 87.081344 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGHHHHHHEN LYFQMNTPTD LKVTKRDGRL EAIDLDKIHR VVTWAAEGLE NVSVSQVELK SHIQFYNGIR TDDIHETIIK AAADLISED TPDYQYLAAR LAIFHLRKIA YGEYEPPHLY NHVKKLTDAG KYDRHILEDY SREEFDELNA YIDHERDMSF S YAAVKQLE ...文字列: MGHHHHHHEN LYFQMNTPTD LKVTKRDGRL EAIDLDKIHR VVTWAAEGLE NVSVSQVELK SHIQFYNGIR TDDIHETIIK AAADLISED TPDYQYLAAR LAIFHLRKIA YGEYEPPHLY NHVKKLTDAG KYDRHILEDY SREEFDELNA YIDHERDMSF S YAAVKQLE GKYLVQNRVT RQIYETPQFL YVLVAMCLFS KYPKEARLDY VKRFYDAVST FKVSLPTPIM SGVRTPTRQF SS CVLIECD DSLDSINATT SAIVKYVSQR AGIGINAGRI RGLDSEIRGG EARHTGCIPF FKMFQAAVKS CSQGGVRGGA ATL FYPLWH IEAESLLVLK NNRGVEDNRI RQLDYGVQIN RLLYTRLIKG GNITLFSPNE VSGLYEAFFA DQDEFERLYT KYEQ DPNIR KRIIPAADLF STLMQERAGT GRIYIQNVDH CNTHSPFDPR VAPVHQSNLC MEIALPTKPL DNINDPDGEI ALCTL SAFN LGALNSLDEL EGLADLTVRA LDALLDYQGY PVEAARTSTM DRRSLGIGVI NYAYYLAKNG VRYSDGSALG LTHRTF EAI QYYLLKASAN LAKEYGACTL FNQTVYSQGK LPIDTYKKDL DAVCGEPLHY DWESLRADIV KYGLRNSTLT ALMPSET SS QIANATNGIE PPRGLVTVKA SKDGILKQVV PEFETLKNAY ETLWQLPGNE GYLKLVGVMQ KFVDQAISAN TAYDPGKF E GNKVSMKQML KDLLTAYKYG VKTLYYHNTR DGADDTQTDI QDDGCAGGAC KI UniProtKB: Ribonucleoside-diphosphate reductase |
-分子 #2: Ribonucleoside-diphosphate reductase subunit beta
| 分子 | 名称: Ribonucleoside-diphosphate reductase subunit beta / タイプ: protein_or_peptide / ID: 2 / コピー数: 4 / 光学異性体: LEVO / EC番号: ribonucleoside-diphosphate reductase |
|---|---|
| 由来(天然) | 生物種:  Neisseria gonorrhoeae (淋菌) / 株: ATCC 700825 / FA 1090 Neisseria gonorrhoeae (淋菌) / 株: ATCC 700825 / FA 1090 |
| 分子量 | 理論値: 45.107785 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGHHHHHHEN LYFQMSYSTF PKTKNDALKE PMFFGQPVNV ARYDQQKYEV FEKLIEKQLS FFWRPEEIDV SRDRIDYANL PEHEKHIFI SNLKYQTLLD SIQGRSPNVA LLPLVSIPEL ETWVETWSFS ETIHSRSYTH IIRNIVNDPS VVFDDIVENE Y ITARAEDI ...文字列: MGHHHHHHEN LYFQMSYSTF PKTKNDALKE PMFFGQPVNV ARYDQQKYEV FEKLIEKQLS FFWRPEEIDV SRDRIDYANL PEHEKHIFI SNLKYQTLLD SIQGRSPNVA LLPLVSIPEL ETWVETWSFS ETIHSRSYTH IIRNIVNDPS VVFDDIVENE Y ITARAEDI ACYYDDLIEY TQYYNLLGEG VHNVGGKPVT VSLRGLKKKL YLCLMCVNVL EAIRFYVSFA CSFAFAEREL ME GNAKIIK DIARDEALHL TGTQHMLNLM RSGVDDPEMA EIAAELQDEC FQLFKKAAEQ EKEWAAYLFK DGSMIGLNKE ILS QYVEYI TNLRMQAVGL PAGFEGANQN PIPWINAWLS SDNVQVAPQE VEISSYLIGQ IDSEVNTDDL GDFEL UniProtKB: Ribonucleoside-diphosphate reductase |
-分子 #3: CYTIDINE-5'-DIPHOSPHATE
| 分子 | 名称: CYTIDINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 3 / コピー数: 4 / 式: CDP |
|---|---|
| 分子量 | 理論値: 403.176 Da |
| Chemical component information |  ChemComp-CDP: |
-分子 #4: 2'-DEOXYADENOSINE 5'-TRIPHOSPHATE
| 分子 | 名称: 2'-DEOXYADENOSINE 5'-TRIPHOSPHATE / タイプ: ligand / ID: 4 / コピー数: 8 / 式: DTP |
|---|---|
| 分子量 | 理論値: 491.182 Da |
| Chemical component information |  ChemComp-DTP: |
-分子 #5: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 5 / コピー数: 8 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #6: MU-OXO-DIIRON
| 分子 | 名称: MU-OXO-DIIRON / タイプ: ligand / ID: 6 / コピー数: 4 / 式: FEO |
|---|---|
| 分子量 | 理論値: 127.689 Da |
| Chemical component information |  ChemComp-FEO: |
-分子 #7: water
| 分子 | 名称: water / タイプ: ligand / ID: 7 / コピー数: 24 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 6 mg/mL | ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.6 構成要素:
| ||||||||||
| 凍結 | 凍結剤: ETHANE 詳細: Plunged using the chameleon (SPT labtech) Glow discharged at 12 mA for 250 s. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 撮影したグリッド数: 1 / 実像数: 620 / 平均露光時間: 7.0 sec. / 平均電子線量: 53.15 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.1 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 92000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー