+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | E1435Q Ycf1 mutant in inward-facing wide conformation | |||||||||
 マップデータ マップデータ | Sharpened refine map constructed on unsharpened map using provided mask by Sharpen 3D - cisTEM. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | ABC Transporter / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報ABC-type Cd2+ transporter / ABC-type cadmium transporter activity / Recycling of bile acids and salts / Heme degradation / Aspirin ADME / Atorvastatin ADME / Paracetamol ADME / P-type cadmium transporter activity / bilirubin transmembrane transporter activity / bilirubin transport ...ABC-type Cd2+ transporter / ABC-type cadmium transporter activity / Recycling of bile acids and salts / Heme degradation / Aspirin ADME / Atorvastatin ADME / Paracetamol ADME / P-type cadmium transporter activity / bilirubin transmembrane transporter activity / bilirubin transport / ABC-type glutathione-S-conjugate transporter / ABC-type glutathione S-conjugate transporter activity / vacuole fusion, non-autophagic / ABC-family proteins mediated transport / response to metal ion / fungal-type vacuole membrane / ATPase-coupled transmembrane transporter activity / response to cadmium ion / glutathione metabolic process / cell redox homeostasis / transmembrane transport / membrane raft / ATP hydrolysis activity / ATP binding / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
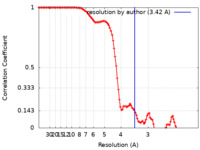
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.42 Å | |||||||||
 データ登録者 データ登録者 | Khandelwal NK / Millan CR | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: The structural basis for regulation of the glutathione transporter Ycf1 by regulatory domain phosphorylation. 著者: Nitesh Kumar Khandelwal / Cinthia R Millan / Samantha I Zangari / Samantha Avila / Dewight Williams / Tarjani M Thaker / Thomas M Tomasiak /  要旨: Yeast Cadmium Factor 1 (Ycf1) sequesters heavy metals and glutathione into the vacuole to counter cell stress. Ycf1 belongs to the ATP binding cassette C-subfamily (ABCC) of transporters, many of ...Yeast Cadmium Factor 1 (Ycf1) sequesters heavy metals and glutathione into the vacuole to counter cell stress. Ycf1 belongs to the ATP binding cassette C-subfamily (ABCC) of transporters, many of which are regulated by phosphorylation on intrinsically-disordered domains. The regulatory mechanism of phosphorylation is still poorly understood. Here, we report two cryo-EM structures of Ycf1 at 3.4 Å and 4.0 Å resolution in inward-facing open conformations that capture previously unobserved ordered states of the intrinsically disordered regulatory domain (R-domain). R-domain phosphorylation is clearly evident and induces a topology promoting electrostatic and hydrophobic interactions with Nucleotide Binding Domain 1 (NBD1) and the Lasso motif. These interactions stay constant between the structures and are related by rigid body movements of the NBD1/R-domain complex. Biochemical data further show R-domain phosphorylation reorganizes the Ycf1 architecture and is required for maximal ATPase activity. Together, we provide insights into how R-domains control ABCC transporter activity. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23691.map.gz emd_23691.map.gz | 95.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23691-v30.xml emd-23691-v30.xml emd-23691.xml emd-23691.xml | 21.2 KB 21.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_23691_fsc.xml emd_23691_fsc.xml | 12.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_23691.png emd_23691.png | 44.4 KB | ||
| マスクデータ |  emd_23691_msk_1.map emd_23691_msk_1.map | 103 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-23691.cif.gz emd-23691.cif.gz | 7.4 KB | ||
| その他 |  emd_23691_additional_1.map.gz emd_23691_additional_1.map.gz emd_23691_half_map_1.map.gz emd_23691_half_map_1.map.gz emd_23691_half_map_2.map.gz emd_23691_half_map_2.map.gz | 95.4 MB 33.8 MB 33.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23691 http://ftp.pdbj.org/pub/emdb/structures/EMD-23691 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23691 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23691 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_23691_validation.pdf.gz emd_23691_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_23691_full_validation.pdf.gz emd_23691_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_23691_validation.xml.gz emd_23691_validation.xml.gz | 18.2 KB | 表示 | |
| CIF形式データ |  emd_23691_validation.cif.gz emd_23691_validation.cif.gz | 23.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23691 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23691 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23691 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23691 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7m69MC  7m68C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23691.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23691.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened refine map constructed on unsharpened map using provided mask by Sharpen 3D - cisTEM. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.031 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_23691_msk_1.map emd_23691_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
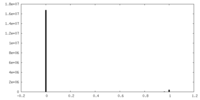
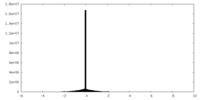
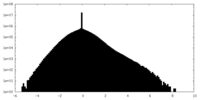
| 密度ヒストグラム |
-追加マップ: Unsharpened map obtained from Manual Refine - cisTEM.
| ファイル | emd_23691_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened map obtained from Manual Refine - cisTEM. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
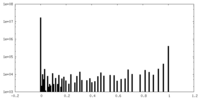
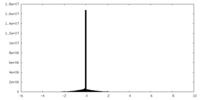
| 密度ヒストグラム |
-ハーフマップ: The second half-map from unsharpened refine map generated...
| ファイル | emd_23691_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | The second half-map from unsharpened refine map generated in Generate 3D - cisTEM. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
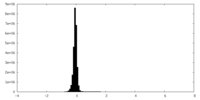
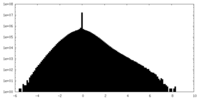
| 密度ヒストグラム |
-ハーフマップ: The first half-map from unsharpened refine map generated...
| ファイル | emd_23691_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | The first half-map from unsharpened refine map generated in Generate 3D - cisTEM. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
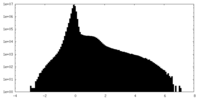
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Ycf1
| 全体 | 名称: Ycf1 |
|---|---|
| 要素 |
|
-超分子 #1: Ycf1
| 超分子 | 名称: Ycf1 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 176.66831 KDa |
-分子 #1: Metal resistance protein YCF1
| 分子 | 名称: Metal resistance protein YCF1 / タイプ: protein_or_peptide / ID: 1 詳細: Phosphorylated residues (S908, T911 and S914) are present in the structure file. コピー数: 1 / 光学異性体: LEVO / EC番号: ABC-type Cd2+ transporter |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 176.318328 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: ASDYKDDDDK GALEVLFQGP SSPMAGNLVS WACKLCRSPE GFGPISFYGD FTQCFIDGVI LNLSAIFMIT FGIRDLVNLC KKKHSGIKY RRNWIIVSRM ALVLLEIAFV SLASLNISKE EAENFTIVSQ YASTMLSLFV ALALHWIEYD RSVVANTVLL F YWLFETFG ...文字列: ASDYKDDDDK GALEVLFQGP SSPMAGNLVS WACKLCRSPE GFGPISFYGD FTQCFIDGVI LNLSAIFMIT FGIRDLVNLC KKKHSGIKY RRNWIIVSRM ALVLLEIAFV SLASLNISKE EAENFTIVSQ YASTMLSLFV ALALHWIEYD RSVVANTVLL F YWLFETFG NFAKLINILI RHTYEGIWYS GQTGFILTLF QVITCASILL LEALPKKPLM PHQHIHQTLT RRKPNPYDSA NI FSRITFS WMSGLMKTGY EKYLVEADLY KLPRNFSSEE LSQKLEKNWE NELKQKSNPS LSWAICRTFG SKMLLAAFFK AIH DVLAFT QPQLLRILIK FVTDYNSERQ DDHSSLQGFE NNHPQKLPIV RGFLIAFAMF LVGFTQTSVL HQYFLNVFNT GMYI KSALT ALIYQKSLVL SNEASGLSST GDIVNLMSVD VQKLQDLTQW LNLIWSGPFQ IIICLYSLYK LLGNSMWVGV IILVI MMPL NSFLMRIQKK LQKSQMKYKD ERTRVISEIL NNIKSLKLYA WEKPYREKLE EVRNNKELKN LTKLGCYMAV TSFQFN IVP FLVSCCTFAV FVYTEDRALT TDLVFPALTL FNLLSFPLMI IPMVLNSFIE ASVSIGRLFT FFTNEELQPD SVQRLPK VK NIGDVAINIG DDATFLWQRK PEYKVALKNI NFQAKKGNLT CIVGKVGSGK TALLSCMLGD LFRVKGFATV HGSVAYVS Q VPWIMNGTVK ENILFGHRYD AEFYEKTIKA CALTIDLAIL MDGDKTLVGE KGISLSGGQK ARLSLARAVY ARADTYLLD DPLAAVDEHV ARHLIEHVLG PNGLLHTKTK VLATNKVSAL SIADSIALLD NGEITQQGTY DEITKDADSP LWKLLNNYGK KNNGKSNEF GDSSESSVRE SSIPVEGELE QLQKLNDLDF GNSDAISLRR A(SEP)DA(TPO)LG(SEP)ID FGDDENIAK REHREQGKVK WNIYLEYAKA CNPKSVCVFI LFIVISMFLS VMGNVWLKHW SEVNSRYGSN PNAARYLAIY FALGIGSALA TLIQTIVLW VFCTIHASKY LHNLMTNSVL RAPMTFFETT PIGRILNRFS NDIYKVDALL GRTFSQFFVN AVKVTFTITV I CATTWQFI FIIIPLSVFY IYYQQYYLRT SRELRRLDSI TRSPIYSHFQ ETLGGLATVR GYSQQKRFSH INQCRIDNNM SA FYPSINA NRWLAYRLEL IGSIIILGAA TLSVFRLKQG TLTAGMVGLS LSYALQITQT LNWIVRMTVE VETNIVSVER IKE YADLKS EAPLIVEGHR PPKEWPSQGD IKFNNYSTRY RPELDLVLKH INIHIKPNEK VGIVGRTGAG KSSLTLALFR MIEA SEGNI VIDNIAINEI GLYDLRHKLS IIPQDSQVFE GTVRENIDPI NQYTDEAIWR ALELSHLKEH VLSMSNDGLD AQLTE GGGN LSVGQRQLLC LARAMLVPSK ILVLDQATAA VDVETDKVVQ ETIRTAFKDR TILTIAHRLN TIMDSDRIIV LDNGKV AEF DSPGQLLSDN KSLFYSLCME AGLVNENGLV PRGSSAHHHH HHHHHHGA UniProtKB: Metal resistance protein YCF1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5.94 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7 構成要素:
詳細: Solution were made fresh in cold distilled water and final pH was adjusted to 7.0 with HCl of cold buffer. The digitonin detergent was added to final .06 % in buffer after pH adjustment. | |||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 400 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: OTHER | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 80 % / チャンバー内温度: 283.15 K / 装置: LEICA EM GP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 8499 / 平均露光時間: 2.9 sec. / 平均電子線量: 54.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 倍率(補正後): 22500 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.1 µm / 最小 デフォーカス(公称値): 0.9 µm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)