+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23608 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | DpK2 bacteriophage tail spike depolymerase | |||||||||
 マップデータ マップデータ | postprocessed map, C3 point group symmetry | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | bateriophage tail spike / depolymerase / klebsiella targeting / SUGAR BINDING PROTEIN | |||||||||
| 機能・相同性 | biological process involved in interaction with host / Pectin lyase fold/virulence factor / viral life cycle / virion component / Depolymerase 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Bacteriophage sp. (ファージ) / Bacteriophage sp. (ファージ) /  Klebsiella phage GH-K3 (ファージ) Klebsiella phage GH-K3 (ファージ) | |||||||||
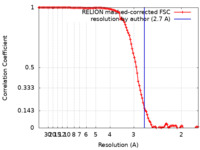
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.7 Å | |||||||||
 データ登録者 データ登録者 | Belousoff MJ / Bamert RS | |||||||||
| 資金援助 |  オーストラリア, 1件 オーストラリア, 1件
| |||||||||
 引用 引用 |  ジャーナル: Microbiol Spectr / 年: 2021 ジャーナル: Microbiol Spectr / 年: 2021タイトル: Mechanistic Insights into the Capsule-Targeting Depolymerase from a Klebsiella pneumoniae Bacteriophage. 著者: Rhys A Dunstan / Rebecca S Bamert / Matthew J Belousoff / Francesca L Short / Christopher K Barlow / Derek J Pickard / Jonathan J Wilksch / Ralf B Schittenhelm / Richard A Strugnell / Gordon ...著者: Rhys A Dunstan / Rebecca S Bamert / Matthew J Belousoff / Francesca L Short / Christopher K Barlow / Derek J Pickard / Jonathan J Wilksch / Ralf B Schittenhelm / Richard A Strugnell / Gordon Dougan / Trevor Lithgow /   要旨: The production of capsular polysaccharides by Klebsiella pneumoniae protects the bacterial cell from harmful environmental factors such as antimicrobial compounds and infection by bacteriophages ...The production of capsular polysaccharides by Klebsiella pneumoniae protects the bacterial cell from harmful environmental factors such as antimicrobial compounds and infection by bacteriophages (phages). To bypass this protective barrier, some phages encode polysaccharide-degrading enzymes referred to as depolymerases to provide access to cell surface receptors. Here, we characterized the phage RAD2, which infects K. pneumoniae strains that produce the widespread, hypervirulence-associated K2-type capsular polysaccharide. Using transposon-directed insertion sequencing, we have shown that the production of capsule is an absolute requirement for efficient RAD2 infection by serving as a first-stage receptor. We have identified the depolymerase responsible for recognition and degradation of the capsule, determined that the depolymerase forms globular appendages on the phage virion tail tip, and present the cryo-electron microscopy structure of the RAD2 capsule depolymerase at 2.7-Å resolution. A putative active site for the enzyme was identified, comprising clustered negatively charged residues that could facilitate the hydrolysis of target polysaccharides. Enzymatic assays coupled with mass spectrometric analyses of digested oligosaccharide products provided further mechanistic insight into the hydrolase activity of the enzyme, which, when incubated with K. pneumoniae, removes the capsule and sensitizes the cells to serum-induced killing. Overall, these findings expand our understanding of how phages target the Klebsiella capsule for infection, providing a framework for the use of depolymerases as antivirulence agents against this medically important pathogen. Klebsiella pneumoniae is a medically important pathogen that produces a thick protective capsule that is essential for pathogenicity. Phages are natural predators of bacteria, and many encode diverse "capsule depolymerases" which specifically degrade the capsule of their hosts, an exploitable trait for potential therapies. We have determined the first structure of a depolymerase that targets the clinically relevant K2 capsule and have identified its putative active site, providing hints to its mechanism of action. We also show that Klebsiella cells treated with a recombinant form of the depolymerase are stripped of capsule, inhibiting their ability to grow in the presence of serum, demonstrating the anti-infective potential of these robust and readily producible enzymes against encapsulated bacterial pathogens such as K. pneumoniae. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23608.map.gz emd_23608.map.gz | 117.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23608-v30.xml emd-23608-v30.xml emd-23608.xml emd-23608.xml | 22.9 KB 22.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_23608_fsc.xml emd_23608_fsc.xml | 11.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_23608.png emd_23608.png | 59.2 KB | ||
| Filedesc metadata |  emd-23608.cif.gz emd-23608.cif.gz | 6 KB | ||
| その他 |  emd_23608_additional_1.map.gz emd_23608_additional_1.map.gz emd_23608_additional_2.map.gz emd_23608_additional_2.map.gz emd_23608_additional_3.map.gz emd_23608_additional_3.map.gz emd_23608_additional_4.map.gz emd_23608_additional_4.map.gz emd_23608_half_map_1.map.gz emd_23608_half_map_1.map.gz emd_23608_half_map_2.map.gz emd_23608_half_map_2.map.gz | 78.3 MB 98.3 MB 117.3 MB 98.6 MB 99 MB 99 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23608 http://ftp.pdbj.org/pub/emdb/structures/EMD-23608 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23608 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23608 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_23608_validation.pdf.gz emd_23608_validation.pdf.gz | 786.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_23608_full_validation.pdf.gz emd_23608_full_validation.pdf.gz | 786 KB | 表示 | |
| XML形式データ |  emd_23608_validation.xml.gz emd_23608_validation.xml.gz | 18.6 KB | 表示 | |
| CIF形式データ |  emd_23608_validation.cif.gz emd_23608_validation.cif.gz | 24.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23608 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23608 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23608 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23608 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23608.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23608.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | postprocessed map, C3 point group symmetry | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.895 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: local resolution filtered map, C3 point group symmetry
| ファイル | emd_23608_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | local resolution filtered map, C3 point group symmetry | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
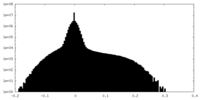
| 密度ヒストグラム |
-追加マップ: unprocessed map, C3 point group symmetry
| ファイル | emd_23608_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unprocessed map, C3 point group symmetry | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
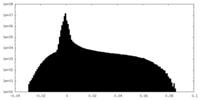
| 密度ヒストグラム |
-追加マップ: post-processed map, C1 point group symmetry
| ファイル | emd_23608_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | post-processed map, C1 point group symmetry | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: unprocessed map, C1 point group symmetry
| ファイル | emd_23608_additional_4.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unprocessed map, C1 point group symmetry | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 1, C3 point group symmetry
| ファイル | emd_23608_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 1, C3 point group symmetry | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
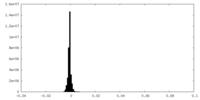
| 密度ヒストグラム |
-ハーフマップ: half map 2, C3 point group symmetry
| ファイル | emd_23608_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2, C3 point group symmetry | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
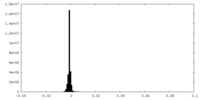
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : DpK2 depolymerase tail spike complex
| 全体 | 名称: DpK2 depolymerase tail spike complex |
|---|---|
| 要素 |
|
-超分子 #1: DpK2 depolymerase tail spike complex
| 超分子 | 名称: DpK2 depolymerase tail spike complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Bacteriophage sp. (ファージ) Bacteriophage sp. (ファージ) |
-分子 #1: Depolymerase
| 分子 | 名称: Depolymerase / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Klebsiella phage GH-K3 (ファージ) Klebsiella phage GH-K3 (ファージ) |
| 分子量 | 理論値: 98.437008 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MALYREGKAA MAADGTVTGT GTKWQSSLSL IRPGATIMFL SSPIQMAVVN KVVSDTEIKA ITTSGAVVAS TDYAILLSDS LTVDGLAQD VAETLRHYQS QETVIADAVE FFKSFDFDSL QNLANQIKAD SESAESSAAA AAASESKAKT SEDNAKSSEN A AKNSEVAA ...文字列: MALYREGKAA MAADGTVTGT GTKWQSSLSL IRPGATIMFL SSPIQMAVVN KVVSDTEIKA ITTSGAVVAS TDYAILLSDS LTVDGLAQD VAETLRHYQS QETVIADAVE FFKSFDFDSL QNLANQIKAD SESAESSAAA AAASESKAKT SEDNAKSSEN A AKNSEVAA ETTRDQIQQI IDNAGDQSTL VVLAQPDGFD SIGRVSSFAA LRNLKPKKSG QHVLLTSYYD GWAAENKMPT GG GEFISSI GTATDDGGYI AAGPGYYWTR VVNNNSFTAE DFGCKTTATP PPNFNVLPAE LFDNTAMMQA AFNLAISKSF KLN LSTGTY YFESSDTLRI TGPIHIEGRP GTVFYHNPSN KANPKTDAFM NISGCSAGRI SSINCFSNSY LGKGINFDRS VGDN RKLVL EHVYVDTFRW GFYVGEPECI NQIEFHSCRA QSNYFQGIFI ESFKEGQEYG HSAPVHFFNT ICNGNGPTSF ALGAT YKTT KNEYIKVMDS VNDVGCQAYF QGLSNVQYIG GQLSGHGSPR NTSLATITQC NSFIIYGTDL EDINGFTTDG TAITAD NID AIESNYLKDI SGAAIVVSSC PGFKIDSPHI FKIKTLSTIK LMNNTYNYEI GGFTPDEALK YNVWDANGLA TNRISGV IH PRLVNSRLGI NSVAFDNMSN KLDVSSLIHN ETSQIVGLTP STGSNVPHTR KMWSNGAMYS STDLNNGFRL NYLSNHNE P LTPMHLYNEF SVSEFGGSVT ESNALDEIKY IFIQTTYANS GDGRFIIQAL DASGSVLSSN WYSPQSFNST FPISGFVRF DVPTGAKKIR YGFVNSANYT GSLRSHFMSG FAYNKRFFLK IYAVYNDLGR YGQFEPPYSV AIDRFRVGDN TTQMPSIPAS SATDVAGVN EVINSLLASL KANGFMSS UniProtKB: Depolymerase |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.4 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 200 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / 平均電子線量: 43.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)