+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23570 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of yeast DNA Polymerase Zeta (apo) | |||||||||
 マップデータ マップデータ | cryo-em map of apo-PolZ | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | nucleic acid binding / DNA polymerase / metal ion binding / catalytic activity / DNA BINDING PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報delta DNA polymerase complex / H3-H4 histone complex chaperone activity / DNA amplification / zeta DNA polymerase complex / RNA-templated DNA biosynthetic process / Processive synthesis on the lagging strand / Removal of the Flap Intermediate / DNA replication, removal of RNA primer / Translesion synthesis by REV1 / Translesion synthesis by POLK ...delta DNA polymerase complex / H3-H4 histone complex chaperone activity / DNA amplification / zeta DNA polymerase complex / RNA-templated DNA biosynthetic process / Processive synthesis on the lagging strand / Removal of the Flap Intermediate / DNA replication, removal of RNA primer / Translesion synthesis by REV1 / Translesion synthesis by POLK / Translesion synthesis by POLI / lagging strand elongation / double-strand break repair via break-induced replication / postreplication repair / DNA strand elongation involved in DNA replication / DNA metabolic process / error-free translesion synthesis / leading strand elongation / error-prone translesion synthesis / mismatch repair / nucleotide-excision repair / double-strand break repair via homologous recombination / base-excision repair / 4 iron, 4 sulfur cluster binding / DNA replication / DNA-directed DNA polymerase / DNA-directed DNA polymerase activity / nucleotide binding / chromatin / mitochondrion / DNA binding / nucleus / metal ion binding / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.11 Å | |||||||||
 データ登録者 データ登録者 | Truong CD / Craig TA | |||||||||
 引用 引用 |  ジャーナル: J Biol Chem / 年: 2021 ジャーナル: J Biol Chem / 年: 2021タイトル: Cryo-EM reveals conformational flexibility in apo DNA polymerase ζ. 著者: Chloe Du Truong / Theodore A Craig / Gaofeng Cui / Maria Victoria Botuyan / Rachel A Serkasevich / Ka-Yi Chan / Georges Mer / Po-Lin Chiu / Rajiv Kumar /  要旨: The translesion synthesis (TLS) DNA polymerases Rev1 and Polζ function together in DNA lesion bypass during DNA replication, acting as nucleotide inserter and extender polymerases, respectively. ...The translesion synthesis (TLS) DNA polymerases Rev1 and Polζ function together in DNA lesion bypass during DNA replication, acting as nucleotide inserter and extender polymerases, respectively. While the structural characterization of the Saccharomyces cerevisiae Polζ in its DNA-bound state has illuminated how this enzyme synthesizes DNA, a mechanistic understanding of TLS also requires probing conformational changes associated with DNA- and Rev1 binding. Here, we used single-particle cryo-electron microscopy to determine the structure of the apo Polζ holoenzyme. We show that compared with its DNA-bound state, apo Polζ displays enhanced flexibility that correlates with concerted motions associated with expansion of the Polζ DNA-binding channel upon DNA binding. We also identified a lysine residue that obstructs the DNA-binding channel in apo Polζ, suggesting a gating mechanism. The Polζ subunit Rev7 is a hub protein that directly binds Rev1 and is a component of several other protein complexes such as the shieldin DNA double-strand break repair complex. We analyzed the molecular interactions of budding yeast Rev7 in the context of Polζ and those of human Rev7 in the context of shieldin using a crystal structure of Rev7 bound to a fragment of the shieldin-3 protein. Overall, our study provides new insights into Polζ mechanism of action and the manner in which Rev7 recognizes partner proteins. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23570.map.gz emd_23570.map.gz | 37.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23570-v30.xml emd-23570-v30.xml emd-23570.xml emd-23570.xml | 25.2 KB 25.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_23570.png emd_23570.png | 53.9 KB | ||
| Filedesc metadata |  emd-23570.cif.gz emd-23570.cif.gz | 8.3 KB | ||
| その他 |  emd_23570_additional_1.map.gz emd_23570_additional_1.map.gz emd_23570_additional_2.map.gz emd_23570_additional_2.map.gz | 37.7 MB 37.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23570 http://ftp.pdbj.org/pub/emdb/structures/EMD-23570 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23570 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23570 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_23570_validation.pdf.gz emd_23570_validation.pdf.gz | 554.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_23570_full_validation.pdf.gz emd_23570_full_validation.pdf.gz | 553.8 KB | 表示 | |
| XML形式データ |  emd_23570_validation.xml.gz emd_23570_validation.xml.gz | 5.9 KB | 表示 | |
| CIF形式データ |  emd_23570_validation.cif.gz emd_23570_validation.cif.gz | 6.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23570 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23570 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23570 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23570 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23570.map.gz / 形式: CCP4 / 大きさ: 40.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23570.map.gz / 形式: CCP4 / 大きさ: 40.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | cryo-em map of apo-PolZ | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.491 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
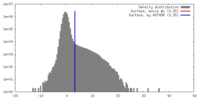
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Half-map 1
| ファイル | emd_23570_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
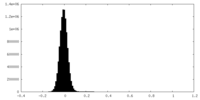
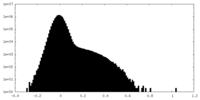
| 密度ヒストグラム |
-追加マップ: Half-map 2
| ファイル | emd_23570_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
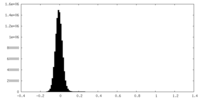
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : DNA polymerase Zeta
| 全体 | 名称: DNA polymerase Zeta |
|---|---|
| 要素 |
|
-超分子 #1: DNA polymerase Zeta
| 超分子 | 名称: DNA polymerase Zeta / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 詳細: DNA polymerase Zeta is generated from yeast without DNA binding. |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 172 kDa/nm |
-分子 #1: DNA polymerase zeta catalytic subunit
| 分子 | 名称: DNA polymerase zeta catalytic subunit / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: DNA-directed DNA polymerase |
|---|---|
| 由来(天然) | 生物種:  株: ATCC 204508 / S288c |
| 分子量 | 理論値: 173.197531 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSRESNDTIQ SDTVRSSSKS DYFRIQLNNQ DYYMSKPTFL DPSHGESLPL NQFSQVPNIR VFGALPTGHQ VLCHVHGILP YMFIKYDGQ ITDTSTLRHQ RCAQVHKTLE VKIRASFKRK KDDKHDLAGD KLGNLNFVAD VSVVKGIPFY GYHVGWNLFY K ISLLNPSC ...文字列: MSRESNDTIQ SDTVRSSSKS DYFRIQLNNQ DYYMSKPTFL DPSHGESLPL NQFSQVPNIR VFGALPTGHQ VLCHVHGILP YMFIKYDGQ ITDTSTLRHQ RCAQVHKTLE VKIRASFKRK KDDKHDLAGD KLGNLNFVAD VSVVKGIPFY GYHVGWNLFY K ISLLNPSC LSRISELIRD GKIFGKKFEI YESHIPYLLQ WTADFNLFGC SWINVDRCYF RSPVLNSILD IDKLTINDDL QL LLDRFCD FKCNVLSRRD FPRVGNGLIE IDILPQFIKN REKLQHRDIH HDFLEKLGDI SDIPVKPYVS SARDMINELT MQR EELSLK EYKEPPETKR HVSGHQWQSS GEFEAFYKKA QHKTSTFDGQ IPNFENFIDK NQKFSAINTP YEALPQLWPR LPQI EINNN SMQDKKNDDQ VNASFTEYEI CGVDNENEGV KGSNIKSRSY SWLPESIASP KDSTILLDHQ TKYHNTINFS MDCAM TQNM ASKRKLRSSV SANKTSLLSR KRKKVMAAGL RYGKRAFVYG EPPFGYQDIL NKLEDEGFPK IDYKDPFFSN PVDLEN KPY AYAGKRFEIS STHVSTRIPV QFGGETVSVY NKPTFDMFSS WKYALKPPTY DAVQKWYNKV PSMGNKKTES QISMHTP HS KFLYKFASDV SGKQKRKKSS VHDSLTHLTL EIHANTRSDK IPDPAIDEVS MIIWCLEEET FPLDLDIAYE GIMIVHKA S EDSTFPTKIQ HCINEIPVMF YESEFEMFEA LTDLVLLLDP DILSGFEIHN FSWGYIIERC QKIHQFDIVR ELARVKCQI KTKLSDTWGY AHSSGIMITG RHMINIWRAL RSDVNLTQYT IESAAFNILH KRLPHFSFES LTNMWNAKKS TTELKTVLNY WLSRAQINI QLLRKQDYIA RNIEQARLIG IDFHSVYYRG SQFKVESFLI RICKSESFIL LSPGKKDVRK QKALECVPLV M EPESAFYK SPLIVLDFQS LYPSIMIGYN YCYSTMIGRV REINLTENNL GVSKFSLPRN ILALLKNDVT IAPNGVVYAK TS VRKSTLS KMLTDILDVR VMIKKTMNEI GDDNTTLKRL LNNKQLALKL LANVTYGYTS ASFSGRMPCS DLADSIVQTG RET LEKAID IIEKDETWNA KVVYGDTDSL FVYLPGKTAI EAFSIGHAMA ERVTQNNPKP IFLKFEKVYH PSILISKKRY VGFS YESPS QTLPIFDAKG IETVRRDGIP AQQKIIEKCI RLLFQTKDLS KIKKYLQNEF FKIQIGKVSA QDFCFAKEVK LGAYK SEKT APAGAVVVKR RINEDHRAEP QYKERIPYLV VKGKQGQLLR ERCVSPEEFL EGENLELDSE YYINKILIPP LDRLFN LIG INVGNWAQEI VKSKRASTTT TKVENITRVG TSATCCNCGE ELTKICSLQL CDDCLEKRST TTLSFLIKKL KRQKEYQ TL KTVCRTCSYR YTSDAGIEND HIASKCNSYD CPVFYSRVKA ERYLRDNQSV QREEALISLN DW UniProtKB: DNA polymerase zeta catalytic subunit |
-分子 #2: DNA polymerase zeta processivity subunit
| 分子 | 名称: DNA polymerase zeta processivity subunit / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  株: ATCC 204508 / S288c |
| 分子量 | 理論値: 28.791654 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MNRWVEKWLR VYLKCYINLI LFYRNVYPPQ SFDYTTYQSF NLPQFVPINR HPALIDYIEE LILDVLSKLT HVYRFSICII NKKNDLCIE KYVLDFSELQ HVDKDDQIIT ETEVFDEFRS SLNSLIMHLE KLPKVNDDTI TFEAVINAIE LELGHKLDRN R RVDSLEEK ...文字列: MNRWVEKWLR VYLKCYINLI LFYRNVYPPQ SFDYTTYQSF NLPQFVPINR HPALIDYIEE LILDVLSKLT HVYRFSICII NKKNDLCIE KYVLDFSELQ HVDKDDQIIT ETEVFDEFRS SLNSLIMHLE KLPKVNDDTI TFEAVINAIE LELGHKLDRN R RVDSLEEK AEIERDSNWV KCQEDENLPD NNGFQPPKIK LTSLVGSDVG PLIIHQFSEK LISGDDKILN GVYSQYEEGE SI FGSLF UniProtKB: DNA polymerase zeta processivity subunit |
-分子 #3: DNA polymerase delta small subunit
| 分子 | 名称: DNA polymerase delta small subunit / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO / EC番号: DNA-directed DNA polymerase |
|---|---|
| 由来(天然) | 生物種:  株: ATCC 204508 / S288c |
| 分子量 | 理論値: 55.603992 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: LHMDALLTKF NEDRSLQDEN LSQPRTRVRI VDDNLYNKSN PFQLCYKKRD YGSQYYHIYQ YRLKTFRERV LKECDKRWDA GFTLNGQLV LKKDKVLDIQ GNQPCWCVGS IYCEMKYKPN VLDEVINDTY GAPDLTKSYT DKEGGSDEIM LEDESGRVLL V GDFIRSTP ...文字列: LHMDALLTKF NEDRSLQDEN LSQPRTRVRI VDDNLYNKSN PFQLCYKKRD YGSQYYHIYQ YRLKTFRERV LKECDKRWDA GFTLNGQLV LKKDKVLDIQ GNQPCWCVGS IYCEMKYKPN VLDEVINDTY GAPDLTKSYT DKEGGSDEIM LEDESGRVLL V GDFIRSTP FITGVVVGIL GMEAEAGTFQ VLDICYPTPL PQNPFPAPIA TCPTRGKIAL VSGLNLNNTS PDRLLRLEIL RE FLMGRIN NKIDDISLIG RLLICGNSVD FDIKSVNKDE LMISLTEFSK FLHNILPSIS VDIMPGTNDP SDKSLPQQPF HKS LFDKSL ESYFNGSNKE ILNLVTNPYE FSYNGVDVLA VSGKNINDIC KYVIPSNDNG ESENKVEEGE SNDFKDDIEH RLDL MECTM KWQNIAPTAP DTLWCYPYTD KDPFVLDKWP HVYIVANQPY FGTRVVEIGG KNIKIISVPE FSSTGMIILL DLETL EAET VKIDI UniProtKB: DNA polymerase delta small subunit |
-分子 #4: DNA polymerase delta subunit 3
| 分子 | 名称: DNA polymerase delta subunit 3 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  株: ATCC 204508 / S288c |
| 分子量 | 理論値: 40.377715 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDQKASYFIN EKLFTEVKPV LFTDLIHHLK IGPSMAKKLM FDYYKQTTNA KYNCVVICCY KDQTIKIIHD LSNIPQQDSI IDCFIYAFN PMDSFIPYYD IIDQKDCLTI KNSYELKVSE SSKIIERTKT LEEKSKPLVR PTARSKTTPE ETTGRKSKSK D MGLRSTAL ...文字列: MDQKASYFIN EKLFTEVKPV LFTDLIHHLK IGPSMAKKLM FDYYKQTTNA KYNCVVICCY KDQTIKIIHD LSNIPQQDSI IDCFIYAFN PMDSFIPYYD IIDQKDCLTI KNSYELKVSE SSKIIERTKT LEEKSKPLVR PTARSKTTPE ETTGRKSKSK D MGLRSTAL LAKMKKDRDD KETSRQNELR KRKEENLQKI NKQNPEREAQ MKELNNLFVE DDLDTEEVNG GSKPNSPKET DS NDKDKNN DDLEDLLETT AEDSLMDVPK IQQTKPSETE HSKEPKSEEE PSSFIDEDGY IVTKRPATST PPRKPSPVVK RAL SSSKKQ ETPSSNKRLK KQGTLESFFK RKAK UniProtKB: DNA polymerase delta subunit 3 |
-分子 #5: IRON/SULFUR CLUSTER
| 分子 | 名称: IRON/SULFUR CLUSTER / タイプ: ligand / ID: 5 / コピー数: 1 / 式: SF4 |
|---|---|
| 分子量 | 理論値: 351.64 Da |
| Chemical component information |  ChemComp-FS1: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.12 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 6.9 構成要素:
詳細: Solutions were made fresh from concentrate to avoid microbial contamination. They are further filtered using 0.2 micrometer filtering membrane and a pressure/vacuum filtration unit. | |||||||||||||||
| グリッド | モデル: C-flat-2/1 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 20 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 101.325 kPa | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK II / 詳細: Blot for 6 seconds before plunging. | |||||||||||||||
| 詳細 | This sample was mono disperse. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3710 pixel / デジタル化 - サイズ - 縦: 3838 pixel / 撮影したグリッド数: 1 / 実像数: 11698 / 平均露光時間: 6.0 sec. / 平均電子線量: 45.7 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 倍率(補正後): 48780 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




















 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)