+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
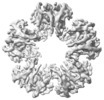
| タイトル | Cryo-EM structure of Mal de Rio Cuarto virus P9-1 viroplasm protein (decamer) | |||||||||||||||
 マップデータ マップデータ | Cryo-EM structure of Mal de Rio Cuarto virus P9-1 viroplasm protein (decamer) | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | Fijivirus / MRCV / Wheat / Maize / Reoviridae / viroplasm / VIRAL PROTEIN | |||||||||||||||
| 機能・相同性 | Reovirus P9-like / Reovirus P9-like family / p9-1 機能・相同性情報 機能・相同性情報 | |||||||||||||||
| 生物種 |  Mal de Rio Cuarto virus (ウイルス) Mal de Rio Cuarto virus (ウイルス) | |||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.7 Å | |||||||||||||||
 データ登録者 データ登録者 | Llauger G / Melero R | |||||||||||||||
| 資金援助 |  アルゼンチン, 4件 アルゼンチン, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: mBio / 年: 2023 ジャーナル: mBio / 年: 2023タイトル: A Fijivirus Major Viroplasm Protein Shows RNA-Stimulated ATPase Activity by Adopting Pentameric and Hexameric Assemblies of Dimers. 著者: Gabriela Llauger / Roberto Melero / Demián Monti / Gabriela Sycz / Cristián Huck-Iriart / María L Cerutti / Sebastián Klinke / Evelyn Mikkelsen / Ariel Tijman / Rocío Arranz / Victoria ...著者: Gabriela Llauger / Roberto Melero / Demián Monti / Gabriela Sycz / Cristián Huck-Iriart / María L Cerutti / Sebastián Klinke / Evelyn Mikkelsen / Ariel Tijman / Rocío Arranz / Victoria Alfonso / Sofía M Arellano / Fernando A Goldbaum / Yann G J Sterckx / José-María Carazo / Sergio B Kaufman / Pablo D Dans / Mariana Del Vas / Lisandro H Otero /    要旨: Fijiviruses replicate and package their genomes within viroplasms in a process involving RNA-RNA and RNA-protein interactions. Here, we demonstrate that the 24 C-terminal residues (C-arm) of the P9-1 ...Fijiviruses replicate and package their genomes within viroplasms in a process involving RNA-RNA and RNA-protein interactions. Here, we demonstrate that the 24 C-terminal residues (C-arm) of the P9-1 major viroplasm protein of the mal de Río Cuarto virus (MRCV) are required for its multimerization and the formation of viroplasm-like structures. Using an integrative structural approach, the C-arm was found to be dispensable for P9-1 dimer assembly but essential for the formation of pentamers and hexamers of dimers (decamers and dodecamers), which favored RNA binding. Although both P9-1 and P9-1ΔC-arm catalyzed ATP with similar activities, an RNA-stimulated ATPase activity was only detected in the full-length protein, indicating a C-arm-mediated interaction between the ATP catalytic site and the allosteric RNA binding sites in the (do)decameric assemblies. A stronger preference to bind phosphate moieties in the decamer was predicted, suggesting that the allosteric modulation of ATPase activity by RNA is favored in this structural conformation. Our work reveals the structural versatility of a fijivirus major viroplasm protein and provides clues to its mechanism of action. The mal de Río Cuarto virus (MRCV) causes an important maize disease in Argentina. MRCV replicates in several species of plants and planthopper vectors. The viral factories, also called viroplasms, have been studied in detail in animal reovirids. This work reveals that a major viroplasm protein of MRCV forms previously unidentified structural arrangements and provides evidence that it may simultaneously adopt two distinct quaternary assemblies. Furthermore, our work uncovers an allosteric communication between the ATP and RNA binding sites that is favored in the multimeric arrangements. Our results contribute to the understanding of plant reovirids viroplasm structure and function and pave the way for the design of antiviral strategies for disease control. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23046.map.gz emd_23046.map.gz | 48.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23046-v30.xml emd-23046-v30.xml emd-23046.xml emd-23046.xml | 14.2 KB 14.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_23046.png emd_23046.png | 87.5 KB | ||
| Filedesc metadata |  emd-23046.cif.gz emd-23046.cif.gz | 6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23046 http://ftp.pdbj.org/pub/emdb/structures/EMD-23046 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23046 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23046 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_23046_validation.pdf.gz emd_23046_validation.pdf.gz | 545.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_23046_full_validation.pdf.gz emd_23046_full_validation.pdf.gz | 545.2 KB | 表示 | |
| XML形式データ |  emd_23046_validation.xml.gz emd_23046_validation.xml.gz | 6.1 KB | 表示 | |
| CIF形式データ |  emd_23046_validation.cif.gz emd_23046_validation.cif.gz | 6.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23046 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23046 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23046 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23046 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7kvcMC  7kvdC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23046.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23046.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of Mal de Rio Cuarto virus P9-1 viroplasm protein (decamer) | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.85 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Decameric complex of Mal de Rio Cuarto virus P9-1 viroplasm protein
| 全体 | 名称: Decameric complex of Mal de Rio Cuarto virus P9-1 viroplasm protein |
|---|---|
| 要素 |
|
-超分子 #1: Decameric complex of Mal de Rio Cuarto virus P9-1 viroplasm protein
| 超分子 | 名称: Decameric complex of Mal de Rio Cuarto virus P9-1 viroplasm protein タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Mal de Rio Cuarto virus (ウイルス) Mal de Rio Cuarto virus (ウイルス) |
-分子 #1: p9-1
| 分子 | 名称: p9-1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 10 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Mal de Rio Cuarto virus (ウイルス) Mal de Rio Cuarto virus (ウイルス) |
| 分子量 | 理論値: 44.933328 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MRGSHHHHHH GMASMTGGQQ MGRDLYDDDD KDRWGSELEI CSWYHGGRGN SIADLERRTF GSYKIEEITI KNDQQQKTTN QQQISNNEQ RISTKKIPIL DDGIFDLINY LLNGTHFDKT HYCGFDYSHL PTLERDFNTA SNYVSENYSI IVEEIDLNKY E RSESISLK ...文字列: MRGSHHHHHH GMASMTGGQQ MGRDLYDDDD KDRWGSELEI CSWYHGGRGN SIADLERRTF GSYKIEEITI KNDQQQKTTN QQQISNNEQ RISTKKIPIL DDGIFDLINY LLNGTHFDKT HYCGFDYSHL PTLERDFNTA SNYVSENYSI IVEEIDLNKY E RSESISLK SPDFTVVLEY FKKHVEGQTE QEENKTESTS SELPAKIVRE LPLLPIMCRE SEDSISEDIL EGEGAVIQVL KM FMKGFLV HLGENPNSYD RQLTIEKYRP LLISIIGYEF TVGTRATHTK INHIYYQLAT FDNYPFDLLR FQLQSLIDTP NVI KERIEK DGLFKVITTT NARGQYQSVL LRGINGSESY LNLKRYRKFK VRVVGNVDNV IKNDFSSLKL DV UniProtKB: p9-1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.6 / 構成要素:
| ||||||
|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER/RHODIUM / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY | ||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: LEICA EM CPC | ||||||
| 詳細 | No previous negative glow discharge was applied on the grids in order to avoid protein aggregation |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 平均電子線量: 30.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最小 デフォーカス(補正後): 0.8 µm / 照射モード: OTHER / 撮影モード: OTHER 最大 デフォーカス(公称値): 3.8000000000000003 µm 倍率(公称値): 120000 |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















