[English] 日本語
 Yorodumi
Yorodumi- EMDB-22853: Ebola virus GP (mucin deleted, Makona strain) bound to antibody F... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: EMDB / ID: EMD-22853 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | Ebola virus GP (mucin deleted, Makona strain) bound to antibody Fab EBOV-437 and EBOV-515 | |||||||||
 Map data Map data | Ebola virus GP (mucin deleted, Makona strain) bound to antibody Fab EBOV-437 and EBOV-515 | |||||||||
 Sample Sample |
| |||||||||
 Keywords Keywords | ebolavirus / glycan cap / antibody / broadly neutralizing / filovirus / VIRAL PROTEIN / VIRAL PROTEIN-IMMUNE SYSTEM complex | |||||||||
| Function / homology |  Function and homology information Function and homology informationclathrin-dependent endocytosis of virus by host cell / symbiont-mediated-mediated suppression of host tetherin activity / entry receptor-mediated virion attachment to host cell / symbiont-mediated suppression of host innate immune response / fusion of virus membrane with host endosome membrane / viral envelope / lipid binding / host cell plasma membrane / virion membrane / extracellular region / membrane Similarity search - Function | |||||||||
| Biological species |  Homo sapiens (human) / Homo sapiens (human) /  | |||||||||
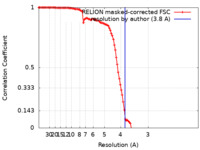
| Method | single particle reconstruction / cryo EM / Resolution: 3.8 Å | |||||||||
 Authors Authors | Murin CD / Ward AB | |||||||||
| Funding support |  United States, 2 items United States, 2 items
| |||||||||
 Citation Citation |  Journal: Cell Rep / Year: 2021 Journal: Cell Rep / Year: 2021Title: Convergence of a common solution for broad ebolavirus neutralization by glycan cap-directed human antibodies. Authors: Charles D Murin / Pavlo Gilchuk / Philipp A Ilinykh / Kai Huang / Natalia Kuzmina / Xiaoli Shen / Jessica F Bruhn / Aubrey L Bryan / Edgar Davidson / Benjamin J Doranz / Lauren E Williamson ...Authors: Charles D Murin / Pavlo Gilchuk / Philipp A Ilinykh / Kai Huang / Natalia Kuzmina / Xiaoli Shen / Jessica F Bruhn / Aubrey L Bryan / Edgar Davidson / Benjamin J Doranz / Lauren E Williamson / Jeffrey Copps / Tanwee Alkutkar / Andrew I Flyak / Alexander Bukreyev / James E Crowe / Andrew B Ward /  Abstract: Antibodies that target the glycan cap epitope on the ebolavirus glycoprotein (GP) are common in the adaptive response of survivors. A subset is known to be broadly neutralizing, but the details of ...Antibodies that target the glycan cap epitope on the ebolavirus glycoprotein (GP) are common in the adaptive response of survivors. A subset is known to be broadly neutralizing, but the details of their epitopes and basis for neutralization are not well understood. Here, we present cryoelectron microscopy (cryo-EM) structures of diverse glycan cap antibodies that variably synergize with GP base-binding antibodies. These structures describe a conserved site of vulnerability that anchors the mucin-like domains (MLDs) to the glycan cap, which we call the MLD anchor and cradle. Antibodies that bind to the MLD cradle share common features, including use of IGHV1-69 and IGHJ6 germline genes, which exploit hydrophobic residues and form β-hairpin structures to mimic the MLD anchor, disrupt MLD attachment, destabilize GP quaternary structure, and block cleavage events required for receptor binding. Our results provide a molecular basis for ebolavirus neutralization by broadly reactive glycan cap antibodies. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Movie |
 Movie viewer Movie viewer |
|---|---|
| Structure viewer | EM map:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| Supplemental images |
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_22853.map.gz emd_22853.map.gz | 8.7 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-22853-v30.xml emd-22853-v30.xml emd-22853.xml emd-22853.xml | 14.3 KB 14.3 KB | Display Display |  EMDB header EMDB header |
| FSC (resolution estimation) |  emd_22853_fsc.xml emd_22853_fsc.xml | 11.4 KB | Display |  FSC data file FSC data file |
| Images |  emd_22853.png emd_22853.png | 72.7 KB | ||
| Filedesc metadata |  emd-22853.cif.gz emd-22853.cif.gz | 6.2 KB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22853 http://ftp.pdbj.org/pub/emdb/structures/EMD-22853 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22853 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22853 | HTTPS FTP |
-Related structure data
| Related structure data |  7kfhMC  7kejC  7kewC  7kexC  7kf9C  7kfbC  7kfeC  7kfgC M: atomic model generated by this map C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| Related items in Molecule of the Month |
- Map
Map
| File |  Download / File: emd_22853.map.gz / Format: CCP4 / Size: 125 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_22853.map.gz / Format: CCP4 / Size: 125 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Ebola virus GP (mucin deleted, Makona strain) bound to antibody Fab EBOV-437 and EBOV-515 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 1.03 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Density |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
CCP4 map header:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-Supplemental data
- Sample components
Sample components
-Entire : Ebola virus GP (mucin deleted, Makona strain) bound to antibody F...
| Entire | Name: Ebola virus GP (mucin deleted, Makona strain) bound to antibody Fab EBOV-437 and EBOV-515 |
|---|---|
| Components |
|
-Supramolecule #1: Ebola virus GP (mucin deleted, Makona strain) bound to antibody F...
| Supramolecule | Name: Ebola virus GP (mucin deleted, Makona strain) bound to antibody Fab EBOV-437 and EBOV-515 type: complex / ID: 1 / Parent: 0 / Macromolecule list: #1-#4 Details: Fab and GP expressed in HEK 293F cells, complexed and purified by size exclusion chromatography |
|---|---|
| Source (natural) | Organism:  Homo sapiens (human) Homo sapiens (human) |
-Macromolecule #1: Virion spike glycoprotein 1
| Macromolecule | Name: Virion spike glycoprotein 1 / type: protein_or_peptide / ID: 1 / Number of copies: 3 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  |
| Molecular weight | Theoretical: 34.978613 KDa |
| Recombinant expression | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Sequence | String: MDAMKRGLCC VLLLCGAVFV SPSQEIHARF RRGARSIPLG VIHNSTLQVS DVDKLVCRDK LSSTNQLRSV GLNLEGNGVA TDVPSVTKR WGFRSGVPPK VVNYEAGEWA ENCYNLEIKK PDGSECLPAA PDGIRGFPRC RYVHKVSGTG PCAGDFAFHK E GAFFLYDR ...String: MDAMKRGLCC VLLLCGAVFV SPSQEIHARF RRGARSIPLG VIHNSTLQVS DVDKLVCRDK LSSTNQLRSV GLNLEGNGVA TDVPSVTKR WGFRSGVPPK VVNYEAGEWA ENCYNLEIKK PDGSECLPAA PDGIRGFPRC RYVHKVSGTG PCAGDFAFHK E GAFFLYDR LASTVIYRGT TFAEGVVAFL ILPQAKKDFF SSHPLREPVN ATEDPSSGYY STTIRYQATG FGTNETEYLF EV DNLTYVQ LESRFTPQFL LQLNETIYAS GKRSNTTGKL IWKVNPEIDT TIGEWAFWET KKNLTRKIRS EELSFT UniProtKB: Envelope glycoprotein |
-Macromolecule #2: Antibody Fab EBOV-437 light chain
| Macromolecule | Name: Antibody Fab EBOV-437 light chain / type: protein_or_peptide / ID: 2 / Number of copies: 3 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Molecular weight | Theoretical: 23.474068 KDa |
| Recombinant expression | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Sequence | String: QIVLTQSPGT LSLSPGERAT LSCRASQSVS NSYLAWYQQK PGQAPRLLIY GASSRATGIP DRFSGSGSGT DFTLSISRLE PEDFAVYYC QQYGTSPCTF GQGTKLEIKR TVAAPSVFIF PPSDEQLKSG TASVVCLLNN FYPREAKVQW KVDNALQSGN S QESVTEQD ...String: QIVLTQSPGT LSLSPGERAT LSCRASQSVS NSYLAWYQQK PGQAPRLLIY GASSRATGIP DRFSGSGSGT DFTLSISRLE PEDFAVYYC QQYGTSPCTF GQGTKLEIKR TVAAPSVFIF PPSDEQLKSG TASVVCLLNN FYPREAKVQW KVDNALQSGN S QESVTEQD SKDSTYSLSS TLTLSKADYE KHKVYACEVT HQGLRSPVTK SFNRGEC |
-Macromolecule #3: Virion spike glycoprotein 2
| Macromolecule | Name: Virion spike glycoprotein 2 / type: protein_or_peptide / ID: 3 / Number of copies: 3 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  |
| Molecular weight | Theoretical: 22.158592 KDa |
| Recombinant expression | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Sequence | String: NNNTHHQDTG EESASSGKLG LITNTIAGVA GLITGGRRTR REVIVNAQPK CNPNLHYWTT QDEGAAIGLA WIPYFGPAAE GIYTEGLMH NQDGLICGLR QLANETTQAL QLFLRATTEL RTFSILNRKA IDFLLQRWGG TCHILGPDCC IEPHDWTKNI T DKIDQIIH ...String: NNNTHHQDTG EESASSGKLG LITNTIAGVA GLITGGRRTR REVIVNAQPK CNPNLHYWTT QDEGAAIGLA WIPYFGPAAE GIYTEGLMH NQDGLICGLR QLANETTQAL QLFLRATTEL RTFSILNRKA IDFLLQRWGG TCHILGPDCC IEPHDWTKNI T DKIDQIIH DDDDKAGWSH PQFEKGGGSG GGSGGGSWSH PQFEK UniProtKB: Envelope glycoprotein |
-Macromolecule #4: Antibody Fab EBOV-437 heavy chain
| Macromolecule | Name: Antibody Fab EBOV-437 heavy chain / type: protein_or_peptide / ID: 4 / Number of copies: 3 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Molecular weight | Theoretical: 25.268438 KDa |
| Recombinant expression | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Sequence | String: QVQLVQSGAE VKKPGSSVKV SCKASGGTFS RDIFSWVRQA PGQGLEWMGG IIPIFGTANY AQKFQGRVTI TADKSTSTVF MELSGLRSE DTAVYFCARG PPLRGERSWF GESEKYDYFY MDVWGKGTTV TVSSASTKGP SVFPLAPSSK STSGGTAALG C LVKDYFPE ...String: QVQLVQSGAE VKKPGSSVKV SCKASGGTFS RDIFSWVRQA PGQGLEWMGG IIPIFGTANY AQKFQGRVTI TADKSTSTVF MELSGLRSE DTAVYFCARG PPLRGERSWF GESEKYDYFY MDVWGKGTTV TVSSASTKGP SVFPLAPSSK STSGGTAALG C LVKDYFPE PVTVSWNSGA LTSGVHTFPA VLQSSGLYSL SSVVTVPSSS LGTQTYICNV NHKPSNTKVD KKVEPKSCD |
-Macromolecule #7: 2-acetamido-2-deoxy-beta-D-glucopyranose
| Macromolecule | Name: 2-acetamido-2-deoxy-beta-D-glucopyranose / type: ligand / ID: 7 / Number of copies: 9 / Formula: NAG |
|---|---|
| Molecular weight | Theoretical: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | single particle reconstruction |
| Aggregation state | particle |
- Sample preparation
Sample preparation
| Buffer | pH: 7.4 |
|---|---|
| Vitrification | Cryogen name: ETHANE |
- Electron microscopy
Electron microscopy
| Microscope | FEI TITAN KRIOS |
|---|---|
| Image recording | Film or detector model: GATAN K2 SUMMIT (4k x 4k) / Average electron dose: 60.0 e/Å2 |
| Electron beam | Acceleration voltage: 300 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | Illumination mode: FLOOD BEAM / Imaging mode: BRIGHT FIELD |
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
 Movie
Movie Controller
Controller


















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)