+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22682 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Plasmodium vivax M17 leucyl aminopeptidase Pv-M17 | |||||||||
 マップデータ マップデータ | Plasmodium vivax M17 leucyl aminopeptidase Pv-M17 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | M17 aminopeptidase / leucyl aminopeptidase / HYDROLASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報leucyl aminopeptidase / metalloaminopeptidase activity / manganese ion binding / proteolysis / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
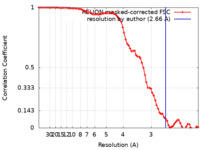
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.66 Å | |||||||||
 データ登録者 データ登録者 | Malcolm TR / McGowan S | |||||||||
| 資金援助 |  オーストラリア, 1件 オーストラリア, 1件
| |||||||||
 引用 引用 |  ジャーナル: J Biol Chem / 年: 2021 ジャーナル: J Biol Chem / 年: 2021タイトル: Active site metals mediate an oligomeric equilibrium in Plasmodium M17 aminopeptidases. 著者: Tess R Malcolm / Matthew J Belousoff / Hariprasad Venugopal / Natalie A Borg / Nyssa Drinkwater / Sarah C Atkinson / Sheena McGowan /  要旨: M17 leucyl aminopeptidases are metal-dependent exopeptidases that rely on oligomerization to diversify their functional roles. The M17 aminopeptidases from Plasmodium falciparum (PfA-M17) and ...M17 leucyl aminopeptidases are metal-dependent exopeptidases that rely on oligomerization to diversify their functional roles. The M17 aminopeptidases from Plasmodium falciparum (PfA-M17) and Plasmodium vivax (Pv-M17) function as catalytically active hexamers to generate free amino acids from human hemoglobin and are drug targets for the design of novel antimalarial agents. However, the molecular basis for oligomeric assembly is not fully understood. In this study, we found that the active site metal ions essential for catalytic activity have a secondary structural role mediating the formation of active hexamers. We found that PfA-M17 and Pv-M17 exist in a metal-dependent dynamic equilibrium between active hexameric species and smaller inactive species that can be controlled by manipulating the identity and concentration of metals available. Mutation of residues involved in metal ion binding impaired catalytic activity and the formation of active hexamers. Structural resolution of Pv-M17 by cryoelectron microscopy and X-ray crystallography together with solution studies revealed that PfA-M17 and Pv-M17 bind metal ions and substrates in a conserved fashion, although Pv-M17 forms the active hexamer more readily and processes substrates faster than PfA-M17. On the basis of these studies, we propose a dynamic equilibrium between monomer ↔ dimer ↔ tetramer ↔ hexamer, which becomes directional toward the large oligomeric states with the addition of metal ions. This sophisticated metal-dependent dynamic equilibrium may apply to other M17 aminopeptidases and underpin the moonlighting capabilities of this enzyme family. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22682.map.gz emd_22682.map.gz | 5.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22682-v30.xml emd-22682-v30.xml emd-22682.xml emd-22682.xml | 20.1 KB 20.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_22682_fsc.xml emd_22682_fsc.xml | 7.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_22682.png emd_22682.png | 66 KB | ||
| マスクデータ |  emd_22682_msk_1.map emd_22682_msk_1.map | 38.4 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-22682.cif.gz emd-22682.cif.gz | 6 KB | ||
| その他 |  emd_22682_additional_1.map.gz emd_22682_additional_1.map.gz emd_22682_additional_2.map.gz emd_22682_additional_2.map.gz emd_22682_half_map_1.map.gz emd_22682_half_map_1.map.gz emd_22682_half_map_2.map.gz emd_22682_half_map_2.map.gz | 25.1 MB 29.4 MB 29.4 MB 29.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22682 http://ftp.pdbj.org/pub/emdb/structures/EMD-22682 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22682 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22682 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_22682_validation.pdf.gz emd_22682_validation.pdf.gz | 821.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_22682_full_validation.pdf.gz emd_22682_full_validation.pdf.gz | 821.5 KB | 表示 | |
| XML形式データ |  emd_22682_validation.xml.gz emd_22682_validation.xml.gz | 13.6 KB | 表示 | |
| CIF形式データ |  emd_22682_validation.cif.gz emd_22682_validation.cif.gz | 19.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22682 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22682 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22682 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22682 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22682.map.gz / 形式: CCP4 / 大きさ: 38.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22682.map.gz / 形式: CCP4 / 大きさ: 38.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Plasmodium vivax M17 leucyl aminopeptidase Pv-M17 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
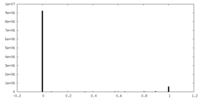
| ファイル |  emd_22682_msk_1.map emd_22682_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Local resolution map
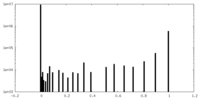
| ファイル | emd_22682_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Local resolution map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Unfiltered map
| ファイル | emd_22682_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unfiltered map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half-volume 1
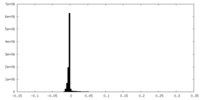
| ファイル | emd_22682_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half-volume 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half-volume 2
| ファイル | emd_22682_half_map_2.map | ||||||||||||
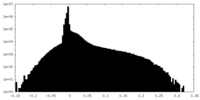
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half-volume 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : PvM17-hexamer
| 全体 | 名称: PvM17-hexamer |
|---|---|
| 要素 |
|
-超分子 #1: PvM17-hexamer
| 超分子 | 名称: PvM17-hexamer / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: M17 leucyl aminopeptidase, putative
| 分子 | 名称: M17 leucyl aminopeptidase, putative / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO / EC番号: leucyl aminopeptidase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 60.442328 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MATTVPQVVS LDPTTIPIDY HTPIDDLSIE VKDISAEACP ADEGLIVFLL NSAPKHSSSG GSGGNGGSAG SSGNGEGGAQ IKINSSVKD NTINEFLKEG NMENFTGKLG TSKSFYIAND QKKYVSLAYV GCGPANEETE LEIRKVAYAL VTLLHDSKHK K VSIIFEIK ...文字列: MATTVPQVVS LDPTTIPIDY HTPIDDLSIE VKDISAEACP ADEGLIVFLL NSAPKHSSSG GSGGNGGSAG SSGNGEGGAQ IKINSSVKD NTINEFLKEG NMENFTGKLG TSKSFYIAND QKKYVSLAYV GCGPANEETE LEIRKVAYAL VTLLHDSKHK K VSIIFEIK IEEALFRFFL EHLFYEYVTD ERFKSADKST ETDFIKNLSL HIANADAYKG QIDKARVYFY GTYYAAQLIA AP SNYCNPV SLSNAAVELA QKVNLECKIL DVKELEELKM GAYLSVGKGS MYPNKFIHLT YKGAQTGASQ NEKKKIALIG KGI TFDSGG YNLKAAPGSM IDLMKFDMSG CAAVLGCAYC IGTIKPDNVE VHFLSAVCEN MVSKNSYRPG DIITASNGKT IEVG NTDAE GRLTLADALV YAEKLGVDYI VDIATLTGAM LYSLGTSYAG VFGNNDQLIN KILSSSKTSN EPVWWLPIIN EYRSS LNSK YADLNNISSS VKASSVVASL FLKEFIENTP WAHIDIAGVS WNFKARKPKG FGVRLLTEFV LNDAVHHHHH H UniProtKB: leucyl aminopeptidase |
-分子 #2: CARBONATE ION
| 分子 | 名称: CARBONATE ION / タイプ: ligand / ID: 2 / コピー数: 6 / 式: CO3 |
|---|---|
| 分子量 | 理論値: 60.009 Da |
| Chemical component information |  ChemComp-CO3: |
-分子 #3: MANGANESE (II) ION
| 分子 | 名称: MANGANESE (II) ION / タイプ: ligand / ID: 3 / コピー数: 12 / 式: MN |
|---|---|
| 分子量 | 理論値: 54.938 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.9 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil / 材質: COPPER / 前処理 - タイプ: GLOW DISCHARGE | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV 詳細: Blot for 3 seconds with blot force of -1. Drain time of 1 second.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 63.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z
Z Y
Y X
X