[English] 日本語
 Yorodumi
Yorodumi- EMDB-22403: cryo-EM structure of human proton-activated chloride channel PAC ... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: EMDB / ID: EMD-22403 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Title | cryo-EM structure of human proton-activated chloride channel PAC at pH 8 | |||||||||||||||
 Map data Map data | Sharpened map | |||||||||||||||
 Sample Sample |
| |||||||||||||||
 Keywords Keywords | PAC / PACC1 / PAORAC / ASOR / TMEM206 / MEMBRANE PROTEIN | |||||||||||||||
| Function / homology | pH-gated chloride channel activity / TMEM206 protein / TMEM206 protein family / chloride transport / chloride channel complex / cell surface / plasma membrane / Proton-activated chloride channel Function and homology information Function and homology information | |||||||||||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | |||||||||||||||
| Method | single particle reconstruction / cryo EM / Resolution: 3.6 Å | |||||||||||||||
 Authors Authors | Lu W / Ruan R | |||||||||||||||
| Funding support |  United States, 4 items United States, 4 items
| |||||||||||||||
 Citation Citation |  Journal: Nature / Year: 2020 Journal: Nature / Year: 2020Title: Structures and pH-sensing mechanism of the proton-activated chloride channel. Authors: Zheng Ruan / James Osei-Owusu / Juan Du / Zhaozhu Qiu / Wei Lü /  Abstract: The proton-activated chloride channel (PAC) is active across a wide range of mammalian cells and is involved in acid-induced cell death and tissue injury. PAC has recently been shown to represent a ...The proton-activated chloride channel (PAC) is active across a wide range of mammalian cells and is involved in acid-induced cell death and tissue injury. PAC has recently been shown to represent a novel and evolutionarily conserved protein family. Here we present two cryo-electron microscopy structures of human PAC in a high-pH resting closed state and a low-pH proton-bound non-conducting state. PAC is a trimer in which each subunit consists of a transmembrane domain (TMD), which is formed of two helices (TM1 and TM2), and an extracellular domain (ECD). Upon a decrease of pH from 8 to 4, we observed marked conformational changes in the ECD-TMD interface and the TMD. The rearrangement of the ECD-TMD interface is characterized by the movement of the histidine 98 residue, which is, after acidification, decoupled from the resting position and inserted into an acidic pocket that is about 5 Å away. Within the TMD, TM1 undergoes a rotational movement, switching its interaction partner from its cognate TM2 to the adjacent TM2. The anion selectivity of PAC is determined by the positively charged lysine 319 residue on TM2, and replacing lysine 319 with a glutamate residue converts PAC to a cation-selective channel. Our data provide a glimpse of the molecular assembly of PAC, and a basis for understanding the mechanism of proton-dependent activation. | |||||||||||||||
| History |
|
- Structure visualization
Structure visualization
| Movie |
 Movie viewer Movie viewer |
|---|---|
| Structure viewer | EM map:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| Supplemental images |
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_22403.map.gz emd_22403.map.gz | 2.5 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-22403-v30.xml emd-22403-v30.xml emd-22403.xml emd-22403.xml | 23.2 KB 23.2 KB | Display Display |  EMDB header EMDB header |
| Images |  emd_22403.png emd_22403.png | 161.6 KB | ||
| Filedesc metadata |  emd-22403.cif.gz emd-22403.cif.gz | 6.6 KB | ||
| Others |  emd_22403_additional_1.map.gz emd_22403_additional_1.map.gz emd_22403_additional_2.map.gz emd_22403_additional_2.map.gz emd_22403_additional_3.map.gz emd_22403_additional_3.map.gz | 22.9 MB 22.9 MB 2.8 MB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22403 http://ftp.pdbj.org/pub/emdb/structures/EMD-22403 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22403 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22403 | HTTPS FTP |
-Related structure data
| Related structure data |  7jnaMC  7jncC M: atomic model generated by this map C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- Map
Map
| File |  Download / File: emd_22403.map.gz / Format: CCP4 / Size: 30.5 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_22403.map.gz / Format: CCP4 / Size: 30.5 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Sharpened map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 1.026 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
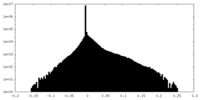
| Density |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
CCP4 map header:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-Supplemental data
-Additional map: Unsharpened map
| File | emd_22403_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Unsharpened map | ||||||||||||
| Projections & Slices |
| ||||||||||||
| Density Histograms |
-Additional map: Unsharpened map obtained using ECD signal
| File | emd_22403_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Unsharpened map obtained using ECD signal | ||||||||||||
| Projections & Slices |
| ||||||||||||
| Density Histograms |
-Additional map: Sharpened map obtained using ECD signal
| File | emd_22403_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Sharpened map obtained using ECD signal | ||||||||||||
| Projections & Slices |
| ||||||||||||
| Density Histograms |
- Sample components
Sample components
-Entire : human proton-activated chloride channel PAC
| Entire | Name: human proton-activated chloride channel PAC |
|---|---|
| Components |
|
-Supramolecule #1: human proton-activated chloride channel PAC
| Supramolecule | Name: human proton-activated chloride channel PAC / type: complex / ID: 1 / Parent: 0 / Macromolecule list: #1 |
|---|---|
| Source (natural) | Organism:  Homo sapiens (human) Homo sapiens (human) |
-Macromolecule #1: Proton-activated chloride channel
| Macromolecule | Name: Proton-activated chloride channel / type: protein_or_peptide / ID: 1 / Number of copies: 3 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Molecular weight | Theoretical: 40.092047 KDa |
| Recombinant expression | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Sequence | String: MIRQERSTSY QELSEELVQV VENSELADEQ DKETVRVQGP GILPGLDSES ASSSIRFSKA CLKNVFSVLL IFIYLLLMAV AVFLVYRTI TDFREKLKHP VMSVSYKEVD RYDAPGIALY PGQAQLLSCK HHYEVIPPLT SPGQPGDMNC TTQRINYTDP F SNQTVKSA ...String: MIRQERSTSY QELSEELVQV VENSELADEQ DKETVRVQGP GILPGLDSES ASSSIRFSKA CLKNVFSVLL IFIYLLLMAV AVFLVYRTI TDFREKLKHP VMSVSYKEVD RYDAPGIALY PGQAQLLSCK HHYEVIPPLT SPGQPGDMNC TTQRINYTDP F SNQTVKSA LIVQGPREVK KRELVFLQFR LNKSSEDFSA IDYLLFSSFQ EFLQSPNRVG FMQACESAYS SWKFSGGFRT WV KMSLVKT KEEDGREAVE FRQETSVVNY IDQRPAAKKS AQLFFVVFEW KDPFIQKVQD IVTANPWNTI ALLCGAFLAL FKA AEFAKL SIKWMIKIRK RYLKRRGQAT SHIS UniProtKB: Proton-activated chloride channel |
-Macromolecule #3: 2-acetamido-2-deoxy-beta-D-glucopyranose
| Macromolecule | Name: 2-acetamido-2-deoxy-beta-D-glucopyranose / type: ligand / ID: 3 / Number of copies: 9 / Formula: NAG |
|---|---|
| Molecular weight | Theoretical: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | single particle reconstruction |
| Aggregation state | particle |
- Sample preparation
Sample preparation
| Concentration | 4 mg/mL |
|---|---|
| Buffer | pH: 8 |
| Grid | Pretreatment - Type: PLASMA CLEANING / Details: unspecified |
| Vitrification | Cryogen name: ETHANE |
- Electron microscopy
Electron microscopy
| Microscope | FEI TITAN KRIOS |
|---|---|
| Image recording | Film or detector model: GATAN K2 SUMMIT (4k x 4k) / Detector mode: SUPER-RESOLUTION / Average exposure time: 8.0 sec. / Average electron dose: 49.6 e/Å2 |
| Electron beam | Acceleration voltage: 300 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | Illumination mode: FLOOD BEAM / Imaging mode: BRIGHT FIELD / Nominal defocus max: 1.9000000000000001 µm / Nominal defocus min: 1.2 µm |
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
 Movie
Movie Controller
Controller












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)