+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22402 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human DPP9-CARD8 complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報CARD8 inflammasome complex assembly / NACHT domain binding / Formation of apoptosome / cysteine-type endopeptidase activator activity / inhibition of cysteine-type endopeptidase activity / negative regulation of NLRP3 inflammasome complex assembly / NLRP3 inflammasome complex / CARD domain binding / dipeptidyl-peptidase IV / negative regulation of lipopolysaccharide-mediated signaling pathway ...CARD8 inflammasome complex assembly / NACHT domain binding / Formation of apoptosome / cysteine-type endopeptidase activator activity / inhibition of cysteine-type endopeptidase activity / negative regulation of NLRP3 inflammasome complex assembly / NLRP3 inflammasome complex / CARD domain binding / dipeptidyl-peptidase IV / negative regulation of lipopolysaccharide-mediated signaling pathway / self proteolysis / Regulation of the apoptosome activity / dipeptidyl-peptidase activity / negative regulation of programmed cell death / 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素 / regulation of canonical NF-kappaB signal transduction / negative regulation of interleukin-1 beta production / pattern recognition receptor activity / negative regulation of NF-kappaB transcription factor activity / cell leading edge / pyroptotic inflammatory response / cysteine-type endopeptidase activator activity involved in apoptotic process / positive regulation of cysteine-type endopeptidase activity involved in apoptotic process / antiviral innate immune response / negative regulation of tumor necrosis factor-mediated signaling pathway / aminopeptidase activity / negative regulation of canonical NF-kappaB signal transduction / serine-type peptidase activity / molecular condensate scaffold activity / positive regulation of interleukin-1 beta production / peptidase activity / regulation of apoptotic process / defense response to virus / microtubule / protein homodimerization activity / protein-containing complex / proteolysis / nucleoplasm / identical protein binding / nucleus / cytoplasm / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Sharif H / Hollingsworth LR | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Immunity / 年: 2021 ジャーナル: Immunity / 年: 2021タイトル: Dipeptidyl peptidase 9 sets a threshold for CARD8 inflammasome formation by sequestering its active C-terminal fragment. 著者: Humayun Sharif / L Robert Hollingsworth / Andrew R Griswold / Jeffrey C Hsiao / Qinghui Wang / Daniel A Bachovchin / Hao Wu /  要旨: CARD8 detects intracellular danger signals and forms a caspase-1 activating inflammasome. Like the related inflammasome sensor NLRP1, CARD8 autoprocesses into noncovalently associated N-terminal (NT) ...CARD8 detects intracellular danger signals and forms a caspase-1 activating inflammasome. Like the related inflammasome sensor NLRP1, CARD8 autoprocesses into noncovalently associated N-terminal (NT) and C-terminal (CT) fragments and binds the cellular dipeptidyl peptidases DPP8 and 9 (DPP8/9). Certain danger-associated signals, including the DPP8/9 inhibitor Val-boroPro (VbP) and HIV protease, induce proteasome-mediated NT degradation and thereby liberate the inflammasome-forming CT. Here, we report cryoelectron microscopy (cryo-EM) structures of CARD8 bound to DPP9, revealing a repressive ternary complex consisting of DPP9, full-length CARD8, and CARD8-CT. Unlike NLRP1-CT, CARD8-CT does not interact with the DPP8/9 active site and is not directly displaced by VbP. However, larger DPP8/9 active-site probes can directly weaken this complex in vitro, and VbP itself nevertheless appears to disrupt this complex, perhaps indirectly, in cells. Thus, DPP8/9 inhibitors can activate the CARD8 inflammasome by promoting CARD8 NT degradation and by weakening ternary complex stability. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22402.map.gz emd_22402.map.gz | 15.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22402-v30.xml emd-22402-v30.xml emd-22402.xml emd-22402.xml | 16.2 KB 16.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_22402_fsc.xml emd_22402_fsc.xml | 14.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_22402.png emd_22402.png | 167.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22402 http://ftp.pdbj.org/pub/emdb/structures/EMD-22402 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22402 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22402 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_22402_validation.pdf.gz emd_22402_validation.pdf.gz | 354.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_22402_full_validation.pdf.gz emd_22402_full_validation.pdf.gz | 354.1 KB | 表示 | |
| XML形式データ |  emd_22402_validation.xml.gz emd_22402_validation.xml.gz | 13.9 KB | 表示 | |
| CIF形式データ |  emd_22402_validation.cif.gz emd_22402_validation.cif.gz | 18.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22402 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22402 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22402 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22402 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7jn7MC  7jkqC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10596 (タイトル: Human DPP9-CARD8 complex-VbP / Data size: 2.1 TB EMPIAR-10596 (タイトル: Human DPP9-CARD8 complex-VbP / Data size: 2.1 TBData #1: Unaligned multi frame micographs of CARD8-DPP9-VbP-noTILT [micrographs - multiframe] Data #2: Unaligned multi frame micographs of CARD8-DPP9-VbP-TILT [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22402.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22402.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.8187 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : DPP9-CARD8 complex
| 全体 | 名称: DPP9-CARD8 complex |
|---|---|
| 要素 |
|
-超分子 #1: DPP9-CARD8 complex
| 超分子 | 名称: DPP9-CARD8 complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換細胞: HEK293 / 組換プラスミド: pcDNA3.1 Homo sapiens (ヒト) / 組換細胞: HEK293 / 組換プラスミド: pcDNA3.1 |
-分子 #1: Dipeptidyl peptidase 9
| 分子 | 名称: Dipeptidyl peptidase 9 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号: dipeptidyl-peptidase IV |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 101.761984 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MSYYHHHHHH DYDIPTTENL YFQGAMGSMA TTGTPTADRG DAAATDDPAA RFQVQKHSWD GLRSIIHGSR KYSGLIVNKA PHDFQFVQK TDESGPHSHR LYYLGMPYGS RENSLLYSEI PKKVRKEALL LLSWKQMLDH FQATPHHGVY SREEELLRER K RLGVFGIT ...文字列: MSYYHHHHHH DYDIPTTENL YFQGAMGSMA TTGTPTADRG DAAATDDPAA RFQVQKHSWD GLRSIIHGSR KYSGLIVNKA PHDFQFVQK TDESGPHSHR LYYLGMPYGS RENSLLYSEI PKKVRKEALL LLSWKQMLDH FQATPHHGVY SREEELLRER K RLGVFGIT SYDFHSESGL FLFQASNSLF HCRDGGKNGF MVSPMKPLEI KTQCSGPRMD PKICPADPAF FSFINNSDLW VA NIETGEE RRLTFCHQGL SNVLDDPKSA GVATFVIQEE FDRFTGYWWC PTASWEGSEG LKTLRILYEE VDESEVEVIH VPS PALEER KTDSYRYPRT GSKNPKIALK LAEFQTDSQG KIVSTQEKEL VQPFSSLFPK VEYIARAGWT RDGKYAWAMF LDRP QQWLQ LVLLPPALFI PSTENEEQRL ASARAVPRNV QPYVVYEEVT NVWINVHDIF YPFPQSEGED ELCFLRANEC KTGFC HLYK VTAVLKSQGY DWSEPFSPGE DEFKCPIKEE IALTSGEWEV LARHGSKIWV NEETKLVYFQ GTKDTPLEHH LYVVSY EAA GEIVRLTTPG FSHSCSMSQN FDMFVSHYSS VSTPPCVHVY KLSGPDDDPL HKQPRFWASM MEAASCPPDY VPPEIFH FH TRSDVRLYGM IYKPHALQPG KKHPTVLFVY GGPQVQLVNN SFKGIKYLRL NTLASLGYAV VVIDGRGSCQ RGLRFEGA L KNQMGQVEIE DQVEGLQFVA EKYGFIDLSR VAIHGWSYGG FLSLMGLIHK PQVFKVAIAG APVTVWMAYD TGYTERYMD VPENNQHGYE AGSVALHVEK LPNEPNRLLI LHGFLDENVH FFHTNFLVSQ LIRAGKPYQL QIYPNERHSI RCPESGEHYE VTLLHFLQE YL |
-分子 #2: Caspase recruitment domain-containing protein 8
| 分子 | 名称: Caspase recruitment domain-containing protein 8 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 60.716875 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MEKKECPEKS SSSEEELPRR DSGSSRNIDA SKLIRLQGSR KLLVDNSIRE LQYTKTGIFF QAEACVTNDT VYRELPCVSE TLCDISHFF QEDDETEAEP LLFRAVPECQ LSGGDIPSVS EEQESSEGQD SGDICSEENQ IVSSYASKVC FEIEEDYKNR Q FLGPEGNV ...文字列: MEKKECPEKS SSSEEELPRR DSGSSRNIDA SKLIRLQGSR KLLVDNSIRE LQYTKTGIFF QAEACVTNDT VYRELPCVSE TLCDISHFF QEDDETEAEP LLFRAVPECQ LSGGDIPSVS EEQESSEGQD SGDICSEENQ IVSSYASKVC FEIEEDYKNR Q FLGPEGNV DVELIDKSTN RYSVWFPTAG WYLWSATGLG FLVRDEVTVT IAFGSWSQHL ALDLQHHEQW LVGGPLFDVT AE PEEAVAE IHLPHFISLQ AGEVDVSWFL VAHFKNEGMV LEHPARVEPF YAVLESPSFS LMGILLRIAS GTRLSIPITS NTL IYYHPH PEDIKFHLYL VPSDALLTKA IDDEEDRFHG VRLQTSPPME PLNFGSSYIV SNSANLKVMP KELKLSYRSP GEIQ HFSKF YAGQMKEPIQ LEITEKRHGT LVWDTEVKPV DLQLVAASAP PPFSGAAFVK ENHRQLQARM GDLKGVLDDL QDNEV LTEN EKELVEQEKT RQSKNEALLS MVEKKGDLAL DVLFRSISER DPYLVSYLRQ QNL |
-分子 #3: [(2~{R})-1-[(2~{R})-2-azanyl-3-methyl-butanoyl]pyrrolidin-2-yl]bo...
| 分子 | 名称: [(2~{R})-1-[(2~{R})-2-azanyl-3-methyl-butanoyl]pyrrolidin-2-yl]boronic acid タイプ: ligand / ID: 3 / コピー数: 2 / 式: GK2 |
|---|---|
| 分子量 | 理論値: 214.07 Da |
| Chemical component information | 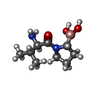 ChemComp-GK2: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 詳細: 25 mM HEPES, pH 7.5, 150 mM NaCl, 1 mM TCEP |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 278 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | #0 - Image recording ID: 1 #0 - フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) #0 - 撮影したグリッド数: 4 / #0 - 実像数: 3306 / #0 - 平均露光時間: 2.22 sec. / #0 - 平均電子線量: 58.5 e/Å2 / #0 - 詳細: stage tilt 0 degrees / #1 - Image recording ID: 2 #1 - フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) #1 - 撮影したグリッド数: 4 / #1 - 実像数: 2488 / #1 - 平均露光時間: 2.25 sec. / #1 - 平均電子線量: 64.99 e/Å2 / #1 - 詳細: stage tilt 37 degrees |
| 電子線 | 加速電圧: 300 kV / 電子線源: OTHER |
| 電子光学系 | 倍率(補正後): 10500 / 照射モード: SPOT SCAN / 撮影モード: OTHER / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): -0.8 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera




















