+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22358 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Connexin-46/50 in a dynamic lipid environment resolved by CryoEM at 1.9 angstroms | |||||||||
 マップデータ マップデータ | Half-map (odd) used for calculation of local resolution-filtered and postprocessed maps. | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報gap junction hemi-channel activity / gap junction-mediated intercellular transport / connexin complex / gap junction channel activity / visual perception / cell-cell signaling / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 1.94 Å | |||||||||
 データ登録者 データ登録者 | Flores JA / Haddad BG / Dolan KD / Myers JB / Yoshioka CC / Copperman J / Zuckerman DM / Reichow SL | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2020 ジャーナル: Nat Commun / 年: 2020タイトル: Connexin-46/50 in a dynamic lipid environment resolved by CryoEM at 1.9 Å. 著者: Jonathan A Flores / Bassam G Haddad / Kimberly A Dolan / Janette B Myers / Craig C Yoshioka / Jeremy Copperman / Daniel M Zuckerman / Steve L Reichow /  要旨: Gap junctions establish direct pathways for cells to transfer metabolic and electrical messages. The local lipid environment is known to affect the structure, stability and intercellular channel ...Gap junctions establish direct pathways for cells to transfer metabolic and electrical messages. The local lipid environment is known to affect the structure, stability and intercellular channel activity of gap junctions; however, the molecular basis for these effects remains unknown. Here, we incorporate native connexin-46/50 (Cx46/50) intercellular channels into a dual lipid nanodisc system, mimicking a native cell-to-cell junction. Structural characterization by CryoEM reveals a lipid-induced stabilization to the channel, resulting in a 3D reconstruction at 1.9 Å resolution. Together with all-atom molecular dynamics simulations, it is shown that Cx46/50 in turn imparts long-range stabilization to the dynamic local lipid environment that is specific to the extracellular lipid leaflet. In addition, ~400 water molecules are resolved in the CryoEM map, localized throughout the intercellular permeation pathway and contributing to the channel architecture. These results illustrate how the aqueous-lipid environment is integrated with the architectural stability, structure and function of gap junction communication channels. #1:  ジャーナル: BioRxiv / 年: 2020 ジャーナル: BioRxiv / 年: 2020タイトル: Connexin-46/50 in a dynamic lipid environment resolved by CryoEM at 1.9 angstroms 著者: Flores JA / Haddad BG / Dolan KA / Myers JB / Yoshioka CC / Copperman J / Zuckerman DM / Reichow SL | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22358.map.gz emd_22358.map.gz | 67.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22358-v30.xml emd-22358-v30.xml emd-22358.xml emd-22358.xml | 25.4 KB 25.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_22358_fsc.xml emd_22358_fsc.xml | 27 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_22358.png emd_22358.png | 206.4 KB | ||
| マスクデータ |  emd_22358_msk_1.map emd_22358_msk_1.map | 103 MB |  マスクマップ マスクマップ | |
| その他 |  emd_22358_additional_1.map.gz emd_22358_additional_1.map.gz emd_22358_additional_2.map.gz emd_22358_additional_2.map.gz emd_22358_additional_3.map.gz emd_22358_additional_3.map.gz emd_22358_half_map_1.map.gz emd_22358_half_map_1.map.gz emd_22358_half_map_2.map.gz emd_22358_half_map_2.map.gz | 94 MB 11.1 MB 93.7 MB 93.8 MB 93.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22358 http://ftp.pdbj.org/pub/emdb/structures/EMD-22358 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22358 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22358 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_22358_validation.pdf.gz emd_22358_validation.pdf.gz | 834.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_22358_full_validation.pdf.gz emd_22358_full_validation.pdf.gz | 833.9 KB | 表示 | |
| XML形式データ |  emd_22358_validation.xml.gz emd_22358_validation.xml.gz | 23.4 KB | 表示 | |
| CIF形式データ |  emd_22358_validation.cif.gz emd_22358_validation.cif.gz | 31.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22358 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22358 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22358 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22358 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7jjpMC  7jkcMC  7jlwC  7jm9C  7jmcC  7jmdC  7jn0C  7jn1C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10480 (タイトル: Connexin-46/50 in a dynamic lipid environment resolved by CryoEM at 1.9 Å EMPIAR-10480 (タイトル: Connexin-46/50 in a dynamic lipid environment resolved by CryoEM at 1.9 ÅData size: 4.1 TB Data #1: Unaligned multiframe micrographs of sheep lens Connexin-46/50 gap junctions embedded in MSP1E1 lipid nanodiscs [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22358.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22358.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map (odd) used for calculation of local resolution-filtered and postprocessed maps. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.649 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
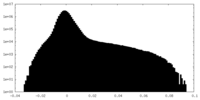
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_22358_msk_1.map emd_22358_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
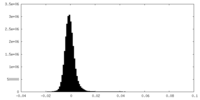
| 密度ヒストグラム |
-追加マップ: Postprocessed map, unmasked.
| ファイル | emd_22358_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Postprocessed map, unmasked. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
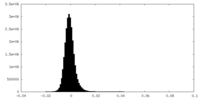
| 密度ヒストグラム |
-追加マップ: Postprocessed map, masked.
| ファイル | emd_22358_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Postprocessed map, masked. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Pre-postprocessed map.
| ファイル | emd_22358_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Pre-postprocessed map. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half-map (even) used for calculation of local resolution-filtered...
| ファイル | emd_22358_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map (even) used for calculation of local resolution-filtered and postprocessed maps. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Postprocessed map, unmasked.
| ファイル | emd_22358_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Postprocessed map, unmasked. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Dodecameric Connexin-50 gap junction channel in MSP1E1-lipid Nanodisc
| 全体 | 名称: Dodecameric Connexin-50 gap junction channel in MSP1E1-lipid Nanodisc |
|---|---|
| 要素 |
|
-超分子 #1: Dodecameric Connexin-50 gap junction channel in MSP1E1-lipid Nanodisc
| 超分子 | 名称: Dodecameric Connexin-50 gap junction channel in MSP1E1-lipid Nanodisc タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 450 KDa |
-分子 #1: Gap junction alpha-8 protein
| 分子 | 名称: Gap junction alpha-8 protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 12 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 49.216809 KDa |
| 配列 | 文字列: MGDWSFLGNI LEEVNEHSTV IGRVWLTVLF IFRILILGTA AEFVWGDEQS DFVCNTQQPG CENVCYDEAF PISHIRLWVL QIIFVSTPS LVYVGHAVHH VRMEEKRKER EAEELSQQSP GNGGERAPLA ADQGSVKKSS SSSKGTKKFR LEGTLLRTYV C HIIFKTLF ...文字列: MGDWSFLGNI LEEVNEHSTV IGRVWLTVLF IFRILILGTA AEFVWGDEQS DFVCNTQQPG CENVCYDEAF PISHIRLWVL QIIFVSTPS LVYVGHAVHH VRMEEKRKER EAEELSQQSP GNGGERAPLA ADQGSVKKSS SSSKGTKKFR LEGTLLRTYV C HIIFKTLF EVGFIVGHYF LYGFRILPLY RCSRWPCPNV VDCFVSRPTE KTIFILFMLS VASVSLFLNI LEMSHLGLKK IR SAFKRPV EQPLGEIPEK SLHSIAVSSI QKAKGYQLLE EEKIVSHYFP LTEVGMVEAS PLSAKPFSQF EEKVGPGPLG DLS RAYQET LPSYAQVGAQ EGVEEEQPIE AAAEPEVGDK SQEAERVSTE GEETLAVLEE EKVEPPEVEK EAEKEETPPE KVSK QELTP EKAPSLCAEL PGEDTRPLSR LSKASSRARS DDLTV |
-分子 #2: 1,2-DIMYRISTOYL-RAC-GLYCERO-3-PHOSPHOCHOLINE
| 分子 | 名称: 1,2-DIMYRISTOYL-RAC-GLYCERO-3-PHOSPHOCHOLINE / タイプ: ligand / ID: 2 / コピー数: 180 / 式: MC3 |
|---|---|
| 分子量 | 理論値: 677.933 Da |
| Chemical component information |  ChemComp-MC3: |
-分子 #3: water
| 分子 | 名称: water / タイプ: ligand / ID: 3 / コピー数: 396 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 1 / 実像数: 2088 / 平均露光時間: 150.0 sec. / 平均電子線量: 52.5 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー












 Z
Z Y
Y X
X