+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22094 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of the holo-SrpI encapsulin complex from Synechococcus elongatus PCC 7942 | |||||||||
 マップデータ マップデータ | Cryo-EM Structure of the holo-Srpl encapsulin complex from Synechococcus elongatus PCC 7942 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | encapsulin / nanocompartment / cysteine desulfurase / HK97-fold / VIRUS LIKE PARTICLE | |||||||||
| 機能・相同性 | Type 2A encapsulin shell protein SrpI-like / : / Type 2A encapsulin shell protein SrpI-like / encapsulin nanocompartment / Type 2A encapsulin shell protein SrpI 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Synechococcus elongatus (strain PCC 7942 / FACHB-805) (バクテリア) Synechococcus elongatus (strain PCC 7942 / FACHB-805) (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.2 Å | |||||||||
 データ登録者 データ登録者 | LaFrance BJ / Nichols RJ / Phillips NR / Oltrogge LM / Valentin-Alvarado LE / Bischoff AJ / Savage DF / Nogales E | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2021 ジャーナル: Elife / 年: 2021タイトル: Discovery and characterization of a novel family of prokaryotic nanocompartments involved in sulfur metabolism. 著者: Robert J Nichols / Benjamin LaFrance / Naiya R Phillips / Devon R Radford / Luke M Oltrogge / Luis E Valentin-Alvarado / Amanda J Bischoff / Eva Nogales / David F Savage /   要旨: Prokaryotic nanocompartments, also known as encapsulins, are a recently discovered proteinaceous organelle-like compartment in prokaryotes that compartmentalize cargo enzymes. While initial studies ...Prokaryotic nanocompartments, also known as encapsulins, are a recently discovered proteinaceous organelle-like compartment in prokaryotes that compartmentalize cargo enzymes. While initial studies have begun to elucidate the structure and physiological roles of encapsulins, bioinformatic evidence suggests that a great diversity of encapsulin nanocompartments remains unexplored. Here, we describe a novel encapsulin in the freshwater cyanobacterium PCC 7942. This nanocompartment is upregulated upon sulfate starvation and encapsulates a cysteine desulfurase enzyme via an N-terminal targeting sequence. Using cryo-electron microscopy, we have determined the structure of the nanocompartment complex to 2.2 Å resolution. Lastly, biochemical characterization of the complex demonstrated that the activity of the cysteine desulfurase is enhanced upon encapsulation. Taken together, our discovery, structural analysis, and enzymatic characterization of this prokaryotic nanocompartment provide a foundation for future studies seeking to understand the physiological role of this encapsulin in various bacteria. #1:  ジャーナル: Biorxiv / 年: 2020 ジャーナル: Biorxiv / 年: 2020タイトル: Discovery and characterization of a novel family of prokaryotic nanocompartments involved in sulfur metabolism 著者: Nichols RJ / LaFrance BJ / Phillips NR / Oltrogge LM / Valentin-Alvarado LE / Bischoff AJ / Nogales E / Savage DF | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22094.map.gz emd_22094.map.gz | 53.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22094-v30.xml emd-22094-v30.xml emd-22094.xml emd-22094.xml | 21 KB 21 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_22094_fsc.xml emd_22094_fsc.xml | 18 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_22094.png emd_22094.png | 200.3 KB | ||
| マスクデータ |  emd_22094_msk_1.map emd_22094_msk_1.map | 512 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-22094.cif.gz emd-22094.cif.gz | 6.5 KB | ||
| その他 |  emd_22094_half_map_1.map.gz emd_22094_half_map_1.map.gz emd_22094_half_map_2.map.gz emd_22094_half_map_2.map.gz | 412.3 MB 412.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22094 http://ftp.pdbj.org/pub/emdb/structures/EMD-22094 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22094 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22094 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_22094_validation.pdf.gz emd_22094_validation.pdf.gz | 165.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_22094_full_validation.pdf.gz emd_22094_full_validation.pdf.gz | 165.2 KB | 表示 | |
| XML形式データ |  emd_22094_validation.xml.gz emd_22094_validation.xml.gz | 497 B | 表示 | |
| CIF形式データ |  emd_22094_validation.cif.gz emd_22094_validation.cif.gz | 373 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22094 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22094 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22094 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22094 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6x8mMC  6x8tC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10506 (タイトル: CryoEM SPA of Holo-SrpI Encapsulin Complex (Raw Frames) EMPIAR-10506 (タイトル: CryoEM SPA of Holo-SrpI Encapsulin Complex (Raw Frames)Data size: 390.3 Data #1: Gain-corrected raw movies for holo-SrpI encapsulin structure (EMD-22094) [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22094.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22094.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM Structure of the holo-Srpl encapsulin complex from Synechococcus elongatus PCC 7942 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8625 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
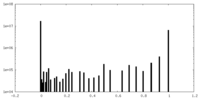
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_22094_msk_1.map emd_22094_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
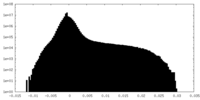
| 密度ヒストグラム |
-ハーフマップ: Even half-map for holo-Srpl encapsulin complex
| ファイル | emd_22094_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Even half-map for holo-Srpl encapsulin complex | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
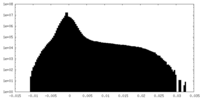
| 密度ヒストグラム |
-ハーフマップ: Odd half-map for holo-Srpl encapsulin complex
| ファイル | emd_22094_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Odd half-map for holo-Srpl encapsulin complex | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : SrpI encapsulin holo complex
| 全体 | 名称: SrpI encapsulin holo complex |
|---|---|
| 要素 |
|
-超分子 #1: SrpI encapsulin holo complex
| 超分子 | 名称: SrpI encapsulin holo complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 分子量 | 理論値: 2.1 MDa |
-超分子 #2: SrpI encapsulin
| 超分子 | 名称: SrpI encapsulin / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Synechococcus elongatus (strain PCC 7942 / FACHB-805) (バクテリア) Synechococcus elongatus (strain PCC 7942 / FACHB-805) (バクテリア) |
-超分子 #3: CyD cargo protein
| 超分子 | 名称: CyD cargo protein / タイプ: complex / ID: 3 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種:  Synechococcus elongatus (strain PCC 7942 / FACHB-805) (バクテリア) Synechococcus elongatus (strain PCC 7942 / FACHB-805) (バクテリア) |
-分子 #1: Protein SrpI
| 分子 | 名称: Protein SrpI / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Synechococcus elongatus (strain PCC 7942 / FACHB-805) (バクテリア) Synechococcus elongatus (strain PCC 7942 / FACHB-805) (バクテリア)株: PCC 7942 / FACHB-805 |
| 分子量 | 理論値: 35.371121 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTDNAPQLAL RDVAARQLAN ATKTVPQLRT ITPRWLVRLL HWTPVEAGIY RVNQVKDASQ ITVACSERDE SELPETFVDY IDNPREYLL SAVNTVVDVH TRISDLYSNP HDQIREQLRL TIEIMKERQE SELINSREYG LLNNVAPGQL VHTRNGAPTP D DLDELLIR ...文字列: MTDNAPQLAL RDVAARQLAN ATKTVPQLRT ITPRWLVRLL HWTPVEAGIY RVNQVKDASQ ITVACSERDE SELPETFVDY IDNPREYLL SAVNTVVDVH TRISDLYSNP HDQIREQLRL TIEIMKERQE SELINSREYG LLNNVAPGQL VHTRNGAPTP D DLDELLIR VWKEPAFFLA HPQAIAAFGR ECTRRGVPPA TVSLFGSSFI TWRGVPLIPS DKVPLENGKT KILLLRVGES RQ GVVGLYQ PNLPGEQGMG LSVRFMGINR KALASYLVSL YCSLAVLTDD ALAVLDNVDV TQYHTYRYNS GHHHHHH UniProtKB: Type 2A encapsulin shell protein SrpI |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.6 mg/mL |
|---|---|
| 緩衝液 | pH: 10 |
| グリッド | モデル: Quantifoil, UltrAuFoil, R1.2/1.3 / 材質: GOLD / メッシュ: 300 詳細: Open hole grids were treated with 2 drops of chloroform, and were not glow discharged before applying sample. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 278 K / 装置: FEI VITROBOT MARK IV 詳細: blot force 5, 3 sec blot, 100 humidity, 4C, 1 sec drain time. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 25 eV |
| ソフトウェア | 名称: SerialEM |
| 詳細 | Residual beam tilt corrected in RELION/3.1. CTF refinement performed in RELION/3.1. |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 撮影したグリッド数: 1 / 実像数: 1027 / 平均電子線量: 35.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.6 mm 最大 デフォーカス(公称値): 2.8000000000000003 µm 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 64000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| ソフトウェア | 名称: PHENIX (ver. dev-2883) |
|---|---|
| 詳細 | One subunit was built completely de novo based on sequence/density, and iteratively real space refined with neighboring subunits using PHENIX. |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 温度因子: 20 / 当てはまり具合の基準: Correlation coefficient |
| 得られたモデル |  PDB-6x8m: |
 ムービー
ムービー コントローラー
コントローラー













 Z
Z Y
Y X
X