+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-21358 | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Type I-F CRISPR-Csy complex with its inhibitor AcrF9 | ||||||||||||||||||
 マップデータ マップデータ | Type I-F CRISPR-Csy complex with its inhibitor AcrF9 | ||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
 キーワード キーワード | CRISPR / Type I-F / Csy / AcrF9 / anti-CRISPR / RNA BINDING PROTEIN-RNA-INHIBITOR complex | ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報maintenance of CRISPR repeat elements / endonuclease activity / defense response to virus / 加水分解酵素; エステル加水分解酵素 / RNA binding 類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |  | ||||||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.57 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Zhang K / Li S | ||||||||||||||||||
| 資金援助 |  米国, 米国,  中国, 5件 中国, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2020 ジャーナル: Proc Natl Acad Sci U S A / 年: 2020タイトル: Inhibition mechanisms of AcrF9, AcrF8, and AcrF6 against type I-F CRISPR-Cas complex revealed by cryo-EM. 著者: Kaiming Zhang / Shuo Wang / Shanshan Li / Yuwei Zhu / Grigore D Pintilie / Tung-Chung Mou / Michael F Schmid / Zhiwei Huang / Wah Chiu /   要旨: Prokaryotes and viruses have fought a long battle against each other. Prokaryotes use CRISPR-Cas-mediated adaptive immunity, while conversely, viruses evolve multiple anti-CRISPR (Acr) proteins to ...Prokaryotes and viruses have fought a long battle against each other. Prokaryotes use CRISPR-Cas-mediated adaptive immunity, while conversely, viruses evolve multiple anti-CRISPR (Acr) proteins to defeat these CRISPR-Cas systems. The type I-F CRISPR-Cas system in requires the crRNA-guided surveillance complex (Csy complex) to recognize the invading DNA. Although some Acr proteins against the Csy complex have been reported, other relevant Acr proteins still need studies to understand their mechanisms. Here, we obtain three structures of previously unresolved Acr proteins (AcrF9, AcrF8, and AcrF6) bound to the Csy complex using electron cryo-microscopy (cryo-EM), with resolution at 2.57 Å, 3.42 Å, and 3.15 Å, respectively. The 2.57-Å structure reveals fine details for each molecular component within the Csy complex as well as the direct and water-mediated interactions between proteins and CRISPR RNA (crRNA). Our structures also show unambiguously how these Acr proteins bind differently to the Csy complex. AcrF9 binds to key DNA-binding sites on the Csy spiral backbone. AcrF6 binds at the junction between Cas7.6f and Cas8f, which is critical for DNA duplex splitting. AcrF8 binds to a distinct position on the Csy spiral backbone and forms interactions with crRNA, which has not been seen in other Acr proteins against the Csy complex. Our structure-guided mutagenesis and biochemistry experiments further support the anti-CRISPR mechanisms of these Acr proteins. Our findings support the convergent consequence of inhibiting degradation of invading DNA by these Acr proteins, albeit with different modes of interactions with the type I-F CRISPR-Cas system. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_21358.map.gz emd_21358.map.gz | 203.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-21358-v30.xml emd-21358-v30.xml emd-21358.xml emd-21358.xml | 24.6 KB 24.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_21358.png emd_21358.png | 210.8 KB | ||
| Filedesc metadata |  emd-21358.cif.gz emd-21358.cif.gz | 6.9 KB | ||
| その他 |  emd_21358_additional.map.gz emd_21358_additional.map.gz emd_21358_half_map_1.map.gz emd_21358_half_map_1.map.gz emd_21358_half_map_2.map.gz emd_21358_half_map_2.map.gz | 108.1 MB 200.6 MB 200.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-21358 http://ftp.pdbj.org/pub/emdb/structures/EMD-21358 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21358 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21358 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_21358_validation.pdf.gz emd_21358_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_21358_full_validation.pdf.gz emd_21358_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_21358_validation.xml.gz emd_21358_validation.xml.gz | 15.2 KB | 表示 | |
| CIF形式データ |  emd_21358_validation.cif.gz emd_21358_validation.cif.gz | 17.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21358 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21358 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21358 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21358 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_21358.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_21358.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Type I-F CRISPR-Csy complex with its inhibitor AcrF9 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.65 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Unmasked map
| ファイル | emd_21358_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unmasked map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
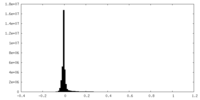
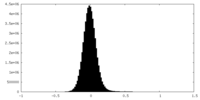
| 密度ヒストグラム |
-ハーフマップ: Half map 1
| ファイル | emd_21358_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
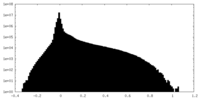
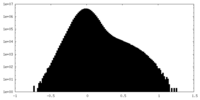
| 密度ヒストグラム |
-ハーフマップ: Half map 2
| ファイル | emd_21358_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
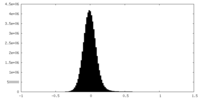
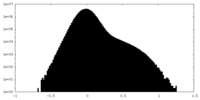
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Type I-F CRISPR-Csy complex with its inhibitor AcrF9
| 全体 | 名称: Type I-F CRISPR-Csy complex with its inhibitor AcrF9 |
|---|---|
| 要素 |
|
-超分子 #1: Type I-F CRISPR-Csy complex with its inhibitor AcrF9
| 超分子 | 名称: Type I-F CRISPR-Csy complex with its inhibitor AcrF9 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 350 KDa |
-分子 #1: AcrF9
| 分子 | 名称: AcrF9 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 7.8139 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKAAYIIKEV QNINSEREGT QIEATSLSQA KRIASKEQCF HGTVMRIETV NGLWLAYKED GKRWVDCQ |
-分子 #2: CRISPR-associated protein Csy1
| 分子 | 名称: CRISPR-associated protein Csy1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 49.194168 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTSPLPTPTW QELRQFIESF IQERLQGKLD KLQPDEDDKR QTLLATHRRE AWLADAARRV GQLQLVTHTL KPIHPDARGS NLHSLPQAP GQPGLAGSHE LGDRLVSDVV GNAAALDVFK FLSLQYQGKN LLNWLTEDSA EALQALSDNA EQAREWRQAF I GITTVKGA ...文字列: MTSPLPTPTW QELRQFIESF IQERLQGKLD KLQPDEDDKR QTLLATHRRE AWLADAARRV GQLQLVTHTL KPIHPDARGS NLHSLPQAP GQPGLAGSHE LGDRLVSDVV GNAAALDVFK FLSLQYQGKN LLNWLTEDSA EALQALSDNA EQAREWRQAF I GITTVKGA PASHSLAKQL YFPLPGSGYH LLAPLFPTSL VHHVHALLRE ARFGDAAKAA REARSRQESW PHGFSEYPNL AI QKFGGTK PQNISQLNNE RRGENWLLPS LPPNWQRQNV NAPMRHSSVF EHDFGRTPEV SRLTRTLQRF LAKTVHNNLA IRQ RRAQLV AQICDEALQY AARLRELEPG WSATPGCQLH DAEQLWLDPL RAQTDETFLQ RRLRGDWPAE VGNRFANWLN RAVS SDSQI LGSPEAAQWS QELSKELTMF KEILEDERD UniProtKB: CRISPR-associated protein Csy1 |
-分子 #3: CRISPR-associated protein Csy2
| 分子 | 名称: CRISPR-associated protein Csy2 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 36.244074 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSVTDPEALL LLPRLSIQNA NAISSPLTWG FPSPGAFTGF VHALQRRVGI SLDIELDGVG IVCHRFEAQI SQPAGKRTKV FNLTRNPLN RDGSTAAIVE EGRAHLEVSL LLGVHGDGLD DHPAQEIARQ VQEQAGAMRL AGGSILPWCN ERFPAPNAEL L MLGGSDEQ ...文字列: MSVTDPEALL LLPRLSIQNA NAISSPLTWG FPSPGAFTGF VHALQRRVGI SLDIELDGVG IVCHRFEAQI SQPAGKRTKV FNLTRNPLN RDGSTAAIVE EGRAHLEVSL LLGVHGDGLD DHPAQEIARQ VQEQAGAMRL AGGSILPWCN ERFPAPNAEL L MLGGSDEQ RRKNQRRLTR RLLPGFALVS REALLQQHLE TLRTTLPEAT TLDALLDLCR INFEPPATSS EEEASPPDAA WQ VRDKPGW LVPIPAGYNA LSPLYLPGEV RNARDRETPL RFVENLFGLG EWLSPHRVAA LSDLLWYHHA EPDKGLYRWS TPR FVEHAI A UniProtKB: Uncharacterized protein |
-分子 #4: CRISPR-associated protein Csy3
| 分子 | 名称: CRISPR-associated protein Csy3 / タイプ: protein_or_peptide / ID: 4 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 39.778594 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKSSHHHHHH ENLYFQSNAS KPILSTASVL AFERKLDPSD ALMSAGAWAQ RDASQEWPAV TVREKSVRGT ISNRLKTKDR DPAKLDASI QSPNLQTVDV ANLPSDADTL KVRFTLRVLG GAGTPSACND AAYRDKLLQT VATYVNDQGF AELARRYAHN L ANARFLWR ...文字列: MKSSHHHHHH ENLYFQSNAS KPILSTASVL AFERKLDPSD ALMSAGAWAQ RDASQEWPAV TVREKSVRGT ISNRLKTKDR DPAKLDASI QSPNLQTVDV ANLPSDADTL KVRFTLRVLG GAGTPSACND AAYRDKLLQT VATYVNDQGF AELARRYAHN L ANARFLWR NRVGAEAVEV RINHIRQGEV ARAWRFDALA IGLRDFKADA ELDALAELIA SGLSGSGHVL LEVVAFARIG DG QEVFPSQ ELILDKGDKK GQKSKTLYSV RDAAAIHSQK IGNALRTIDT WYPDEDGLGP IAVEPYGSVT SQGKAYRQPK QKL DFYTLL DNWVLRDEAP AVEQQHYVIA NLIRGGVFGE AEEK UniProtKB: CRISPR type I-F/YPEST-associated protein Csy3 |
-分子 #5: CRISPR-associated endonuclease Cas6/Csy4
| 分子 | 名称: CRISPR-associated endonuclease Cas6/Csy4 / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO EC番号: 加水分解酵素; エステル加水分解酵素 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 21.427504 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDHYLDIRLR PDPEFPPAQL MSVLFGKLHQ ALVAQGGDRI GVSFPDLDES RSRLGERLRI HASADDLRAL LARPWLEGLR DHLQFGEPA VVPHPTPYRQ VSRVQAKSNP ERLRRRLMRR HDLSEEEARK RIPDTVARAL DLPFVTLRSQ STGQHFRLFI R HGPLQVTA EEGGFTCYGL SKGGFVPWF UniProtKB: CRISPR-associated endonuclease Cas6/Csy4 |
-分子 #6: CrRNA (60-MER)
| 分子 | 名称: CrRNA (60-MER) / タイプ: rna / ID: 6 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 19.249404 KDa |
| 配列 | 文字列: CUAAGAAAUU CACGGCGGGC UUGAUGUCCG CGUCUACCUG GUUCACUGCC GUAUAGGCAG GENBANK: GENBANK: HQ326201.1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.15 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 40 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 実像数: 9267 / 平均露光時間: 6.0 sec. / 平均電子線量: 7.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 2.57 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 3.0.2) / 使用した粒子像数: 332404 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー
















 Z
Z Y
Y X
X