+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20283 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Active State of Manduca sexta soluble Guanylate Cyclase | |||||||||
 マップデータ マップデータ | Map of the Active Ms sGC dimer | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Nitric oxide / cyclase / H-NOX / stimulator / SIGNALING PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報guanylate cyclase / guanylate cyclase complex, soluble / guanylate cyclase activity / response to oxygen levels / cGMP-mediated signaling / heme binding / GTP binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Manduca sexta (蝶・蛾) Manduca sexta (蝶・蛾) | |||||||||
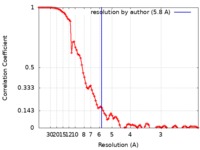
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 5.8 Å | |||||||||
 データ登録者 データ登録者 | Yokom AL / Horst BG | |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2019 ジャーナル: Elife / 年: 2019タイトル: Allosteric activation of the nitric oxide receptor soluble guanylate cyclase mapped by cryo-electron microscopy. 著者: Benjamin G Horst / Adam L Yokom / Daniel J Rosenberg / Kyle L Morris / Michal Hammel / James H Hurley / Michael A Marletta /  要旨: Soluble guanylate cyclase (sGC) is the primary receptor for nitric oxide (NO) in mammalian nitric oxide signaling. We determined structures of full-length sGC in both inactive and active states ...Soluble guanylate cyclase (sGC) is the primary receptor for nitric oxide (NO) in mammalian nitric oxide signaling. We determined structures of full-length sGC in both inactive and active states using cryo-electron microscopy. NO and the sGC-specific stimulator YC-1 induce a 71° rotation of the heme-binding β H-NOX and PAS domains. Repositioning of the β H-NOX domain leads to a straightening of the coiled-coil domains, which, in turn, use the motion to move the catalytic domains into an active conformation. YC-1 binds directly between the β H-NOX domain and the two CC domains. The structural elongation of the particle observed in cryo-EM was corroborated in solution using small angle X-ray scattering (SAXS). These structures delineate the endpoints of the allosteric transition responsible for the major cyclic GMP-dependent physiological effects of NO. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20283.map.gz emd_20283.map.gz | 1.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20283-v30.xml emd-20283-v30.xml emd-20283.xml emd-20283.xml | 15.3 KB 15.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_20283_fsc.xml emd_20283_fsc.xml | 8.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_20283.png emd_20283.png | 36.3 KB | ||
| Filedesc metadata |  emd-20283.cif.gz emd-20283.cif.gz | 5.9 KB | ||
| その他 |  emd_20283_half_map_1.map.gz emd_20283_half_map_1.map.gz emd_20283_half_map_2.map.gz emd_20283_half_map_2.map.gz | 59.4 MB 59.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20283 http://ftp.pdbj.org/pub/emdb/structures/EMD-20283 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20283 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20283 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_20283_validation.pdf.gz emd_20283_validation.pdf.gz | 896.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_20283_full_validation.pdf.gz emd_20283_full_validation.pdf.gz | 896.5 KB | 表示 | |
| XML形式データ |  emd_20283_validation.xml.gz emd_20283_validation.xml.gz | 16.4 KB | 表示 | |
| CIF形式データ |  emd_20283_validation.cif.gz emd_20283_validation.cif.gz | 21.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20283 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20283 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20283 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20283 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20283.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20283.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map of the Active Ms sGC dimer | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.137 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: Half Map 2 of the Active Ms sGC
| ファイル | emd_20283_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
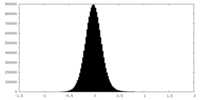
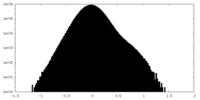
| 注釈 | Half Map 2 of the Active Ms sGC | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half Map 1 of the Active Ms sGC dimer
| ファイル | emd_20283_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
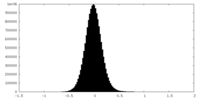
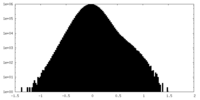
| 注釈 | Half Map 1 of the Active Ms sGC dimer | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Active state of heterodimeric Ms sGC
| 全体 | 名称: Active state of heterodimeric Ms sGC |
|---|---|
| 要素 |
|
-超分子 #1: Active state of heterodimeric Ms sGC
| 超分子 | 名称: Active state of heterodimeric Ms sGC / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Manduca sexta (蝶・蛾) Manduca sexta (蝶・蛾) |
-分子 #1: Soluble guanylyl cyclase alpha-1 subunit
| 分子 | 名称: Soluble guanylyl cyclase alpha-1 subunit / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Manduca sexta (蝶・蛾) Manduca sexta (蝶・蛾) |
| 分子量 | 理論値: 78.598727 KDa |
| 組換発現 | 生物種:  Spodoptera aff. frugiperda 2 RZ-2014 (蝶・蛾) Spodoptera aff. frugiperda 2 RZ-2014 (蝶・蛾) |
| 配列 | 文字列: MTCPFRRASS QHQFANGGSS APKKPEFRSR TSSVHLTGPE EEDGERNTLT LKHMSEALQL LTAPSNECLH AAVTSLTKNQ SDHYHKYNC LRRLPDDVKT CRNYAYLQEI YDAVRATDSV NTKDFMAKLG EYLILTAFSH NCRLERAFKC LGTNLTEFLT T LDSVHDVL ...文字列: MTCPFRRASS QHQFANGGSS APKKPEFRSR TSSVHLTGPE EEDGERNTLT LKHMSEALQL LTAPSNECLH AAVTSLTKNQ SDHYHKYNC LRRLPDDVKT CRNYAYLQEI YDAVRATDSV NTKDFMAKLG EYLILTAFSH NCRLERAFKC LGTNLTEFLT T LDSVHDVL HDQDTPLKDE TMEYEANFVC TTSQEGKIQL HLTTESEPVA YLLVGSLKAI AKRLYDTQTD IRLRSYTNDP RR FRYEINA VPLHQKSKED SCELVNEAAS VATSTKVTDL KIGVASFCKA FPWHFITDKR LELVQLGAGF MRLFGTHLAT HGS SLGTYF RLLRPRGVPL DFREILKRVN TPFMFCLKMP GSTALAEGLE IKGQMVFCAE SDSLLFVGSP FLDGLEGLTG RGLF ISDIP LHDATRDVIL VGEQARAQDG LRRRMDKLKN SIEEASKAVD KEREKNVSLL HLIFPPHIAK RLWLGEKIEA KSHDD VTML FSDIVGFTSI CATATPMMVI AMLEDLYSVF DIFCEELDVY KVETIGDAYC VASGLHRKVE THAPQIAWMA LRMVET CAQ HLTHEGNPIK MRIGLHTGTV LAGVVGKTML KYCLFGHNVT LANKFESGSE PLKINVSPTT YEWLIKFPGF DMEPRDR SC LPNSFPKDIH GTCYFLHKYT HPGTDPGEPQ VKHIREALKD YGIGQANSTD VDTEEPT UniProtKB: guanylate cyclase |
-分子 #2: Soluble guanylyl cyclase beta-1 subunit
| 分子 | 名称: Soluble guanylyl cyclase beta-1 subunit / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Manduca sexta (蝶・蛾) Manduca sexta (蝶・蛾) |
| 分子量 | 理論値: 68.182 KDa |
| 組換発現 | 生物種:  Spodoptera aff. frugiperda 2 RZ-2014 (蝶・蛾) Spodoptera aff. frugiperda 2 RZ-2014 (蝶・蛾) |
| 配列 | 文字列: MYGFVNYALE LLVMKTFDEE TWETIKKKAD VAMEGSFLVR QIYEDEITYN LITAAVEVLQ IPADAILELF GKTFFEFCQD SGYDKILQV LGATPRDFLQ NLDGLHDHLG TLYPGMRSPS FRCTERPEDG ALVLHYYSDR PGLEHIVIGI VKTVASKLHN T EVKVEILK ...文字列: MYGFVNYALE LLVMKTFDEE TWETIKKKAD VAMEGSFLVR QIYEDEITYN LITAAVEVLQ IPADAILELF GKTFFEFCQD SGYDKILQV LGATPRDFLQ NLDGLHDHLG TLYPGMRSPS FRCTERPEDG ALVLHYYSDR PGLEHIVIGI VKTVASKLHN T EVKVEILK TKEECDHVQF LITETSTTGR VSAPEIAEIE TLSLEPKVSP ATFCRVFPFH LMFDRDLNIV QAGRTVSRLL PR VTRPGCK ITDVLDTVRP HLEMTFANVL AHINTVYVLK TKPEEMSVTD PHEEIASLRL KGQMLYIPET DVVVFQCYPS VTN LDDLTR RGLCIADIPL HDATRDLVLM SEQFEADYKL TQNLEVLTDK LQQTFRELEL EKQKTDRLLY SVLPISVATE LRHR RPVPA RRYDTVTLLF SGIVGFANYC ARNSDHKGAM KIVRMLNDLY TAFDVLTDPK RNPNVYKVET VGDKYMAVSG LPEYE VAHA KHISLLALDM MDLSQTVTVD GEPVGITIGI HSGEVVTGVI GHRMPRYCLF GNTVNLTSRC ETTGVPGTIN VSEDTY NYL MREDNHDEQF ELTYRGHVTM KGKAEPMQTW FLTRKIH UniProtKB: Guanylate cyclase soluble subunit beta-1 |
-分子 #3: PROTOPORPHYRIN IX CONTAINING FE
| 分子 | 名称: PROTOPORPHYRIN IX CONTAINING FE / タイプ: ligand / ID: 3 / コピー数: 1 / 式: HEM |
|---|---|
| 分子量 | 理論値: 616.487 Da |
| Chemical component information |  ChemComp-HEM: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 1.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





















 Z
Z Y
Y X
X