+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Baseplate core of bacteriophage JBD30 computed in C3 symmetry | |||||||||
 マップデータ マップデータ | main_map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Bacteriophage JBD30 / virion / baseplate core / baseplate upper protein / distal tail protein / baseplate hub protein / VIRUS | |||||||||
| 機能・相同性 | Bacteriophage phiJL001, Gp84 / Bacteriophage phiJL001, Gp84, C-terminal / Phage conserved hypothetical protein BR0599 / Bacteriophage phiJL001 Gp84 C-terminal domain-containing protein / Uncharacterized protein / Virion structural protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Pseudomonas phage JBD30 (ファージ) Pseudomonas phage JBD30 (ファージ) | |||||||||
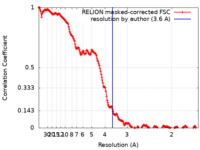
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.6 Å | |||||||||
 データ登録者 データ登録者 | Valentova L / Fuzik T / Plevka P | |||||||||
| 資金援助 |  チェコ, European Union, 2件 チェコ, European Union, 2件
| |||||||||
 引用 引用 |  ジャーナル: Embo J. / 年: 2024 ジャーナル: Embo J. / 年: 2024タイトル: Structure and replication of Pseudomonas aeruginosa phage JBD30 著者: Valentova L / Plevka P / Fuzik T / Novacek J / Pospisil J | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_19262.map.gz emd_19262.map.gz | 31.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-19262-v30.xml emd-19262-v30.xml emd-19262.xml emd-19262.xml | 22.4 KB 22.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_19262_fsc.xml emd_19262_fsc.xml | 19.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_19262.png emd_19262.png | 129 KB | ||
| マスクデータ |  emd_19262_msk_1.map emd_19262_msk_1.map | 600.7 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-19262.cif.gz emd-19262.cif.gz | 7.2 KB | ||
| その他 |  emd_19262_half_map_1.map.gz emd_19262_half_map_1.map.gz emd_19262_half_map_2.map.gz emd_19262_half_map_2.map.gz | 487.5 MB 487.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-19262 http://ftp.pdbj.org/pub/emdb/structures/EMD-19262 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19262 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19262 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8rk6MC  19256  19257  19258  19259  19260  19261  19263  19264  19265  19266  19267  19268  19269  19270  19271  19272  19273  19274  19280  19281  19285  19439  19561  8rk3C  8rk4C  8rk5C  8rk7C  8rk8C  8rk9C  8rkaC  8rkbC  8rkcC  8rknC  8rkoC  8rkxC  8rqeC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_19262.map.gz / 形式: CCP4 / 大きさ: 600.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_19262.map.gz / 形式: CCP4 / 大きさ: 600.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | main_map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8336 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_19262_msk_1.map emd_19262_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Pseudomonas phage JBD30
| 全体 | 名称:  Pseudomonas phage JBD30 (ファージ) Pseudomonas phage JBD30 (ファージ) |
|---|---|
| 要素 |
|
-超分子 #1: Pseudomonas phage JBD30
| 超分子 | 名称: Pseudomonas phage JBD30 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 詳細: Phage JBD30 was propagated in P. aeruginosa strain BAA-28 and purified using CsCl gradient. NCBI-ID: 1223260 / 生物種: Pseudomonas phage JBD30 / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  |
| 分子量 | 理論値: 516 KDa |
| ウイルス殻 | Shell ID: 1 / 名称: JBD30 capsid / 直径: 640.0 Å / T番号(三角分割数): 7 |
-分子 #1: DUF2163 domain-containing protein
| 分子 | 名称: DUF2163 domain-containing protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas phage JBD30 (ファージ) Pseudomonas phage JBD30 (ファージ) |
| 分子量 | 理論値: 29.979053 KDa |
| 配列 | 文字列: MSFNSRESSL ADGQPVRLYQ FSRGAIRWSY NSSDRDITYQ NQIFRTVPGG ITDNGIICSG DPQSDQFVIT APADLDVALL YKTRSPSDA IDLVVYDMHY GDAEAAVSWV GQIGDVDWPT VDSCRITCVS EDELMDQPGL TDTYCRTCTA IVGDHRCKVN L VPYRVTLT ...文字列: MSFNSRESSL ADGQPVRLYQ FSRGAIRWSY NSSDRDITYQ NQIFRTVPGG ITDNGIICSG DPQSDQFVIT APADLDVALL YKTRSPSDA IDLVVYDMHY GDAEAAVSWV GQIGDVDWPT VDSCRITCVS EDELMDQPGL TDTYCRTCTA IVGDHRCKVN L VPYRVTLT PQSVSAWVIS SGVVAGYADG WFTGGYVEWQ VDGDNYDRRF IEQHAGADLH ILGGTEGIPA GGQLRVYPGC DG LAQTCDD KFSNLPNFRG FNAMQGKSPF DGDQVW UniProtKB: Bacteriophage phiJL001 Gp84 C-terminal domain-containing protein |
-分子 #2: Virion structural protein
| 分子 | 名称: Virion structural protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas phage JBD30 (ファージ) Pseudomonas phage JBD30 (ファージ) |
| 分子量 | 理論値: 62.163148 KDa |
| 配列 | 文字列: MATFPGFQVP KPVEGIVAGI TPNIDALELN QDISLAAVAA STWGGAYGAH QPVEVIHSTY QAVHQSALEE NYYNRLWLIP TAMELGNVV STQIRPASVW NAYFSPRTLT AIDREAADGI TLSGQASPPL GFAALEERTW TVSIGTDGPP VVNARIVWRL Q GEPNLVLV ...文字列: MATFPGFQVP KPVEGIVAGI TPNIDALELN QDISLAAVAA STWGGAYGAH QPVEVIHSTY QAVHQSALEE NYYNRLWLIP TAMELGNVV STQIRPASVW NAYFSPRTLT AIDREAADGI TLSGQASPPL GFAALEERTW TVSIGTDGPP VVNARIVWRL Q GEPNLVLV ITGNRIIAWT FAPDWGDSIV ERLSASTNIL QSESAVTQRR AMRLAPRREF DANMYAVDRE RQLLDMTLFG WG ARIWALP IWPDIQLLHQ PLAAGSLGIP CDTAGLDFRD GGLAMLRGED AFTYEVVEVK TVTASGLDLV RPVQAAWGTG SRL YPVRTA QLTEQPTLTR LTDTAQSARV SFLVMEPSAW PELMPATTYR GRPVLEQRPD ESEDLTSSYQ RLLSTLDNGS AIPR VTDVA GMALPVIGHR WIGMGRAERS AFRSLVYALR GQQKPLWVPT HADDLTLVAT VSQLSTALDV RNIGYARFAN GRPGR RDIR IELYDGTVYH RRILTSTELD ADTERVAIDA ALGRLVEPTD VARICFMALC SAASDVVEIE HVTDSEGVAT AALTFK GVR DDEF UniProtKB: Virion structural protein |
-分子 #3: Tip attachment protein J domain-containing protein
| 分子 | 名称: Tip attachment protein J domain-containing protein / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas phage JBD30 (ファージ) Pseudomonas phage JBD30 (ファージ) |
| 分子量 | 理論値: 79.898781 KDa |
| 配列 | 文字列: MGAKPKAQTV GWRYYFDIHF ALGKKVDEVC AIRASGKTAW KGSITSNGQV RINAPELFGG DKGEGGLDGT LDVLFGEEDQ GVLPRLAAM LGGLVPAFRG VTTCFYSGLV TSVNPYPKKW EILRRGGNRL WDGNPWYPEK QFVWLADGQI KAMNPAHILY L VYTGRDFR ...文字列: MGAKPKAQTV GWRYYFDIHF ALGKKVDEVC AIRASGKTAW KGSITSNGQV RINAPELFGG DKGEGGLDGT LDVLFGEEDQ GVLPRLAAM LGGLVPAFRG VTTCFYSGLV TSVNPYPKKW EILRRGGNRL WDGNPWYPEK QFVWLADGQI KAMNPAHILY L VYTGRDFR GLARTRMDEA SWRAAADTLY AEGFGLCFEW TRSDSFKNFC ETVKSHIGAE VYPNRQTGQI SIRLLRDDYN VA DLPLFDE DSGLLEITQE KTGSTSLAPS QLIVKYIDQI DGAQRQIIVN NNAVAASQGR RSSEEIEFLG VPTGELAGRV GER EMRLKT TGLKRYKGVF DRRARSLNPG QPFLIRSTPR GIPETVVRVG RIEDNFLGDG KITLTVVQDQ FNLPATTGVA PPPP GWTPP DRTPRAITVR RLIEAPYREL AGVIDPANLQ LLDVSASYLA ALAEAPTSLS QSYTLTDRVG SSGAFVDRGT GDWCP TGLL AAELPLAAGP NVVTLTNATR LEDVTVGQAA VVDDEIVRVD AVNYASGTVT LARGCADTVP AKHLAGARVW FYDTFE AVD ETVYSQGVTL QARLLTNTSE GQLAPALAAT DSLTLTGRQG KPYPPGQFRI NGSAYPTKVY GALSVSWAKR DRIGQAD QL IDTTVGNIGP EDGATVTLQV YSGTTLKRTY AGLTSSSWSY PLAEDMADGP LQDVRLVLRS VRDGIDSWQQ HDITIERH G LGFRLGEELG GVSA UniProtKB: Uncharacterized protein |
-分子 #4: FE (III) ION
| 分子 | 名称: FE (III) ION / タイプ: ligand / ID: 4 / コピー数: 1 / 式: FE |
|---|---|
| 分子量 | 理論値: 55.845 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 構成要素:
詳細: 10 mM MgSO4, 10 mM NaCl, 50 mM Tris pH 8 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 15 sec. / 前処理 - 雰囲気: OTHER / 詳細: Gatan Solarus II | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV 詳細: blotting force 0, blotting time 2 s, waiting time 15 s. | ||||||||||||
| 詳細 | phage titer 10^11 PFU |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 10 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 1 / 実像数: 12356 / 平均露光時間: 2.0 sec. / 平均電子線量: 34.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.6 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-8rk6: |
 ムービー
ムービー コントローラー
コントローラー




 Z
Z Y
Y X
X