+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Streptococcus pneumoniae NADPH oxidase in complex with NADPH | |||||||||
 マップデータ マップデータ | Main map used for atomic model building and figures. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | NADPH oxidase / ROS producing / flavoprotein / heme protein / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報2 iron, 2 sulfur cluster binding / flavin adenine dinucleotide binding / oxidoreductase activity / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
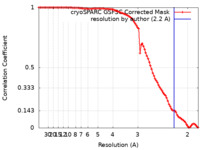
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.2 Å | |||||||||
 データ登録者 データ登録者 | Dubach VRA / San Segundo-Acosta P / Murphy BJ | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published / 年: 2024 ジャーナル: To Be Published / 年: 2024タイトル: Structural and mechanistic insight into Streptococcus pneumoniae NADPH oxidase 著者: Dubach VRA / San Segundo-Acosta P / Murphy BJ | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18645.map.gz emd_18645.map.gz | 79 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18645-v30.xml emd-18645-v30.xml emd-18645.xml emd-18645.xml | 22.9 KB 22.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_18645_fsc.xml emd_18645_fsc.xml | 9.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_18645.png emd_18645.png | 112.7 KB | ||
| マスクデータ |  emd_18645_msk_1.map emd_18645_msk_1.map | 83.7 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-18645.cif.gz emd-18645.cif.gz | 7.2 KB | ||
| その他 |  emd_18645_additional_1.map.gz emd_18645_additional_1.map.gz emd_18645_half_map_1.map.gz emd_18645_half_map_1.map.gz emd_18645_half_map_2.map.gz emd_18645_half_map_2.map.gz | 42.3 MB 77.8 MB 77.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18645 http://ftp.pdbj.org/pub/emdb/structures/EMD-18645 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18645 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18645 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_18645_validation.pdf.gz emd_18645_validation.pdf.gz | 931 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_18645_full_validation.pdf.gz emd_18645_full_validation.pdf.gz | 930.5 KB | 表示 | |
| XML形式データ |  emd_18645_validation.xml.gz emd_18645_validation.xml.gz | 17.3 KB | 表示 | |
| CIF形式データ |  emd_18645_validation.cif.gz emd_18645_validation.cif.gz | 22.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18645 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18645 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18645 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18645 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18645.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18645.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Main map used for atomic model building and figures. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.9168 Å | ||||||||||||||||||||||||||||||||||||
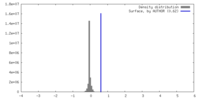
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_18645_msk_1.map emd_18645_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
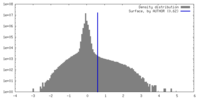
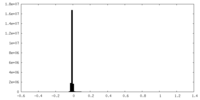
| 密度ヒストグラム |
-追加マップ: Raw unsharpened map obtained in cryoSPARC after merging...
| ファイル | emd_18645_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Raw unsharpened map obtained in cryoSPARC after merging of the half maps. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
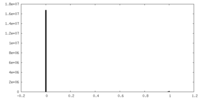
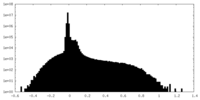
| 密度ヒストグラム |
-ハーフマップ: Half map B used during refinement and gold...
| ファイル | emd_18645_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B used during refinement and gold standard FSC resolution estimation. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
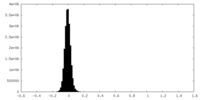
| 密度ヒストグラム |
-ハーフマップ: Half map A used during refinement and gold...
| ファイル | emd_18645_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A used during refinement and gold standard FSC resolution estimation. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
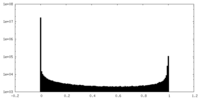
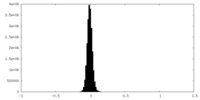
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Streptococcus pneumoniae NADPH oxidase with FAD and heme cofactors
| 全体 | 名称: Streptococcus pneumoniae NADPH oxidase with FAD and heme cofactors |
|---|---|
| 要素 |
|
-超分子 #1: Streptococcus pneumoniae NADPH oxidase with FAD and heme cofactors
| 超分子 | 名称: Streptococcus pneumoniae NADPH oxidase with FAD and heme cofactors タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 46 KDa |
-分子 #1: FAD-binding FR-type domain-containing protein
| 分子 | 名称: FAD-binding FR-type domain-containing protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 46.060551 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: EFSMKSVKGL LFIIASFILT LLTWMNTSPQ FMIPGLALTS LSLTFILATR LPLLESWFHS LEKVYTVHKF TAFLSIILLI FHNFSMGGL WGSRLAAQFG NLAIYIFASI ILVAYLGKYI QYEAWRWIHR LVYLAYILGL FHIYMIMGNR LLTFNLLSFL V GSYALLGL ...文字列: EFSMKSVKGL LFIIASFILT LLTWMNTSPQ FMIPGLALTS LSLTFILATR LPLLESWFHS LEKVYTVHKF TAFLSIILLI FHNFSMGGL WGSRLAAQFG NLAIYIFASI ILVAYLGKYI QYEAWRWIHR LVYLAYILGL FHIYMIMGNR LLTFNLLSFL V GSYALLGL LAGFYIIFLY QKISFPYLGK ITHLKRLNHD TREIQIHLSR PFNYQSGQFA FLKIFQEGFE SAPHPFSISG GH GQTLYFT VKTSGDHTKN IYDNLQAGSK VTLDRAYGHM IIEEGRENQV WIAGGIGITP FISYIREHPI LDKQVHFYYS FRG DENAVY LDLLRNYAQK NPNFELHLID STKDGYLNFE QKEVPEHATV YMCGPISMMK ALAKQIKKQN PKTELIYEGF KFK UniProtKB: FAD-binding FR-type domain-containing protein |
-分子 #2: NADPH DIHYDRO-NICOTINAMIDE-ADENINE-DINUCLEOTIDE PHOSPHATE
| 分子 | 名称: NADPH DIHYDRO-NICOTINAMIDE-ADENINE-DINUCLEOTIDE PHOSPHATE タイプ: ligand / ID: 2 / コピー数: 1 / 式: NDP |
|---|---|
| 分子量 | 理論値: 745.421 Da |
| Chemical component information |  ChemComp-NDP: |
-分子 #3: FLAVIN-ADENINE DINUCLEOTIDE
| 分子 | 名称: FLAVIN-ADENINE DINUCLEOTIDE / タイプ: ligand / ID: 3 / コピー数: 1 / 式: FAD |
|---|---|
| 分子量 | 理論値: 785.55 Da |
| Chemical component information |  ChemComp-FAD: |
-分子 #4: PROTOPORPHYRIN IX CONTAINING FE
| 分子 | 名称: PROTOPORPHYRIN IX CONTAINING FE / タイプ: ligand / ID: 4 / コピー数: 2 / 式: HEM |
|---|---|
| 分子量 | 理論値: 616.487 Da |
| Chemical component information |  ChemComp-HEM: |
-分子 #5: water
| 分子 | 名称: water / タイプ: ligand / ID: 5 / コピー数: 65 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 6 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||||||||
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 400 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 90 sec. | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK III |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: TFS Selectris X / エネルギーフィルター - スリット幅: 10 eV |
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 1 / 実像数: 22259 / 平均露光時間: 3.96 sec. / 平均電子線量: 70.0 e/Å2 / 詳細: Movies were collected in EER format. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.1 µm 最小 デフォーカス(公称値): 0.7000000000000001 µm 倍率(公称値): 215000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: Other / Chain - Initial model type: experimental model 詳細: Initial model was the previously made SpNOX model without substrate bound |
|---|---|
| 詳細 | Initial model was fit using ChimeraX and refined with Coot and PHENIX. |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT 当てはまり具合の基準: Cross-correlation coefficient |
| 得られたモデル |  PDB-8qt7: |
 ムービー
ムービー コントローラー
コントローラー










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)