+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | a membrane-bound menaquinol:organohalide oxidoreductase complex RDH complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | RDH menaquinol:organohalide oxidoreductase / ELECTRON TRANSPORT | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報tetrachloroethene reductive dehalogenase / 4 iron, 4 sulfur cluster binding / oxidoreductase activity / metal ion binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Desulfitobacterium hafniense TCE1 (バクテリア) Desulfitobacterium hafniense TCE1 (バクテリア) | |||||||||
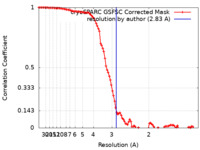
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.83 Å | |||||||||
 データ登録者 データ登録者 | Dongchun N / Ekundayo B / Henning S / Julien M / Holliger C / Cimmino L | |||||||||
| 資金援助 |  スイス, 2件 スイス, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Structure of a membrane-bound menaquinol:organohalide oxidoreductase. 著者: Lorenzo Cimmino / Américo G Duarte / Dongchun Ni / Babatunde E Ekundayo / Inês A C Pereira / Henning Stahlberg / Christof Holliger / Julien Maillard /   要旨: Organohalide-respiring bacteria are key organisms for the bioremediation of soils and aquifers contaminated with halogenated organic compounds. The major players in this process are respiratory ...Organohalide-respiring bacteria are key organisms for the bioremediation of soils and aquifers contaminated with halogenated organic compounds. The major players in this process are respiratory reductive dehalogenases, corrinoid enzymes that use organohalides as substrates and contribute to energy conservation. Here, we present the structure of a menaquinol:organohalide oxidoreductase obtained by cryo-EM. The membrane-bound protein was isolated from Desulfitobacterium hafniense strain TCE1 as a PceAB complex catalysing the dechlorination of tetrachloroethene. Two catalytic PceA subunits are anchored to the membrane by two small integral membrane PceB subunits. The structure reveals two menaquinone molecules bound at the interface of the two different subunits, which are the starting point of a chain of redox cofactors for electron transfer to the active site. In this work, the structure elucidates how energy is conserved during organohalide respiration in menaquinone-dependent organohalide-respiring bacteria. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18148.map.gz emd_18148.map.gz | 97.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18148-v30.xml emd-18148-v30.xml emd-18148.xml emd-18148.xml | 16.7 KB 16.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_18148_fsc.xml emd_18148_fsc.xml | 9.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_18148.png emd_18148.png | 63.9 KB | ||
| Filedesc metadata |  emd-18148.cif.gz emd-18148.cif.gz | 5.8 KB | ||
| その他 |  emd_18148_half_map_1.map.gz emd_18148_half_map_1.map.gz emd_18148_half_map_2.map.gz emd_18148_half_map_2.map.gz | 95.3 MB 95.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18148 http://ftp.pdbj.org/pub/emdb/structures/EMD-18148 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18148 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18148 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_18148_validation.pdf.gz emd_18148_validation.pdf.gz | 189.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_18148_full_validation.pdf.gz emd_18148_full_validation.pdf.gz | 189.4 KB | 表示 | |
| XML形式データ |  emd_18148_validation.xml.gz emd_18148_validation.xml.gz | 502 B | 表示 | |
| CIF形式データ |  emd_18148_validation.cif.gz emd_18148_validation.cif.gz | 373 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18148 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18148 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18148 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18148 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8q4hMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18148.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18148.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.68 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_18148_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_18148_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
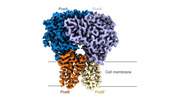
-全体 : a membrane-bound menaquinol:organohalide oxidoreductase
| 全体 | 名称: a membrane-bound menaquinol:organohalide oxidoreductase |
|---|---|
| 要素 |
|
-超分子 #1: a membrane-bound menaquinol:organohalide oxidoreductase
| 超分子 | 名称: a membrane-bound menaquinol:organohalide oxidoreductase タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #2 / 詳細: PceA2B2 |
|---|---|
| 由来(天然) | 生物種:  Desulfitobacterium hafniense TCE1 (バクテリア) Desulfitobacterium hafniense TCE1 (バクテリア) |
| 分子量 | 理論値: 130 kDa/nm |
-分子 #1: Tetrachloroethene reductive dehalogenase
| 分子 | 名称: Tetrachloroethene reductive dehalogenase / タイプ: protein_or_peptide / ID: 1 / 詳細: (COB)(SF4)(SF4)(MQ7) / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Desulfitobacterium hafniense TCE1 (バクテリア) Desulfitobacterium hafniense TCE1 (バクテリア) |
| 分子量 | 理論値: 56.620824 KDa |
| 配列 | 文字列: TSEFPYKVDA KYQRYNSLKN FFEKTFDPEA NKTPIKFHYD DVSKITGKKD TGKDLPTLNA ERLGIKGRPA THTETSILFH TQHLGAMLT QRHNETGWTG LDEALNAGAW AVEFDYSGFN ATGGGPGSVI PLYPINPMTN EIANEPVMVP GLYNWDNIDV E SVRQQGQQ ...文字列: TSEFPYKVDA KYQRYNSLKN FFEKTFDPEA NKTPIKFHYD DVSKITGKKD TGKDLPTLNA ERLGIKGRPA THTETSILFH TQHLGAMLT QRHNETGWTG LDEALNAGAW AVEFDYSGFN ATGGGPGSVI PLYPINPMTN EIANEPVMVP GLYNWDNIDV E SVRQQGQQ WKFESKEEAS KIVKKATRLL GADLVGIAPY DERWTYSTWG RKIYKPCKMP NGRTKYLPWD LPKMLSGGGV EV FGHAKFE PDWEKYAGFK PKSVIVFVLE EDYEAIRTSP SVISSATVGK SYSNMAEVAY KIAVFLRKLG YYAAPCGNDT GIS VPMAVQ AGLGEAGRNG LLITQKFGPR HRIAKVYTDL ELAPDKPRKF GVREFCRLCK KCADACPAQA ISHEKDPKVL QPED CEVAE NPYTEKWHLD SNRCGSFWAY NGSPCSNCVA VCSWNKVETW NHDVARIATQ IPLLQDAARK FDEWFGYNGP VNPDE RLES GYVQNMVKDF WNNPESIKQ UniProtKB: Tetrachloroethene reductive dehalogenase |
-分子 #2: Probable tetrachloroethene reductive dehalogenase membrane anchor...
| 分子 | 名称: Probable tetrachloroethene reductive dehalogenase membrane anchor protein タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Desulfitobacterium hafniense TCE1 (バクテリア) Desulfitobacterium hafniense TCE1 (バクテリア) |
| 分子量 | 理論値: 10.126991 KDa |
| 配列 | 文字列: MNIYDVLIWM ALGMTALLIQ YGIWRYLKGK GKDTIPLQIC GFLANFFFIF ALAWGYSSFS EREYQAIGMG FIFFGGTALI PAIITYRLA UniProtKB: Probable tetrachloroethene reductive dehalogenase membrane anchor protein |
-分子 #3: (~{Z})-1,2-bis(chloranyl)ethene
| 分子 | 名称: (~{Z})-1,2-bis(chloranyl)ethene / タイプ: ligand / ID: 3 / コピー数: 2 / 式: JYF |
|---|---|
| 分子量 | 理論値: 96.943 Da |
| Chemical component information | 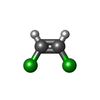 ChemComp-JYF: |
-分子 #4: CO-METHYLCOBALAMIN
| 分子 | 名称: CO-METHYLCOBALAMIN / タイプ: ligand / ID: 4 / コピー数: 2 / 式: COB |
|---|---|
| 分子量 | 理論値: 1.344382 KDa |
| Chemical component information |  ChemComp-COB: |
-分子 #5: IRON/SULFUR CLUSTER
| 分子 | 名称: IRON/SULFUR CLUSTER / タイプ: ligand / ID: 5 / コピー数: 4 / 式: SF4 |
|---|---|
| 分子量 | 理論値: 351.64 Da |
| Chemical component information |  ChemComp-FS1: |
-分子 #6: MENAQUINONE-7
| 分子 | 名称: MENAQUINONE-7 / タイプ: ligand / ID: 6 / コピー数: 2 / 式: MQ7 |
|---|---|
| 分子量 | 理論値: 648.999 Da |
| Chemical component information |  ChemComp-MQ7: |
-分子 #7: water
| 分子 | 名称: water / タイプ: ligand / ID: 7 / コピー数: 14 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー











 Z
Z Y
Y X
X