+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Trimeric prM/E spike of Tick-borne encephalitis virus immature particle | |||||||||
 マップデータ マップデータ | Tick-borne encephalitis virus (strain Neudoerfl) immature particle E/prM spike trimer main map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | immature particle / tick-borne / flavivirus / spike trimer / VIRUS | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報flavivirin / symbiont-mediated suppression of host JAK-STAT cascade via inhibition of STAT2 activity / symbiont-mediated suppression of host JAK-STAT cascade via inhibition of STAT1 activity / double-stranded RNA binding / viral capsid / nucleoside-triphosphate phosphatase / mRNA (guanine-N7)-methyltransferase / methyltransferase cap1 / clathrin-dependent endocytosis of virus by host cell / mRNA (nucleoside-2'-O-)-methyltransferase activity ...flavivirin / symbiont-mediated suppression of host JAK-STAT cascade via inhibition of STAT2 activity / symbiont-mediated suppression of host JAK-STAT cascade via inhibition of STAT1 activity / double-stranded RNA binding / viral capsid / nucleoside-triphosphate phosphatase / mRNA (guanine-N7)-methyltransferase / methyltransferase cap1 / clathrin-dependent endocytosis of virus by host cell / mRNA (nucleoside-2'-O-)-methyltransferase activity / mRNA 5'-cap (guanine-N7-)-methyltransferase activity / RNA helicase activity / host cell endoplasmic reticulum membrane / host cell perinuclear region of cytoplasm / protein dimerization activity / symbiont-mediated suppression of host type I interferon-mediated signaling pathway / RNA helicase / induction by virus of host autophagy / RNA-directed RNA polymerase / viral RNA genome replication / serine-type endopeptidase activity / RNA-dependent RNA polymerase activity / virus-mediated perturbation of host defense response / fusion of virus membrane with host endosome membrane / viral envelope / host cell nucleus / virion attachment to host cell / structural molecule activity / virion membrane / ATP hydrolysis activity / proteolysis / extracellular region / ATP binding / membrane / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Tick-borne encephalitis virus (ダニ媒介脳炎ウイルス) Tick-borne encephalitis virus (ダニ媒介脳炎ウイルス) | |||||||||
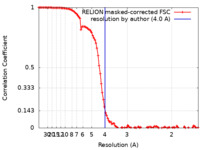
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.0 Å | |||||||||
 データ登録者 データ登録者 | Fuzik T / Plevka P / Smerdova L / Novacek J | |||||||||
| 資金援助 |  チェコ, 2件 チェコ, 2件
| |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2024 ジャーナル: Sci Adv / 年: 2024タイトル: The structure of immature tick-borne encephalitis virus supports the collapse model of flavivirus maturation. 著者: Maria Anastasina / Tibor Füzik / Aušra Domanska / Lauri Ilmari Aurelius Pulkkinen / Lenka Šmerdová / Petra Pokorná Formanová / Petra Straková / Jiří Nováček / Daniel Růžek / ...著者: Maria Anastasina / Tibor Füzik / Aušra Domanska / Lauri Ilmari Aurelius Pulkkinen / Lenka Šmerdová / Petra Pokorná Formanová / Petra Straková / Jiří Nováček / Daniel Růžek / Pavel Plevka / Sarah Jane Butcher /   要旨: We present structures of three immature tick-borne encephalitis virus (TBEV) isolates. Our atomic models of the major viral components, the E and prM proteins, indicate that the pr domains of prM ...We present structures of three immature tick-borne encephalitis virus (TBEV) isolates. Our atomic models of the major viral components, the E and prM proteins, indicate that the pr domains of prM have a critical role in holding the heterohexameric prM3E3 spikes in a metastable conformation. Destabilization of the prM furin-sensitive loop at acidic pH facilitates its processing. The prM topology and domain assignment in TBEV is similar to the mosquito-borne Binjari virus, but is in contrast to other immature flavivirus models. These results support that prM cleavage, the collapse of E protein ectodomains onto the virion surface, the large movement of the membrane domains of both E and M, and the release of the pr fragment from the particle render the virus mature and infectious. Our work favors the collapse model of flavivirus maturation warranting further studies of immature flaviviruses to determine the sequence of events and mechanistic details driving flavivirus maturation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_17947.map.gz emd_17947.map.gz | 7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-17947-v30.xml emd-17947-v30.xml emd-17947.xml emd-17947.xml | 20.5 KB 20.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_17947_fsc.xml emd_17947_fsc.xml | 10.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_17947.png emd_17947.png | 121.1 KB | ||
| マスクデータ |  emd_17947_msk_1.map emd_17947_msk_1.map | 103 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-17947.cif.gz emd-17947.cif.gz | 6.8 KB | ||
| その他 |  emd_17947_half_map_1.map.gz emd_17947_half_map_1.map.gz emd_17947_half_map_2.map.gz emd_17947_half_map_2.map.gz | 80.7 MB 80.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17947 http://ftp.pdbj.org/pub/emdb/structures/EMD-17947 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17947 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17947 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_17947_validation.pdf.gz emd_17947_validation.pdf.gz | 956.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_17947_full_validation.pdf.gz emd_17947_full_validation.pdf.gz | 955.9 KB | 表示 | |
| XML形式データ |  emd_17947_validation.xml.gz emd_17947_validation.xml.gz | 17.1 KB | 表示 | |
| CIF形式データ |  emd_17947_validation.cif.gz emd_17947_validation.cif.gz | 21.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17947 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17947 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17947 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17947 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8puvMC  8ppqC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_17947.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_17947.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Tick-borne encephalitis virus (strain Neudoerfl) immature particle E/prM spike trimer main map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8336 Å | ||||||||||||||||||||||||||||||||||||
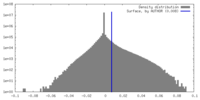
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_17947_msk_1.map emd_17947_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
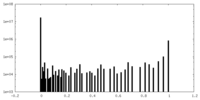
| 密度ヒストグラム |
-ハーフマップ: half map 1 of gold standard reconstruction of...
| ファイル | emd_17947_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 1 of gold standard reconstruction of Tick-borne encephalitis virus (strain Neudoerfl) immature particle E/prM spike trimer | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
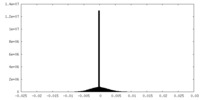
| 密度ヒストグラム |
-ハーフマップ: half map 2 of gold standard reconstruction of...
| ファイル | emd_17947_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2 of gold standard reconstruction of Tick-borne encephalitis virus (strain Neudoerfl) immature particle E/prM spike trimer | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
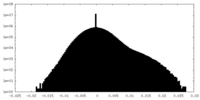
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Tick-borne encephalitis virus
| 全体 | 名称:  Tick-borne encephalitis virus (ダニ媒介脳炎ウイルス) Tick-borne encephalitis virus (ダニ媒介脳炎ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Tick-borne encephalitis virus
| 超分子 | 名称: Tick-borne encephalitis virus / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 詳細: Immature particles produced using tissue cell cultures NCBI-ID: 11084 / 生物種: Tick-borne encephalitis virus / Sci species strain: Neudoerfl / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: Yes / ウイルス・中空状態: No |
|---|---|
| ウイルス殻 | Shell ID: 1 / 名称: immature particle / 直径: 600.0 Å |
-分子 #1: Envelope protein E
| 分子 | 名称: Envelope protein E / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Tick-borne encephalitis virus (ダニ媒介脳炎ウイルス) Tick-borne encephalitis virus (ダニ媒介脳炎ウイルス)株: Neudoerfl |
| 分子量 | 理論値: 53.572297 KDa |
| 配列 | 文字列: SRCTHLENRD FVTGTQGTTR VTLVLELGGC VTITAEGKPS MDVWLDAIYQ ENPAKTREYC LHAKLSDTKV AARCPTMGPA TLAEEHQGG TVCKRDQSDR GWGNHCGLFG KGSIVACVKA ACEAKKKATG HVYDANKIVY TVKVEPHTGD YVAANETHSG R KTASFTIS ...文字列: SRCTHLENRD FVTGTQGTTR VTLVLELGGC VTITAEGKPS MDVWLDAIYQ ENPAKTREYC LHAKLSDTKV AARCPTMGPA TLAEEHQGG TVCKRDQSDR GWGNHCGLFG KGSIVACVKA ACEAKKKATG HVYDANKIVY TVKVEPHTGD YVAANETHSG R KTASFTIS SEKTILTMGE YGDVSLLCRV ASGVDLAQTV ILELDKTVEH LPTAWQVHRD WFNDLALPWK HEGAQNWNNA ER LVEFGAP HAVKMDVYNL GDQTGVLLKA LAGVPVAHIE GTKYHLKSGH VTCEVGLEKL KMKGLTYTMC DKTKFTWKRA PTD SGHDTV VMEVTFSGTK PCRIPVRAVA HGSPDVNVAM LITPNPTIEN NGGGFIEMQL PPGDNIIYVG ELSHQWFQKG SSIG RVFQK TKKGIERLTV IGEHAWDFGS AGGFLSSIGK AVHTVLGGAF NSIFGGVGFL PKLLLGVALA WLGLNMRNPT MSMSF LLAG GLVLAMTLGV GA UniProtKB: Genome polyprotein |
-分子 #2: Protein prM
| 分子 | 名称: Protein prM / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Tick-borne encephalitis virus (ダニ媒介脳炎ウイルス) Tick-borne encephalitis virus (ダニ媒介脳炎ウイルス)株: Neudoerfl |
| 分子量 | 理論値: 18.07667 KDa |
| 配列 | 文字列: TVRKERDGST VIRAEGKDAA TQVRVENGTC VILATDMGSW CDDSLSYECV TIDQGEEPVD VDCFCRNVDG VYLEYGRCGK QEGSRTRRS VLIPSHAQGE LTGRGHKWLE GDSLRTHLTR VEGWVWKNKL LALAMVTVVW LTLESVVTRV AVLVVLLCLA P VYA UniProtKB: Genome polyprotein |
-分子 #3: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 3 / コピー数: 3 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8.5 構成要素:
| ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 280 K / 装置: FEI VITROBOT MARK IV | ||||||||||||
| 詳細 | purified immature particles of Tick-borne encephalitis virus |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 検出モード: INTEGRATING / 撮影したグリッド数: 1 / 実像数: 11246 / 平均露光時間: 2.0 sec. / 平均電子線量: 40.0 e/Å2 詳細: collected in movie mode total 40 fractions per micrograph |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 30.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: AlphaFold / Chain - Initial model type: in silico model |
|---|---|
| 詳細 | inital alhafol generated model was rigidbody fitted into map by Chimera, manually adjusted in Coot and realspace refined in Phenix |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: CC |
| 得られたモデル |  PDB-8puv: |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)