[日本語] English
 万見
万見- EMDB-17794: HK97 small terminase in complex with DNA after focused classification -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | HK97 small terminase in complex with DNA after focused classification | |||||||||
 マップデータ マップデータ | HK97 small terminase | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | small terminase / HK97 / bacteriophage / complex with DNA / DNA BINDING PROTEIN | |||||||||
| 機能・相同性 | Terminase small subunit 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Hendrixvirus (ウイルス) / Hendrixvirus (ウイルス) /  Escherichia phage HK97 (ファージ) / Escherichia phage HK97 (ファージ) /  Byrnievirus HK97 (ウイルス) Byrnievirus HK97 (ウイルス) | |||||||||
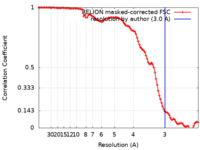
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.0 Å | |||||||||
 データ登録者 データ登録者 | Chechik M / Greive SJ / Antson AA / Jenkins HT | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: bioRxiv / 年: 2023 ジャーナル: bioRxiv / 年: 2023タイトル: Structure of HK97 small terminase:DNA complex unveils a novel DNA binding mechanism by a circular protein. 著者: Maria Chechik / Sandra J Greive / Alfred A Antson / Huw T Jenkins /  要旨: DNA recognition is critical for assembly of double-stranded DNA viruses, in particular for the initiation of packaging the viral genome into the capsid. DNA packaging has been extensively studied for ...DNA recognition is critical for assembly of double-stranded DNA viruses, in particular for the initiation of packaging the viral genome into the capsid. DNA packaging has been extensively studied for three archetypal bacteriophage systems: , and phi29. We identified the minimal site within the region of bacteriophage HK97 specifically recognised by the small terminase and determined a cryoEM structure for the small terminase:DNA complex. This nonameric circular protein utilizes a previously unknown mechanism of DNA binding. While DNA threads through the central tunnel, unexpectedly, DNA-recognition is generated at its exit by a substructure formed by the N- and C-terminal segments of two adjacent protomers of the terminase which are unstructured in the absence of DNA. Such interaction ensures continuous engagement of the small terminase with DNA, allowing sliding along DNA while simultaneously checking the DNA sequence. This mechanism allows locating and instigating packaging initiation and termination precisely at the site. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
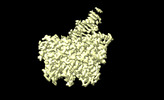
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_17794.map.gz emd_17794.map.gz | 7.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-17794-v30.xml emd-17794-v30.xml emd-17794.xml emd-17794.xml | 19.9 KB 19.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_17794_fsc.xml emd_17794_fsc.xml | 10.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_17794.png emd_17794.png | 36.1 KB | ||
| マスクデータ |  emd_17794_msk_1.map emd_17794_msk_1.map | 8 MB |  マスクマップ マスクマップ | |
| その他 |  emd_17794_half_map_1.map.gz emd_17794_half_map_1.map.gz emd_17794_half_map_2.map.gz emd_17794_half_map_2.map.gz | 7.4 MB 7.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17794 http://ftp.pdbj.org/pub/emdb/structures/EMD-17794 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17794 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17794 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_17794_validation.pdf.gz emd_17794_validation.pdf.gz | 1021.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_17794_full_validation.pdf.gz emd_17794_full_validation.pdf.gz | 1021.2 KB | 表示 | |
| XML形式データ |  emd_17794_validation.xml.gz emd_17794_validation.xml.gz | 13.2 KB | 表示 | |
| CIF形式データ |  emd_17794_validation.cif.gz emd_17794_validation.cif.gz | 17.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17794 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17794 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17794 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17794 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8popMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_17794.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_17794.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | HK97 small terminase | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.18889 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_17794_msk_1.map emd_17794_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
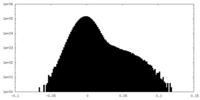
| 密度ヒストグラム |
-ハーフマップ: HK97 small terminase
| ファイル | emd_17794_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | HK97 small terminase | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: HK97 small terminase
| ファイル | emd_17794_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | HK97 small terminase | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : HK97 small terminase in complex with DNA
| 全体 | 名称: HK97 small terminase in complex with DNA |
|---|---|
| 要素 |
|
-超分子 #1: HK97 small terminase in complex with DNA
| 超分子 | 名称: HK97 small terminase in complex with DNA / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Hendrixvirus (ウイルス) Hendrixvirus (ウイルス) |
-分子 #1: Terminase small subunit
| 分子 | 名称: Terminase small subunit / タイプ: protein_or_peptide / ID: 1 / コピー数: 9 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Escherichia phage HK97 (ファージ) Escherichia phage HK97 (ファージ) |
| 分子量 | 理論値: 18.497648 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MADKRIRSDS SAAAVQAMKN AAVDTIDPPS HAGLEKKAEP FWHDNIRSKA LDSWTPADLL AAVELANNQL YITVLRKDLR KEERIRGEE RDEGLIKDLR KQIVELQRTI LAQRRDLQIH SHATNGESRD QKKRNQNDRD ARNTKNEHQD QDDNLIAFPK H G UniProtKB: Terminase small subunit |
-分子 #2: DNA (31-MER)
| 分子 | 名称: DNA (31-MER) / タイプ: dna / ID: 2 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Byrnievirus HK97 (ウイルス) Byrnievirus HK97 (ウイルス) |
| 分子量 | 理論値: 9.600261 KDa |
| 配列 | 文字列: (DT)(DA)(DA)(DA)(DA)(DC)(DT)(DA)(DA)(DA) (DA)(DA)(DA)(DA)(DT)(DC)(DG)(DG)(DG)(DT) (DT)(DA)(DG)(DC)(DG)(DT)(DT)(DA)(DA) (DA)(DT) |
-分子 #3: DNA (31-MER)
| 分子 | 名称: DNA (31-MER) / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Byrnievirus HK97 (ウイルス) Byrnievirus HK97 (ウイルス) |
| 分子量 | 理論値: 9.457115 KDa |
| 配列 | 文字列: (DA)(DT)(DT)(DT)(DA)(DA)(DC)(DG)(DC)(DT) (DA)(DA)(DC)(DC)(DC)(DG)(DA)(DT)(DT)(DT) (DT)(DT)(DT)(DT)(DA)(DG)(DT)(DT)(DT) (DT)(DA) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 180 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.038 kPa | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV | ||||||||||||
| 詳細 | complex was purified on S200 10/30 column |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 2767 / 平均露光時間: 8.0 sec. / 平均電子線量: 53.6 e/Å2 詳細: Two datasets were collected from the same grid. 1st set: 682 images 2nd set: 2085 images |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー



 Z
Z Y
Y X
X