+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Partially dissociated CAND1-CUL1-RBX1-SKP1-SKP2-CKS1-CDK2 | |||||||||||||||
 マップデータ マップデータ | ||||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | CAND1 / substrate receptor exchange factor / cullin-RING ligase / CRL / SCF / ligase / neddylation / DCNL1 co-E3 / ubiquitin signaling | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報SCF complex assembly / positive regulation of protein polyubiquitination / Parkin-FBXW7-Cul1 ubiquitin ligase complex / F-box domain binding / Aberrant regulation of mitotic exit in cancer due to RB1 defects / negative regulation of catalytic activity / PcG protein complex / mitotic cell cycle phase transition / cullin-RING-type E3 NEDD8 transferase / NEDD8 transferase activity ...SCF complex assembly / positive regulation of protein polyubiquitination / Parkin-FBXW7-Cul1 ubiquitin ligase complex / F-box domain binding / Aberrant regulation of mitotic exit in cancer due to RB1 defects / negative regulation of catalytic activity / PcG protein complex / mitotic cell cycle phase transition / cullin-RING-type E3 NEDD8 transferase / NEDD8 transferase activity / cullin-RING ubiquitin ligase complex / cellular response to chemical stress / positive regulation of ubiquitin protein ligase activity / Cul7-RING ubiquitin ligase complex / ubiquitin-dependent protein catabolic process via the C-end degron rule pathway / maintenance of protein location in nucleus / Loss of Function of FBXW7 in Cancer and NOTCH1 Signaling / positive regulation of protein autoubiquitination / protein neddylation / cyclin-dependent protein serine/threonine kinase activator activity / NEDD8 ligase activity / Cul5-RING ubiquitin ligase complex / positive regulation of intracellular estrogen receptor signaling pathway / negative regulation of response to oxidative stress / ubiquitin-ubiquitin ligase activity / SCF ubiquitin ligase complex / Cul2-RING ubiquitin ligase complex / Cul4A-RING E3 ubiquitin ligase complex / negative regulation of type I interferon production / Cul4B-RING E3 ubiquitin ligase complex / SCF-dependent proteasomal ubiquitin-dependent protein catabolic process / Cul3-RING ubiquitin ligase complex / ubiquitin ligase complex scaffold activity / cyclin A1-CDK2 complex / cyclin E2-CDK2 complex / cyclin E1-CDK2 complex / cyclin A2-CDK2 complex / positive regulation of DNA-templated DNA replication initiation / G2 Phase / cyclin-dependent protein kinase activity / Y chromosome / Phosphorylation of proteins involved in G1/S transition by active Cyclin E:Cdk2 complexes / positive regulation of heterochromatin formation / p53-Dependent G1 DNA Damage Response / X chromosome / PTK6 Regulates Cell Cycle / Prolactin receptor signaling / regulation of anaphase-promoting complex-dependent catabolic process / Defective binding of RB1 mutants to E2F1,(E2F2, E2F3) / protein monoubiquitination / centriole replication / Regulation of APC/C activators between G1/S and early anaphase / telomere maintenance in response to DNA damage / centrosome duplication / cullin family protein binding / protein K63-linked ubiquitination / Telomere Extension By Telomerase / G0 and Early G1 / positive regulation of RNA polymerase II transcription preinitiation complex assembly / cellular response to nitric oxide / Activation of the pre-replicative complex / ubiquitin-like ligase-substrate adaptor activity / positive regulation of double-strand break repair via homologous recombination / cyclin-dependent kinase / cyclin-dependent protein serine/threonine kinase activity / TP53 Regulates Transcription of Genes Involved in G1 Cell Cycle Arrest / protein K48-linked ubiquitination / Regulation of MITF-M-dependent genes involved in cell cycle and proliferation / Activation of ATR in response to replication stress / Cyclin E associated events during G1/S transition / Cajal body / Nuclear events stimulated by ALK signaling in cancer / Cyclin A/B1/B2 associated events during G2/M transition / cyclin-dependent protein kinase holoenzyme complex / Cyclin A:Cdk2-associated events at S phase entry / ubiquitin ligase complex / condensed chromosome / mitotic G1 DNA damage checkpoint signaling / regulation of G2/M transition of mitotic cell cycle / regulation of mitotic cell cycle / TBP-class protein binding / positive regulation of TORC1 signaling / post-translational protein modification / regulation of cellular response to insulin stimulus / cyclin binding / intrinsic apoptotic signaling pathway / Regulation of BACH1 activity / MAP3K8 (TPL2)-dependent MAPK1/3 activation / T cell activation / positive regulation of DNA replication / male germ cell nucleus / ubiquitin binding / molecular function activator activity / meiotic cell cycle / SCF-beta-TrCP mediated degradation of Emi1 / NIK-->noncanonical NF-kB signaling / Vpu mediated degradation of CD4 / Degradation of DVL / Dectin-1 mediated noncanonical NF-kB signaling / animal organ morphogenesis 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||
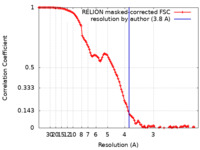
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||||||||
 データ登録者 データ登録者 | Shaaban M / Clapperton JA / Ding S / Maeots ME / Enchev RI / Maslen SL / Skehel JM | |||||||||||||||
| 資金援助 |  英国, 4件 英国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2023 ジャーナル: Mol Cell / 年: 2023タイトル: Structural and mechanistic insights into the CAND1-mediated SCF substrate receptor exchange. 著者: Mohammed Shaaban / Julie A Clapperton / Shan Ding / Simone Kunzelmann / Märt-Erik Mäeots / Sarah L Maslen / J Mark Skehel / Radoslav I Enchev /  要旨: Modular SCF (SKP1-CUL1-Fbox) ubiquitin E3 ligases orchestrate multiple cellular pathways in eukaryotes. Their variable SKP1-Fbox substrate receptor (SR) modules enable regulated substrate recruitment ...Modular SCF (SKP1-CUL1-Fbox) ubiquitin E3 ligases orchestrate multiple cellular pathways in eukaryotes. Their variable SKP1-Fbox substrate receptor (SR) modules enable regulated substrate recruitment and subsequent proteasomal degradation. CAND proteins are essential for the efficient and timely exchange of SRs. To gain structural understanding of the underlying molecular mechanism, we reconstituted a human CAND1-driven exchange reaction of substrate-bound SCF alongside its co-E3 ligase DCNL1 and visualized it by cryo-EM. We describe high-resolution structural intermediates, including a ternary CAND1-SCF complex, as well as conformational and compositional intermediates representing SR- or CAND1-dissociation. We describe in molecular detail how CAND1-induced conformational changes in CUL1/RBX1 provide an optimized DCNL1-binding site and reveal an unexpected dual role for DCNL1 in CAND1-SCF dynamics. Moreover, a partially dissociated CAND1-SCF conformation accommodates cullin neddylation, leading to CAND1 displacement. Our structural findings, together with functional biochemical assays, help formulate a detailed model for CAND-SCF regulation. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_17117.map.gz emd_17117.map.gz | 111.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-17117-v30.xml emd-17117-v30.xml emd-17117.xml emd-17117.xml | 24.6 KB 24.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_17117_fsc.xml emd_17117_fsc.xml | 11.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_17117.png emd_17117.png | 124.5 KB | ||
| Filedesc metadata |  emd-17117.cif.gz emd-17117.cif.gz | 8 KB | ||
| その他 |  emd_17117_half_map_1.map.gz emd_17117_half_map_1.map.gz emd_17117_half_map_2.map.gz emd_17117_half_map_2.map.gz | 98.2 MB 98.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17117 http://ftp.pdbj.org/pub/emdb/structures/EMD-17117 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17117 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17117 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_17117_validation.pdf.gz emd_17117_validation.pdf.gz | 680.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_17117_full_validation.pdf.gz emd_17117_full_validation.pdf.gz | 680.4 KB | 表示 | |
| XML形式データ |  emd_17117_validation.xml.gz emd_17117_validation.xml.gz | 18.7 KB | 表示 | |
| CIF形式データ |  emd_17117_validation.cif.gz emd_17117_validation.cif.gz | 24.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17117 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17117 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17117 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17117 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_17117.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_17117.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||||||||||||||||||
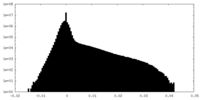
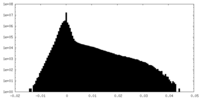
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_17117_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
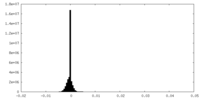
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_17117_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
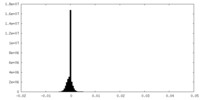
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Partially dissociated CAND1-CUL1-RBX1-SKP1-SKP2-CKS1-CDK2
| 全体 | 名称: Partially dissociated CAND1-CUL1-RBX1-SKP1-SKP2-CKS1-CDK2 |
|---|---|
| 要素 |
|
-超分子 #1: Partially dissociated CAND1-CUL1-RBX1-SKP1-SKP2-CKS1-CDK2
| 超分子 | 名称: Partially dissociated CAND1-CUL1-RBX1-SKP1-SKP2-CKS1-CDK2 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Cullin-1
| 分子 | 名称: Cullin-1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 93.730672 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MWSHPQFEKG SAGSAAGSGA GWSHPQFEKL EVLFQGPMSS TRSQNPHGLK QIGLDQIWDD LRAGIQQVYT RQSMAKSRYM ELYTHVYNY CTSVHQSNQA RGAGVPPSKS KKGQTPGGAQ FVGLELYKRL KEFLKNYLTN LLKDGEDLMD ESVLKFYTQQ W EDYRFSSK ...文字列: MWSHPQFEKG SAGSAAGSGA GWSHPQFEKL EVLFQGPMSS TRSQNPHGLK QIGLDQIWDD LRAGIQQVYT RQSMAKSRYM ELYTHVYNY CTSVHQSNQA RGAGVPPSKS KKGQTPGGAQ FVGLELYKRL KEFLKNYLTN LLKDGEDLMD ESVLKFYTQQ W EDYRFSSK VLNGICAYLN RHWVRRECDE GRKGIYEIYS LALVTWRDCL FRPLNKQVTN AVLKLIEKER NGETINTRLI SG VVQSYVE LGLNEDDAFA KGPTLTVYKE SFESQFLADT ERFYTRESTE FLQQNPVTEY MKKAEARLLE EQRRVQVYLH EST QDELAR KCEQVLIEKH LEIFHTEFQN LLDADKNEDL GRMYNLVSRI QDGLGELKKL LETHIHNQGL AAIEKCGEAA LNDP KMYVQ TVLDVHKKYN ALVMSAFNND AGFVAALDKA CGRFINNNAV TKMAQSSSKS PELLARYCDS LLKKSSKNPE EAELE DTLN QVMVVFKYIE DKDVFQKFYA KMLAKRLVHQ NSASDDAEAS MISKLKQACG FEYTSKLQRM FQDIGVSKDL NEQFKK HLT NSEPLDLDFS IQVLSSGSWP FQQSCTFALP SELERSYQRF TAFYASRHSG RKLTWLYQLS KGELVTNCFK NRYTLQA ST FQMAILLQYN TEDAYTVQQL TDSTQIKMDI LAQVLQILLK SKLLVLEDEN ANVDEVELKP DTLIKLYLGY KNKKLRVN I NVPMKTEQKQ EQETTHKNIE EDRKLLIQAA IVRIMKMRKV LKHQQLLGEV LTQLSSRFKP RVPVIKKCID ILIEKEYLE RVDGEKDTYS YLA UniProtKB: Cullin-1 |
-分子 #2: E3 ubiquitin-protein ligase RBX1
| 分子 | 名称: E3 ubiquitin-protein ligase RBX1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO / EC番号: RING-type E3 ubiquitin transferase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 12.289977 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAAAMDVDTP SGTNSGAGKK RFEVKKWNAV ALWAWDIVVD NCAICRNHIM DLCIECQANQ ASATSEECTV AWGVCNHAFH FHCISRWLK TRQVCPLDNR EWEFQKYGH UniProtKB: E3 ubiquitin-protein ligase RBX1 |
-分子 #3: Cullin-associated NEDD8-dissociated protein 1
| 分子 | 名称: Cullin-associated NEDD8-dissociated protein 1 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 137.358219 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GSPEFPGRMA SASYHISNLL EKMTSSDKDF RFMATNDLMT ELQKDSIKLD DDSERKVVKM ILKLLEDKNG EVQNLAVKCL GPLVSKVKE YQVETIVDTL CTNMLSDKEQ LRDISSIGLK TVIGELPPAS SGSALAANVC KKITGRLTSA IAKQEDVSVQ L EALDIMAD ...文字列: GSPEFPGRMA SASYHISNLL EKMTSSDKDF RFMATNDLMT ELQKDSIKLD DDSERKVVKM ILKLLEDKNG EVQNLAVKCL GPLVSKVKE YQVETIVDTL CTNMLSDKEQ LRDISSIGLK TVIGELPPAS SGSALAANVC KKITGRLTSA IAKQEDVSVQ L EALDIMAD MLSRQGGLLV NFHPSILTCL LPQLTSPRLA VRKRTIIALG HLVMSCGNIV FVDLIEHLLS ELSKNDSMST TR TYIQCIA AISRQAGHRI GEYLEKIIPL VVKFCNVDDD ELREYCIQAF ESFVRRCPKE VYPHVSTIIN ICLKYLTYDP NYN YDDEDE DENAMDADGG DDDDQGSDDE YSDDDDMSWK VRRAAAKCLD AVVSTRHEML PEFYKTVSPA LISRFKEREE NVKA DVFHA YLSLLKQTRP VQSWLCDPDA MEQGETPLTM LQSQVPNIVK ALHKQMKEKS VKTRQCCFNM LTELVNVLPG ALTQH IPVL VPGIIFSLND KSSSSNLKID ALSCLYVILC NHSPQVFHPH VQALVPPVVA CVGDPFYKIT SEALLVTQQL VKVIRP LDQ PSSFDATPYI KDLFTCTIKR LKAADIDQEV KERAISCMGQ IICNLGDNLG SDLPNTLQIF LERLKNEITR LTTVKAL TL IAGSPLKIDL RPVLGEGVPI LASFLRKNQR ALKLGTLSAL DILIKNYSDS LTAAMIDAVL DELPPLISES DMHVSQMA I SFLTTLAKVY PSSLSKISGS ILNELIGLVR SPLLQGGALS AMLDFFQALV VTGTNNLGYM DLLRMLTGPV YSQSTALTH KQSYYSIAKC VAALTRACPK EGPAVVGQFI QDVKNSRSTD SIRLLALLSL GEVGHHIDLS GQLELKSVIL EAFSSPSEEV KSAASYALG SISVGNLPEY LPFVLQEITS QPKRQYLLLH SLKEIISSAS VVGLKPYVEN IWALLLKHCE CAEEGTRNVV A ECLGKLTL IDPETLLPRL KGYLISGSSY ARSSVVTAVK FTISDHPQPI DPLLKNCIGD FLKTLEDPDL NVRRVALVTF NS AAHNKPS LIRDLLDTVL PHLYNETKVR KELIREVEMG PFKHTVDDGL DIRKAAFECM YTLLDSCLDR LDIFEFLNHV EDG LKDHYD IKMLTFLMLV RLSTLCPSAV LQRLDRLVEP LRATCTTKVK ANSVKQEFEK QDELKRSAMR AVAALLTIPE AEKS PLMSE FQSQISSNPE LAAIFESIQK DSSSTNLESM DTS UniProtKB: Cullin-associated NEDD8-dissociated protein 1 |
-分子 #4: S-phase kinase-associated protein 1
| 分子 | 名称: S-phase kinase-associated protein 1 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 18.679965 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MPSIKLQSSD GEIFEVDVEI AKQSVTIKTM LEDLGMDDEG DDDPVPLPNV NAAILKKVIQ WCTHHKDDPP PPEDDENKEK RTDDIPVWD QEFLKVDQGT LFELILAANY LDIKGLLDVT CKTVANMIKG KTPEEIRKTF NIKNDFTEEE EAQVRKENQW C EEK UniProtKB: S-phase kinase-associated protein 1 |
-分子 #5: S-phase kinase-associated protein 2
| 分子 | 名称: S-phase kinase-associated protein 2 / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 48.335312 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GSPEFMHRKH LQEIPDLSSN VATSFTWGWD SSKTSELLSG MGVSALEKEE PDSENIPQEL LSNLGHPESP PRKRLKSKGS DKDFVIVRR PKLNRENFPG VSWDSLPDEL LLGIFSCLCL PELLKVSGVC KRWYRLASDE SLWQTLDLTG KNLHPDVTGR L LSQGVIAF ...文字列: GSPEFMHRKH LQEIPDLSSN VATSFTWGWD SSKTSELLSG MGVSALEKEE PDSENIPQEL LSNLGHPESP PRKRLKSKGS DKDFVIVRR PKLNRENFPG VSWDSLPDEL LLGIFSCLCL PELLKVSGVC KRWYRLASDE SLWQTLDLTG KNLHPDVTGR L LSQGVIAF RCPRSFMDQP LAEHFSPFRV QHMDLSNSVI EVSTLHGILS QCSKLQNLSL EGLRLSDPIV NTLAKNSNLV RL NLSGCSG FSEFALQTLL SSCSRLDELN LSWCFDFTEK HVQVAVAHVS ETITQLNLSG YRKNLQKSDL STLVRRCPNL VHL DLSDSV MLKNDCFQEF FQLNYLQHLS LSRCYDIIPE TLLELGEIPT LKTLQVFGIV PDGTLQLLKE ALPHLQINCS HFTT IARPT IGNKKNQEIW GIKCRLTLQK PSCL UniProtKB: S-phase kinase-associated protein 2 |
-分子 #6: Cyclin-dependent kinases regulatory subunit 1
| 分子 | 名称: Cyclin-dependent kinases regulatory subunit 1 / タイプ: protein_or_peptide / ID: 6 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 8.894114 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GPLGSQIYYS DKYDDEEFEY RHVMLPKDIA KLVPKTHLMS ESEWRNLGVQ QSQGWVHYMI HEPEPHILLF RRPL UniProtKB: Cyclin-dependent kinases regulatory subunit 1 |
-分子 #7: Cyclin-dependent kinase 2
| 分子 | 名称: Cyclin-dependent kinase 2 / タイプ: protein_or_peptide / ID: 7 / コピー数: 1 / 光学異性体: LEVO / EC番号: cyclin-dependent kinase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 33.976488 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MENFQKVEKI GEGTYGVVYK ARNKLTGEVV ALKKIRLDTE TEGVPSTAIR EISLLKELNH PNIVKLLDVI HTENKLYLVF EFLHQDLKK FMDASALTGI PLPLIKSYLF QLLQGLAFCH SHRVLHRDLK PQNLLINTEG AIKLADFGLA RAFGVPVRTY T HEVVTLWY ...文字列: MENFQKVEKI GEGTYGVVYK ARNKLTGEVV ALKKIRLDTE TEGVPSTAIR EISLLKELNH PNIVKLLDVI HTENKLYLVF EFLHQDLKK FMDASALTGI PLPLIKSYLF QLLQGLAFCH SHRVLHRDLK PQNLLINTEG AIKLADFGLA RAFGVPVRTY T HEVVTLWY RAPEILLGCK YYSTAVDIWS LGCIFAEMVT RRALFPGDSE IDQLFRIFRT LGTPDEVVWP GVTSMPDYKP SF PKWARQD FSKVVPPLDE DGRSLLSQML HYDPNKRISA KAALAHPFFQ DVTKPVPHLR L UniProtKB: Cyclin-dependent kinase 2 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV | ||||||||||
| 詳細 | CAND1-CUL1-RBX1-SKP1-SKP2-CKS1-CDK2-CyclinE-DCNL1 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 49.1 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm 最小 デフォーカス(公称値): 0.7000000000000001 µm 倍率(公称値): 130000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)