+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the proximal end of bacteriophage T5 tail : p142 tail terminator protein hexamer and pb6 tail tube protein trimer | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Bacteriophage / Siphophage / T5 / tail terminator / Trp / VIRAL PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報virus tail, tube / symbiont genome ejection through host cell envelope, long flexible tail mechanism / viral tail assembly / virus tail / viral release from host cell by cytolysis 類似検索 - 分子機能 | |||||||||
| 生物種 |  Escherichia phage T5 (ファージ) / Escherichia phage T5 (ファージ) /  Escherichia virus T5 (ウイルス) Escherichia virus T5 (ウイルス) | |||||||||
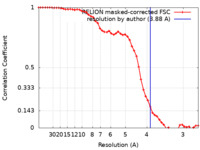
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.88 Å | |||||||||
 データ登録者 データ登録者 | Linares R / Effantin G / Breyton C / Darnault C / Epalle N / Boeri Erba E / Schoehn G | |||||||||
| 資金援助 |  フランス, 2件 フランス, 2件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Cryo-EM structure of the proximal end of bacteriophage T5 tail, in its native state and after interaction with its bacterial receptor 著者: Linares R / Effantin G / Breyton C | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15967.map.gz emd_15967.map.gz | 1.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15967-v30.xml emd-15967-v30.xml emd-15967.xml emd-15967.xml | 20.9 KB 20.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15967_fsc.xml emd_15967_fsc.xml | 5.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15967.png emd_15967.png | 99.9 KB | ||
| マスクデータ |  emd_15967_msk_1.map emd_15967_msk_1.map | 10.5 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-15967.cif.gz emd-15967.cif.gz | 6.4 KB | ||
| その他 |  emd_15967_additional_1.map.gz emd_15967_additional_1.map.gz emd_15967_half_map_1.map.gz emd_15967_half_map_1.map.gz emd_15967_half_map_2.map.gz emd_15967_half_map_2.map.gz | 7.9 MB 7.9 MB 7.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15967 http://ftp.pdbj.org/pub/emdb/structures/EMD-15967 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15967 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15967 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15967_validation.pdf.gz emd_15967_validation.pdf.gz | 795.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15967_full_validation.pdf.gz emd_15967_full_validation.pdf.gz | 795.1 KB | 表示 | |
| XML形式データ |  emd_15967_validation.xml.gz emd_15967_validation.xml.gz | 11.5 KB | 表示 | |
| CIF形式データ |  emd_15967_validation.cif.gz emd_15967_validation.cif.gz | 14.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15967 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15967 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15967 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15967 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8bcpMC  8bcuC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15967.map.gz / 形式: CCP4 / 大きさ: 10.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15967.map.gz / 形式: CCP4 / 大きさ: 10.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.351 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_15967_msk_1.map emd_15967_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
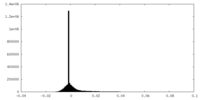
| 投影像・断面図 |
| ||||||||||||
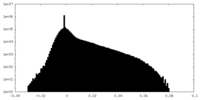
| 密度ヒストグラム |
-追加マップ: unsharpened map
| ファイル | emd_15967_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_15967_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_15967_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Escherichia virus T5
| 全体 | 名称:  Escherichia virus T5 (ウイルス) Escherichia virus T5 (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Escherichia virus T5
| 超分子 | 名称: Escherichia virus T5 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Pure T5 tails obtained by infecting E. coli F strain with the amber mutant phage T5D20am30d NCBI-ID: 2695836 / 生物種: Escherichia virus T5 / Sci species strain: T5D20am30d / ウイルスタイプ: VIRUS-LIKE PARTICLE / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  |
-分子 #1: Tail tube terminator protein p142
| 分子 | 名称: Tail tube terminator protein p142 / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Escherichia phage T5 (ファージ) Escherichia phage T5 (ファージ) |
| 分子量 | 理論値: 18.378643 KDa |
| 配列 | 文字列: MDHRTSIAQA MVDRISKQMD GSQPDEYFNN LYGNVSRQTY KFEEIREFPY VAVHIGTETG QYLPSGQQWM FLELPILVYD KEKTDIQEQ LEKLVADIKT VIDTGGNLEY TVSKPNGSTF PCEATDMIIT SVSTDEGLLA PYGLAEINVT VRYQPPRRSL R R UniProtKB: Tail tube terminator protein p142 |
-分子 #2: Tail tube protein
| 分子 | 名称: Tail tube protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Escherichia phage T5 (ファージ) Escherichia phage T5 (ファージ) |
| 分子量 | 理論値: 50.459215 KDa |
| 配列 | 文字列: MSLQLLRNTR IFVSTVKTGH NKTNTQEILV QDDISWGQDS NSTDITVNEA GPRPTRGSKR FNDSLNAAEW SFSTYILPYK DKNTSKQIV PDYMLWHALS SGRAINLEGT TGAHNNATNF MVNFKDNSYH ELAMLHIYIL TDKTWSYIDS CQINQAEVNV D IEDIGRVT ...文字列: MSLQLLRNTR IFVSTVKTGH NKTNTQEILV QDDISWGQDS NSTDITVNEA GPRPTRGSKR FNDSLNAAEW SFSTYILPYK DKNTSKQIV PDYMLWHALS SGRAINLEGT TGAHNNATNF MVNFKDNSYH ELAMLHIYIL TDKTWSYIDS CQINQAEVNV D IEDIGRVT WSGNGNQLIP LDEQPFDPDQ IGIDDETYMT IQGSYIKNKL TILKIKDMDT NKSYDIPITG GTFTINNNIT YL TPNVMSR VTIPIGSFTG AFELTGSLTA YLNDKSLGSM ELYKDLIKTL KVVNRFEIAL VLGGEYDDER PAAILVAKQA HVN IPTIET DDVLGTSVEF KAIPSDLDAG DEGYLGFSSK YTRTTINNLI VNGDGATDAV TAITVKSAGN VTTLNRSATL QMSV EVTPS SARNKEVTWA ITAGDAATIN ATGLLRADAS KTGAVTVEAT AKDGSGVKGT KVITVTAGG UniProtKB: Tail tube protein pb6 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 構成要素:
| |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER/RHODIUM / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 詳細: 25 mA | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 293.15 K / 装置: FEI VITROBOT MARK IV 詳細: 3 uL of T5 tails sample were deposited on a freshly glow discharged EM grid and plunge-frozen in nitrogen-cooled liquid ethane using a ThermoFisher Mark IV Vitrobot device (100 percent ...詳細: 3 uL of T5 tails sample were deposited on a freshly glow discharged EM grid and plunge-frozen in nitrogen-cooled liquid ethane using a ThermoFisher Mark IV Vitrobot device (100 percent humidity, 20 Celsius degrees, 5 s blotting time, blot force 0). | |||||||||||||||
| 詳細 | Pure T5 tails obtained by infecting E. coli F strain with the amber mutant phage T5D20am30d |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: BACKBONE TRACE / 温度因子: 146 |
|---|---|
| 得られたモデル |  PDB-8bcp: |
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X