+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | p97-p37-SPI substrate complex | ||||||||||||
 マップデータ マップデータ | p97 complex with p37 adaptor and SPI substrate | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | AAA+ ATPase / p97 / VCP / Cdc48 / unfoldase / protein phosphatase-1 / protein maturation / CHAPERONE | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of protein localization to centrosome / PTW/PP1 phosphatase complex / positive regulation of mitotic centrosome separation / regulation of nucleocytoplasmic transport / nuclear membrane reassembly / protein phosphatase regulator activity / protein phosphatase 1 binding / lamin binding / positive regulation of Lys63-specific deubiquitinase activity / flavin adenine dinucleotide catabolic process ...negative regulation of protein localization to centrosome / PTW/PP1 phosphatase complex / positive regulation of mitotic centrosome separation / regulation of nucleocytoplasmic transport / nuclear membrane reassembly / protein phosphatase regulator activity / protein phosphatase 1 binding / lamin binding / positive regulation of Lys63-specific deubiquitinase activity / flavin adenine dinucleotide catabolic process / positive regulation of oxidative phosphorylation / VCP-NSFL1C complex / SHOC2 M1731 mutant abolishes MRAS complex function / Gain-of-function MRAS complexes activate RAF signaling / protein-DNA covalent cross-linking repair / endoplasmic reticulum stress-induced pre-emptive quality control / endosome to lysosome transport via multivesicular body sorting pathway / cellular response to arsenite ion / spindle pole centrosome / Derlin-1 retrotranslocation complex / BAT3 complex binding / positive regulation of protein K63-linked deubiquitination / deubiquitinase activator activity / mitotic spindle disassembly / aggresome assembly / VCP-NPL4-UFD1 AAA ATPase complex / regulation of protein localization to chromatin / vesicle-fusing ATPase / NADH metabolic process / cellular response to misfolded protein / stress granule disassembly / positive regulation of mitochondrial membrane potential / K48-linked polyubiquitin modification-dependent protein binding / negative regulation of protein localization to chromatin / ubiquitin-modified protein reader activity / microtubule organizing center / retrograde protein transport, ER to cytosol / regulation of aerobic respiration / myosin phosphatase activity / protein serine/threonine phosphatase activity / glycogen metabolic process / positive regulation of ATP biosynthetic process / regulation of synapse organization / protein-serine/threonine phosphatase / ATPase complex / ubiquitin-specific protease binding / Triglyceride catabolism / Maturation of hRSV A proteins / entrainment of circadian clock by photoperiod / ubiquitin-like protein ligase binding / MHC class I protein binding / phosphatase activity / Golgi organization / establishment of mitotic spindle orientation / phosphoprotein phosphatase activity / cleavage furrow / RHOH GTPase cycle / blastocyst development / autophagosome maturation / autophagosome assembly / HSF1 activation / polyubiquitin modification-dependent protein binding / proteasomal protein catabolic process / Protein methylation / Amplification of signal from unattached kinetochores via a MAD2 inhibitory signal / endoplasmic reticulum to Golgi vesicle-mediated transport / translesion synthesis / interstrand cross-link repair / enzyme regulator activity / negative regulation of smoothened signaling pathway / ERAD pathway / ATP metabolic process / Mitotic Prometaphase / EML4 and NUDC in mitotic spindle formation / endoplasmic reticulum unfolded protein response / positive regulation of protein dephosphorylation / positive regulation of glial cell proliferation / Attachment and Entry / Resolution of Sister Chromatid Cohesion / protein dephosphorylation / viral genome replication / proteasome complex / lipid droplet / Downregulation of TGF-beta receptor signaling / Josephin domain DUBs / N-glycan trimming in the ER and Calnexin/Calreticulin cycle / ubiquitin binding / RHO GTPases Activate Formins / Hh mutants are degraded by ERAD / macroautophagy / Hedgehog ligand biogenesis / Defective CFTR causes cystic fibrosis / RAF activation / Translesion Synthesis by POLH / circadian regulation of gene expression / positive regulation of protein-containing complex assembly / establishment of protein localization / ABC-family proteins mediated transport / neuron differentiation / regulation of circadian rhythm 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
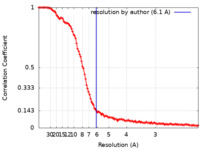
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 6.1 Å | ||||||||||||
 データ登録者 データ登録者 | van den Boom J / Marini G / Meyer H / Saibil H | ||||||||||||
| 資金援助 |  英国, 3件 英国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: EMBO J / 年: 2023 ジャーナル: EMBO J / 年: 2023タイトル: Structural basis of ubiquitin-independent PP1 complex disassembly by p97. 著者: Johannes van den Boom / Guendalina Marini / Hemmo Meyer / Helen R Saibil /   要旨: The AAA+-ATPase p97 (also called VCP or Cdc48) unfolds proteins and disassembles protein complexes in numerous cellular processes, but how substrate complexes are loaded onto p97 and disassembled is ...The AAA+-ATPase p97 (also called VCP or Cdc48) unfolds proteins and disassembles protein complexes in numerous cellular processes, but how substrate complexes are loaded onto p97 and disassembled is unclear. Here, we present cryo-EM structures of p97 in the process of disassembling a protein phosphatase-1 (PP1) complex by extracting an inhibitory subunit from PP1. We show that PP1 and its partners SDS22 and inhibitor-3 (I3) are loaded tightly onto p97, surprisingly via a direct contact of SDS22 with the p97 N-domain. Loading is assisted by the p37 adapter that bridges two adjacent p97 N-domains underneath the substrate complex. A stretch of I3 is threaded into the central channel of the spiral-shaped p97 hexamer, while other elements of I3 are still attached to PP1. Thus, our data show how p97 arranges a protein complex between the p97 N-domain and central channel, suggesting a hold-and-extract mechanism for p97-mediated disassembly. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15861.map.gz emd_15861.map.gz | 116.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15861-v30.xml emd-15861-v30.xml emd-15861.xml emd-15861.xml | 23 KB 23 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15861_fsc.xml emd_15861_fsc.xml | 14.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15861.png emd_15861.png | 166.6 KB | ||
| Filedesc metadata |  emd-15861.cif.gz emd-15861.cif.gz | 7 KB | ||
| その他 |  emd_15861_half_map_1.map.gz emd_15861_half_map_1.map.gz emd_15861_half_map_2.map.gz emd_15861_half_map_2.map.gz | 98.7 MB 98.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15861 http://ftp.pdbj.org/pub/emdb/structures/EMD-15861 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15861 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15861 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15861_validation.pdf.gz emd_15861_validation.pdf.gz | 901.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15861_full_validation.pdf.gz emd_15861_full_validation.pdf.gz | 901.2 KB | 表示 | |
| XML形式データ |  emd_15861_validation.xml.gz emd_15861_validation.xml.gz | 18.4 KB | 表示 | |
| CIF形式データ |  emd_15861_validation.cif.gz emd_15861_validation.cif.gz | 24.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15861 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15861 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15861 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15861 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15861.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15861.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | p97 complex with p37 adaptor and SPI substrate | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||||||||||||||||||
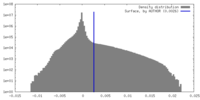
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half map 1
| ファイル | emd_15861_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 | ||||||||||||
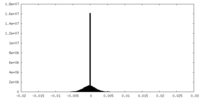
| 投影像・断面図 |
| ||||||||||||
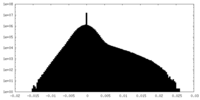
| 密度ヒストグラム |
-ハーフマップ: Half map 2
| ファイル | emd_15861_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 | ||||||||||||
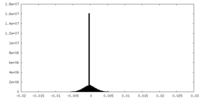
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : p97 complex with p37 adaptor and SPI substrate
| 全体 | 名称: p97 complex with p37 adaptor and SPI substrate |
|---|---|
| 要素 |
|
-超分子 #1: p97 complex with p37 adaptor and SPI substrate
| 超分子 | 名称: p97 complex with p37 adaptor and SPI substrate / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4, #6, #5 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Transitional endoplasmic reticulum ATPase
| 分子 | 名称: Transitional endoplasmic reticulum ATPase / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO / EC番号: vesicle-fusing ATPase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 90.265695 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MHHHHHHASG ADSKGDDLST AILKQKNRPN RLIVDEAINE DNSVVSLSQP KMDELQLFRG DTVLLKGKKR REAVCIVLSD DTCSDEKIR MNRVVRNNLR VRLGDVISIQ PCPDVKYGKR IHVLPIDDTV EGITGNLFEV YLKPYFLEAY RPIRKGDIFL V RGGMRAVE ...文字列: MHHHHHHASG ADSKGDDLST AILKQKNRPN RLIVDEAINE DNSVVSLSQP KMDELQLFRG DTVLLKGKKR REAVCIVLSD DTCSDEKIR MNRVVRNNLR VRLGDVISIQ PCPDVKYGKR IHVLPIDDTV EGITGNLFEV YLKPYFLEAY RPIRKGDIFL V RGGMRAVE FKVVETDPSP YCIVAPDTVI HCEGEPIKRE DEEESLNEVG YDDIGGCRKQ LAQIKEMVEL PLRHPALFKA IG VKPPRGI LLYGPPGTGK TLIARAVANE TGAFFFLING PEIMSKLAGE SESNLRKAFE EAEKNAPAII FIDELDAIAP KRE KTHGEV ERRIVSQLLT LMDGLKQRAH VIVMAATNRP NSIDPALRRF GRFDREVDIG IPDATGRLEI LQIHTKNMKL ADDV DLEQV ANETHGHVGA DLAALCSEAA LQAIRKKMDL IDLEDETIDA EVMNSLAVTM DDFRWALSQS NPSALRETVV EVPQV TWED IGGLEDVKRE LQELVQYPVE HPDKFLKFGM TPSKGVLFYG PPGCGKTLLA KAIANECQAN FISIKGPELL TMWFGE SEA NVREIFDKAR QAAPCVLFFD ELDSIAKARG GNIGDGGGAA DRVINQILTE MDGMSTKKNV FIIGATNRPD IIDPAIL RP GRLDQLIYIP LPDEKSRVAI LKANLRKSPV AKDVDLEFLA KMTNGFSGAD LTEICQRACK LAIRESIESE IRRERERQ T NPSAMEVEED DPVPEIRRDH FEEAMRFARR SVSDNDIRKY EMFAQTLQQS RGFGSFRFPS GNQGGAGPSQ GSGGGTGGS VYTEDNDDDL YG UniProtKB: Transitional endoplasmic reticulum ATPase |
-分子 #2: I3 sequence being threaded through the p97 channel
| 分子 | 名称: I3 sequence being threaded through the p97 channel / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 1.890321 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK) |
-分子 #3: Serine/threonine-protein phosphatase PP1-gamma catalytic subunit
| 分子 | 名称: Serine/threonine-protein phosphatase PP1-gamma catalytic subunit タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO / EC番号: protein-serine/threonine phosphatase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 37.030777 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MADLDKLNID SIIQRLLEVR GSKPGKNVQL QENEIRGLCL KSREIFLSQP ILLELEAPLK ICGDIHGQYY DLLRLFEYGG FPPESNYLF LGDYVDRGKQ SLETICLLLA YKIKYPENFF LLRGNHECAS INRIYGFYDE CKRRYNIKLW KTFTDCFNCL P IAAIVDEK ...文字列: MADLDKLNID SIIQRLLEVR GSKPGKNVQL QENEIRGLCL KSREIFLSQP ILLELEAPLK ICGDIHGQYY DLLRLFEYGG FPPESNYLF LGDYVDRGKQ SLETICLLLA YKIKYPENFF LLRGNHECAS INRIYGFYDE CKRRYNIKLW KTFTDCFNCL P IAAIVDEK IFCCHGGLSP DLQSMEQIRR IMRPTDVPDQ GLLCDLLWSD PDKDVLGWGE NDRGVSFTFG AEVVAKFLHK HD LDLICRA HQVVEDGYEF FAKRQLVTLF SAPNYCGEFD NAGAMMSVDE TLMCSFQILK PAEKKKPNAT RPVTPPRGMI TKQ AKK UniProtKB: Serine/threonine-protein phosphatase PP1-gamma catalytic subunit |
-分子 #4: Protein phosphatase 1 regulatory subunit 7
| 分子 | 名称: Protein phosphatase 1 regulatory subunit 7 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 41.616129 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MAAERGAGQQ QSQEMMEVDR RVESEESGDE EGKKHSSGIV ADLSEQSLKD GEERGEEDPE EEHELPVDME TINLDRDAED VDLNHYRIG KIEGFEVLKK VKTLCLRQNL IKCIENLEEL QSLRELDLYD NQIKKIENLE ALTELEILDI SFNLLRNIEG V DKLTRLKK ...文字列: MAAERGAGQQ QSQEMMEVDR RVESEESGDE EGKKHSSGIV ADLSEQSLKD GEERGEEDPE EEHELPVDME TINLDRDAED VDLNHYRIG KIEGFEVLKK VKTLCLRQNL IKCIENLEEL QSLRELDLYD NQIKKIENLE ALTELEILDI SFNLLRNIEG V DKLTRLKK LFLVNNKISK IENLSNLHQL QMLELGSNRI RAIENIDTLT NLESLFLGKN KITKLQNLDA LTNLTVLSMQ SN RLTKIEG LQNLVNLREL YLSHNGIEVI EGLENNNKLT MLDIASNRIK KIENISHLTE LQEFWMNDNL LESWSDLDEL KGA RSLETV YLERNPLQKD PQYRRKVMLA LPSVRQIDAT FVRF UniProtKB: Protein phosphatase 1 regulatory subunit 7 |
-分子 #5: UBX domain-containing protein 2B
| 分子 | 名称: UBX domain-containing protein 2B / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 1.123238 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: FSGEGQKLGS L UniProtKB: UBX domain-containing protein 2B |
-分子 #6: UBX domain-containing protein 2B
| 分子 | 名称: UBX domain-containing protein 2B / タイプ: protein_or_peptide / ID: 6 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 9.75922 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: AVVLIDDSVP TTKIQIRLAD GSRLIQRFNS THRILDVRNF IVQSRPEFAA LDFILVTSFP NKELTDESLT LLEADILNTV LLQQLK UniProtKB: UBX domain-containing protein 2B |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: C-flat-1.2/1.3 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 85 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均露光時間: 3.0 sec. / 平均電子線量: 48.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD 最大 デフォーカス(公称値): 3.3000000000000003 µm 最小 デフォーカス(公称値): 1.8 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-8b5r: |
 ムービー
ムービー コントローラー
コントローラー






























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)